BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:
URL: http://jams.arakmu.ac.ir/article-1-6111-fa.html


 ، سمیه رئیسی
، سمیه رئیسی 

 2
2
2- استادیار گروه ژنتیک، دانشکده علوم پایه، دانشگاه شهرکرد، شهرکرد، ایران. ، s.reiisi@yahoo.com
مقدمه
سرطان پستان شایعترین نوع بدخیمی و مسئول اغلب مرگها در زنان در سرتاسر جهان است. مطابق با مطالعات آماری سرطان در سال 2018 مشخص شده است که سرطان پستان و تخمدان ازجمله شایعترین انواع سرطان در میان زنان به حساب میآیند. در ایجاد بیماری هم وراثت و هم محیط تأثیرگذار است؛ ازاینرو شناخت عوامل و مکانیسمهای درگیر در بیماری میتواند در تشخیص، پیشآگهی، ملاحظات بالینی و انتخاب روش درمانی مفید باشد [1]. از طرف دیگر، سرطان پستان یک بیماری هتروژن است و ژنهای زیادی را درگیر میکند. درگیر بودن هر ژن خاص در بیماری ممکن است تومورهایی ایجاد کند که از نظر ویژگیهای زیستشناسی با هم متفاوت باشند و نتایج درمانی و بالینی متفاوتی را طلب کنند [2]. در این میان بهتازگی نقش RNA های بلند غیرکدکننده (lncRNA) در شروع و پیشرفت بیماریهایی مانند سرطان بهخوبی آشکار شده است [3].
RNA های بلند غیرکدکننده (lncRNA) نوعی از RNA های غیرکدکننده هستند که بیشتر از 200 نوکلئوتید طول دارند. اصطلاح lncRNA، طیف بسیار گستردهای را دربردارد و تقریباً 80 درصد کل رونوشتهای تهیهشده در سلول را شامل میشوند [4، 5]. ازآنجاکه دهها هزار lncRNA به صورت پلیآدنیله و غیرپلیآدنیله به طور فعال از روی ژنوم انسان رونویسی میشوند، این مولکولها نقشهای مهمی را در تکامل و بیماریهای انسانی ایفا میکنند [7 ،6]. lncRNA ها از تنظیمکنندگان اصلی پرتوانی سلولهای جنینی و الگوبندی محورهای بدن محسوب میشوند. آنها بر خاموشی ژن، سیگنالهای آدنیلاسیون، فاکتورهای مؤثر بر رونویسی و تغییرات هیستونها اثر داشته و در سطوح رونویسی (مانند اپیژنتیک) و پس از رونویسی (مانند دینامیکهای اجزاء سلولی)، مسیرهای رشد و تمایز در سلولهای مختلف نقش دارند [9 ،8].
در مطالعات اخیر مشخص شده است که موتاسیون و همچنین پلیمورفیسمهای تک نوکلئوتیدی (SNP) در ژرم لاین، بهخصوص پلیمورفیسمهای عملکردی در lncRNA ممکن است با خطر سرطانهای مختلف همراهی داشته باشند [10، 11]. بر اساس مطالعات چن و همکاران، همچنین اخیراً این ادعا نیز بیان شده است که واریانتهای ژنتیکی در جایگاههای هدف miRNA ها، به عنوان دسته دیگری از RNA های غیرکدکننده، درون lncRNA ها میتواند با خطر سرطان همراه باشد [11، 12]. برای مثال، یک پلیمورفیسم indel درون ناحیه غیرترجمهشونده '3 در ژن IL-1A قرار دارد، اتصال miR-122 را تحت تأثیر قرار داده و تنظیم بیان آن توسط miRNA را تغییر میدهد [13]. بهعلاوه، پلیمورفیسم در جایگاههای اتصال miRNA در درون توالی lncRNA ممکن است با تغییر سطح بیانی این فاکتورها و افزایش استعداد ابتلا به سرطانهای خاص همراه باشد. وئو و همکاران گزارش کردند که *miR-149 با وجود آلل rs11752942G محکم به lincRNA-uc003opf.1 متصل میشود، سطح بیانی آن و سپس رخداد سرطان سلولهای سنگفرشی مری را کاهش میدهد [14]. در مطالعه لی و همکاران نشان داده شد که پلیمورفیسم rs12325489 C>T درون ناحیه اگزونی lincRNA-ENST00000515084 سبب تخریب جایگاه اتصالی برای miR-370 میشود، به همین دلیل روی فعالیت رونویسی ژن تأثیر گذاشته و تکثیر و رشد سلولی در سرطان پستان را موجب میشود [15]. بر اساس مطالعات همراهی کل ژنوم (GWAS) در نوعی سرطان معده، پنج پلیمورفیسم کاربردی در اگزون lincRNA-NR_024015 مشخص شد. در این مطالعه نشان داده شد که پلیمورفیسم rs8506 در این lncRNA با خطر سرطان معده همراه است و تغییر نوکلئوتیدی C به T در این ناحیه سبب از بین رفتن جایگاه اتصالی miR-526b میشود [16]. این تغییر سبب افزایش استعداد ابتلا به سرطان معده میشود. در مطالعه دیگری اثر این پلیمورفیسم در سرطان سلولهای سنگفرشی مری بررسی شده است و افزایش خطر برای این سرطان و همراهی با پلیمورفیسم rs8506 در توالی lincRNA-NR_024015 را نشان داد [17]؛ اما در مورد سایر سرطانها و ازجمله سرطان پستان هیچگونه مطالعهای در این مورد صورت نگرفته است؛ بنابراین هدف مطالعه ما تعیین نقش پلیمورفیسم rs8506G در همراهی با ریسک ابتلا به سرطان سینه اسپورادیک است. بهعلاوه، ما در این مطالعه همراهی بین پلی مورفیسم ذکرشده و خصوصیات بالینی-پاتولوژیکی را بررسی کردیم.
مواد و روشها
افراد مورد مطالعه
در این مطالعه که به صورت مورد-شاهد انجام شد، نمونههای خونی افراد سرطانی و سالم استفاده شد. نمونههای مورد استفاده مربوط به مطالعات قبلی انجامشده بودند که با کد 0215-91 در کمیته اخلاق دانشگاه علوم پزشکی شهرکرد تأیید شد [18]. نمونهگیری به صورت تصادفی ساده از میان 120 فرد مراجعهکننده به بخش سرطان بیمارستان هاجر شهرکرد با تأیید پاتولوژیک بیماری توسط پزشک متخصص انجام شد. در روش نمونهگیری از افراد واردشده در مطالعه رضایتنامه کتبی دریافت و اطلاعات دموگرافیک و بالینی آنها از طریق پرسشنامه جمعآوری شد. بیماران سرطانی دارای کارسینومای داکتال بودند و تاریخچه خانوادگی در مورد سرطان نداشتند. نمونههای دارای بدخیمیهای ثانویه و عودکننده از مطالعه حذف شدند. از زنان سالمی که به پزشک مراجعه کرده بودند و هیچگونه سابقه بیماری سرطان در آنها یا خانوادهشان وجود نداشت، 120 نمونه به عنوان نمونههای شاهد گرفته شد. نمونههای شاهد و بیمار از نظر سن و منطقه جغرافیایی سکونت با افراد بیمار مطابقت داشتند. همچنین تمامی افراد بیمار و سالم بدون رابطه خویشاوندی با یکدیگر بودند. از هر فرد (کنترل و بیمار) به میزان 2 سیسی خون برای آزمایشهای مولکولی گرفته شد. نمونهها تا زمان استخراج در دمای 20- نگهداری شدند.
تعیین ژنوتیپ در نمونههای سرطانی و سالم
DNA ژنومیک با استفاده از روش فنل-کلروفرم استخراج شد و کیفیت و کمیت DNA استخراجشده با اسپکتروفتومتری (جن وی- انگلستان) بررسی شد. برای تعیین ژنوتیپ نمونهها از روش PCR-RFLP استفاده شد. ابتدا توالی ناحیه مورد نظر که شامل تغییر نوکلئوتیدی است، از پایگاه دادههای ژنومی دریافت و سپس پرایمرهای مورد نظر برای تکثیر آن طراحی شد. پرایمرهای مورد استفاده در مطالعه به صورت پرایمر مستقیم: '5’TTTACCATCAACATGGAAACTTGG3 و پرایمر معکوس: 5’GGTAAATGCTCAGAGTAAACTGC 3’ بودند. شرایط انجام واکنش PCR برای بررسی تغییر مورد نظر روی نمونههای افراد سالم و سرطانی به صورت زیر است: مرحله واسرشت اولیه 95 درجه به مدت 5 دقیقه، 35 سیکل شامل دناتوراسیون 94 درجه به مدت 30 ثانیه، دمای اتصال پرایمرها 60 درجه به مدت 30 ثانیه و سنتز قطعات 72 درجه به مدت 35 ثانیه و طویلسازی نهایی در 72 درجه به مدت 5 دقیقه. همه محصولات برای تأیید نهایی روی ژل آگارز 1 درصد به مدت یک ساعت با ولتاژ 98 الکتروفورز شد و محصولات دارای رنگ فلورسنت تحت تأثیر نور UV مشاهده شدند.
برای تعیین ژنوتیپ محصول PCR تحت تأثیر آنزیم BanII (فرمنتاز-لیتوانی) برای برش قرار گرفت. برای این منظور در هر میکروتیوب 500 نانوگرم از محصولات PCR را با 1 میکرولیتر آنزیم محدودکننده مورد نظر (فرمنتاز- 5 واحد/میکرولیتر) و 2 میکرولیتر بافر اختصاصی آنزیم و 18 میکرولیتر آب عاری از نوکلئاز مخلوط شده و به مدت 16 ساعت در 37 درجه سانتیگراد قرار داده میشود؛ سپس محصولات بهدستآمده روی ژل 10 درصد پلی آکریل آمید به مدت 1 ساعت و با ولتاژ 120 الکتروفورز (پایا پژوهش–ایران) میشوند. در صورت عدم تغییر نوکلئوتیدی در جایگاه واریانت مورد نظر، جایگاه برش برای آنزیم وجود خواهد داشت و روی ژل دو قطعه 108 و 311 جفت بازی مشاهده میشود. در صورت وجود تغییر در توالی، جایگاه برش وجود نخواهد داشت و باند 419 جفت بازی (محصول PCR) دیده میشود. افراد هتروزیگوت روی ژل، سه باند ایجاد میکنند (تصویر شماره 1).
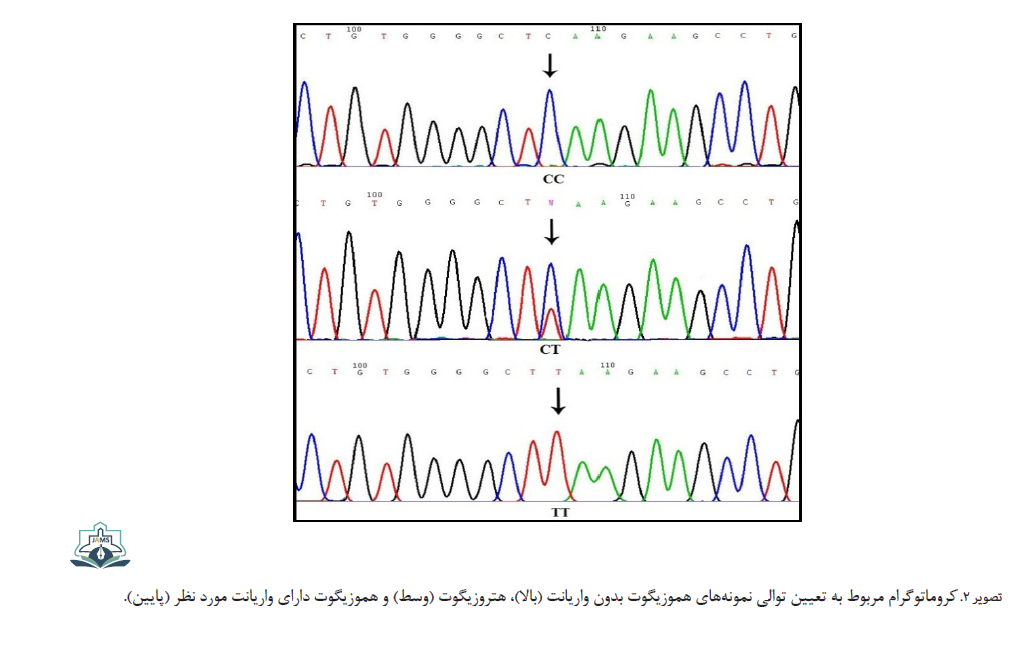
برای تأیید نتایج، درنهایت از روش تعیین توالی استفاده شد. برای این منظور، حدود 10 درصد نمونههای هموزیگوت و هتروزیگوت با حجم بیشتر توسط PCR تکثیر شده و برای تعیین توالی آماده شدند. واکنش تعیین توالی به وسیله دستگاه ABI (Capillary System) 3730XL انجام شد (تصویر شماره 2).
آنالیز آماری
فرکانس آللی پلیمورفیسم برای تعادل هاردی-واینبرگ با استفاده از پایگاه آنلاین آمار عمومی و با اعتبارسنجی انجام شد [19] و برای بررسی فرکانس آللی و ژنوتیپی بین افراد بیمار و سالم از آزمون کای اسکوئر استفاده شد. همچنین برای محاسبه خطر و سپس ارتباط بین فاکتورهای مختلف و ژتونیپهای مورد بررسی از آزمون ANOVA استفاده شد. همه آزمونها از طریق نرمافزار SPPSS V. 20.0 انجام شد. میزان معناداری برای آزمونهای استفادهشده کمتر از 0/05 در نظر گرفته شد.
یافتهها
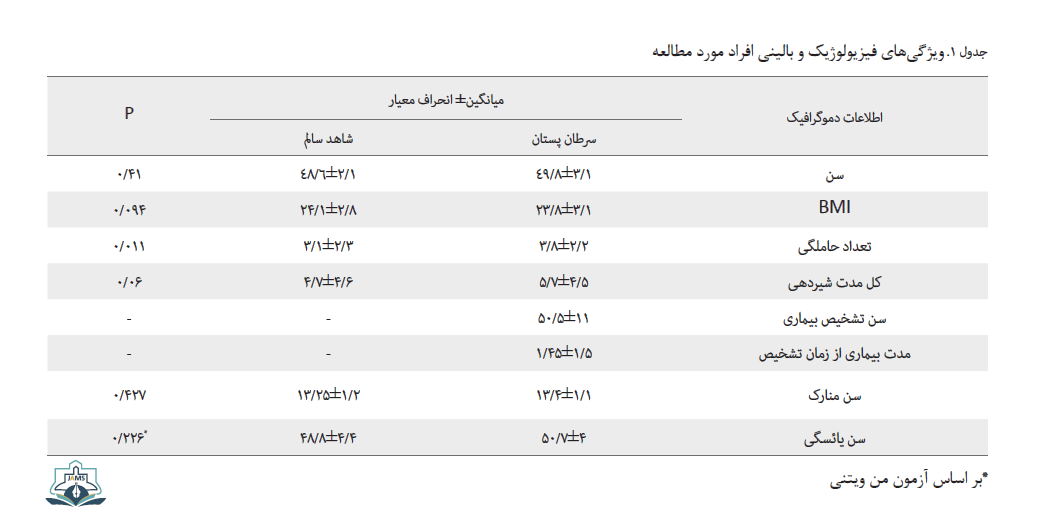
نمونههای مورد مطالعه دارای سرطان اسپورادیک بودند و سن بیماران در دامنه 26 تا 77 سال با میانگین 11±51/8 سال و سن افراد شاهد در دامنه 16 تا 70 سال با میانگین 10/1± 36/9سال بود (0/41=P). هیچ ارتباط معناداری در خصوص فاکتورهای وزن، BMI، زمان شیردهی و همچنین یائسگی و سن منارک بین دو گروه مشاهده نشد (جدول شماره 1).
بعد از تعیین ژنوتیپ پلیمورفیسم rs8506G در افراد شاهد و سرطانی، از آزمون مربع کای برای بررسی پیروی جمعیت از تعادل هاردی واینبرگ استفاده شد. برای گروههای شاهد و بیمار میزان 2 χ بهترتیب برابر 0/04.و 0/6 محاسبه شد. این مورد نشان میدهد که جمعیت کنترل از تعادل هاردی واینبرگ پیروی میکند (تصویرشماره 3).
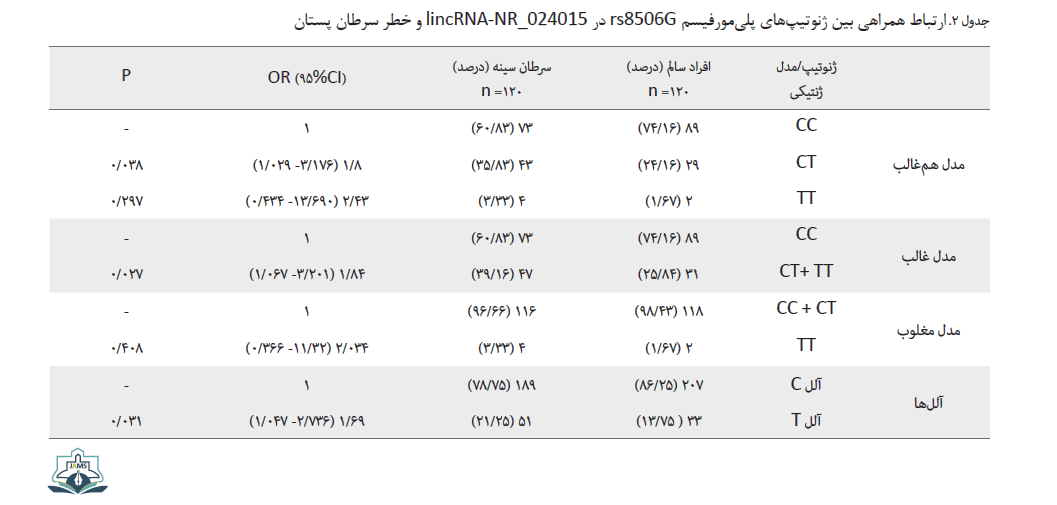
برای مشخص کردن میزان خطر و همراهی واریانت مورد نظر با سرطان پستان، توزیع واریانت میان افراد شاهد و سرطانی بررسی و نتایج آن به صورت خلاصه در جدول شماره 2 ارائه شده است.
مطابق با دادههای بهدستآمده، فراوانی ژنوتیپ هتروزیگوت (CT) و هموزیگوت ( TT) در افراد سرطانی نسبت به افراد سالم بیشتر است، به صورتی که این میزان برای ژنوتیپ هتروزیگوت حدود 36 درصد در افراد سرطانی و 24 درصد در افراد سالم است. فراوانی ژنوتیپ TT در افراد سرطانی حدوداً دو برابر افراد سالم نشان داده شد؛ بنابراین مدلهای ژنتیکی مختلف برای پلیمورفیسم بررسی شد تا بهترین وضعیت برای ژنوتیپها مشخص شود. در مدل غالب، همراهی معناداری میان پلیمورفیسم و سرطان پستان هنگام حضور دو ژنوتیپ TT و CT در جمعیت دیده میشود (0/027=P). با بررسی سایر مدلها نیز مشخص شد که حضور ژنوتیپ TT در مدل مغلوب خطر بیماری را تا حدود دو برابر افزایش میدهد (2/01= OR). در مدل همغالب و غالب نیز در حالتهای حضور ژنوتیپ دارای آلل T میزان خطر بیماری بهترتیب برابر با 2/43 و 1/84 مشاهده شد. در این بررسی مشخص شد که فراوانی آلل T در گروه شاهد برابر با 13 درصد است، ولی در افراد بیمار فراوانی آن بیشتر و به میزان 21 درصد محاسبه شده است. با توجه به میزان خطر محاسبهشده، تغییر آللی سبب افزایش خطر برای وقوع بیماری میشود و این تغییر از نظر آماری ارتباط معناداری را نشان داد (0/031).
تمامی این نتایج تأییدکننده نقش پلیمورفیسم برای استعداد ابتلا به سرطان پستان است. در بررسی ارتباط میان فاکتورهای کلینوپاتولوژی و خطر بیماری هیچگونه ارتباط معناداری میان پلیمورفیسم مورد نظر و این فاکتورها مشاهده نشد. در این مورد با توجه به اطلاعات بیماران، فاکتورهای متاستاز به گره لنفی، درجه توموری و فاکتور HER2 بررسی شد. از نظر درجه توموری (درجه 1 تا 3) با توجه به توزیع متغیر در سه گروه، افراد به دو گروه درجه پایین و درجه بالا تقسیم شدند. در بررسی بین گروههای بالینی و پلیمورفیسم مورد مطالعه هیچ اختلاف معناداری یافت نشد. جدول شماره 3 بررسی این فاکتورها را در مدل ژنتیکی غالب نشان می دهد.
بحث
بیان ژنهای هدف توسط miRNA ها به وسیله اتصال آنها به ناحیه 3’-UTR تنظیم میشود، بنابراین هرگونه تغییر در این ناحیه ممکن است جایگاه اتصالی miRNA را تخریب کند و به دنبال آن روی عملکرد بیانی ژن تأثیر بگذارد [20]. در مطالعه حاضر، ما همراهی میان واریانت عملکردی در lincRNA-NR_024015 و خطر سرطان پستان را بررسی کردیم. در مطالعات محتلف نقش lincRNA-NR_024015 در تومورزایی به طور کامل مشخص نشده است، اما با نتایج حاصل از این مطالعه میتوانیم این احتمال را در نظر بگیریم که تغییر نوکلئوتیدی در lincRNA-NR_024015 که سبب از بین رفتن جایگاه اتصالی miR-526b و به دنبال آن برداشته شدن اثر مهاری miRNA از روی میشود، افزایش خطر سرطان پستان را به همراه دارد.
در حال حاضر، lncRNA ها به عنوان یک دسته از RNA های تنظیمی غیرکدکننده، دارای توجه ویژه برای مطالعات مختلف هستند. این مولکولهای شبه mRNA، فاقد یک چارچوب خوانش مشخص هستند و معمولاً فرایندهای کلاهکگذاری، پردازش و پلیآدنیله شدن در آنها رخ میدهد. چنین ویژگی در lncRNA ها سبب شده است که آنها در فرایندهای مختلف بیولوژیک نقش داشته باشند. علاوهبراین، مطالعات مختلف، عملکرد lncRNA ها را در انواع بیمایها و ازجمله سرطانها گزارش کردهاند. برای مثال در مطالعه کوتاک و همکاران مشخص شد که یک lincRNA غالب، ANRIL به طور عملکردی در پیشرفت سرطان با مهار بیان ژنی به صورت اپی ژنتیک با فراخوانی کمپلکسهای تغییر کروماتین نقش دارد [21]. در مطالعه موریسون و همکاران روی سرطان پستان، GAS5 به عنوان یک lincRNA معروف در تومورزایی گزارش شده است [22]. این شواهد اهمیت lncRNAها را در سرطانزایی، ازجمله سرطان پستان تأیید میکند.
مطالعات میکایلیدو و همکاران روی سرطان پستان توسط آنالیز کل ژنوم (GWAS)، توانسته است تعدادی زیادی از لوکوسهایی را که با تکوین سرطان پستان همراه هستند، شناسایی کند [23]. در این میان، تعداد زیادی از lncRNA ها یافت شدند که به این لوکوسها نزدیک بودند و علاوهبراین، تغییرات ژنتیکی در توالی این lncRNA ها نیز مشخص شد؛ بنابراین پلیمورفیسمهای عملکردی در این توالیها ممکن است روی تکوین و استعداد ابتلا به تومور تأثیر بگذارد. پلیمورفیسم rs920778 در توالی HOTAIR، به عنوان یک lncRNA، با افزایش بیان این فاکتور و استعداد ابتلا به سرطان پستان همراه است؛ بنابراین وجود تغییر ژنتیکی در توالی lncRNA مورد نظر سبب افزایش بدخیمی و تهاجم سلولی در سرطان پستان میشود [24]. لی و همکاران با بررسی ژنوم توسط آنالیز GWAS نشان دادند که پلیمورفیسم rs12325489C>T در توالی اگزونی lincRNA-ENST00000515084 به طور قابلتوجهی با کاهش خطر سرطان پستان همراه است.
در این مطالعه مشخص شد که احتمالاً واریانت یافتشده یک اصلاحکننده ژنتیکی برای سرطان پستان باشد [15]. در مطالعه هان و همکاران در مورد پلیمورفیسم rs8506 در توالی lincRNA-NR_024015 نیز این مورد مشاهده شده است. مطابق با این مطالعه در سرطان سلولهای سنگفرشی مری، وجود پلیمورفیسم باعث از بین رفتن جایگاه اتصالی miR-526b میشود و این اثر با افزایش بیان بیشتر در lincRNA-NR_024015 همراه بوده که افزایش بدخیمی را در این سلولها موجب میشود [17]. با توجه به اینکه در سرطانهای مختلف بیان miR-526b دچار کاهش شده است [25]، وجود چنین واریانتی سبب مهار اتصال آن به lncRNA مورد نظر میشود، بنابراین خطر تکوین تومورزایی و پیشرفت سرطان برای افراد با ژنوتیپ TT افزایش بیشتری خواهد یافت. مشخص شده است که در بیماران سرطانی، میزان بیان lincRNA-NR_024015 دچار افزایش بوده و این مکانیسم یا همان اثر تغییر ژنتیکی در توالی، افزایش بیان را تشدید میکند.
نتیجهگیری
بهطورکلی، مطالعه حاضر نشان داد که حضور واریانت ژنتیکی در lincRNA-NR_024015 میتواند یک فاکتور افزایش خطر برای سرطان پستان باشد، به صورتی که تغییر آلل C به T در توالی ژنی lincRNA-NR_024015 سبب از بین رفتن جایگاه هدف miR-526b روی این توالی شده و درنتیجه با از بین رفتن اثر مهاری، افزایش بیان این lncRNA را به دنبال خواهد داشت. این افزایش با تکوین و پیشرفت سرطان پستان و پیشآگهی بد در این سرطان همراه خواهد بود؛ باوجوداین به دلیل اینکه هیچگونه مطالعهای در مورد سرطان پستان و این lncRNA وجود ندارد، به نظر میرسد بررسی در سایر قسمتها مانند بررسی در تعداد نمونه بیشتر و اثر در لاینهای سلولی ضروری باشد.
ملاحظات اخلاقی
پیروی از اصول اخلاق پژوهش
این مطالعه از سوی کمیته اخلاق دانشگاه علوم پزشکی شهرکرد با کد 0215-91 تأیید شده است. کلیهی اصول اخلاقی در این مقاله رعایت شده است. شرکتکنندگان اجازه داشتند هر زمان که مایل بودند از پژوهش خارج شوند. همچنین همه شرکتکنندگان در جریان روند پژوهش بودند و اطلاعات آنها محرمانه نگهداشته شد
حامی مالی
این مقاله حاصل پایاننامه خانم فاطمه توکلی برای اخذ درجه کارشناسی ارشد در رشته ژنتیک در گروه ژنتیک، دانشکده علوم پایه، دانشگاه شهرکرد، شهرکرد است.
مشارکت نویسندگان
تمامی نویسندگان معیارهای استاندارد نویسندگی بر اساس پیشنهادات کمیته بینالمللی ناشران مجلات پزشکی را دارا بودند.
تعارض منافع
بدینوسیله نویسندگان تصریح میکنند که هیچگونه تضاد منافعی در خصوص پژوهش حاضر وجود ندارد.
تشکر و قدردانی
نویسندگان قدردانی خود را از معاونت پژوهشی دانشگاه شهرکرد که حامی مالی و معنوی این مطالعه بود، اعلام میدارند. همچنین از همه افرادی که در این مطالعه ما را یاری کردند، سپاسگزاری میکنیم.
References
1.Stephens PJ, Tarpey PS, Davies H, Van Loo P, Greenman C, Wedge DC, et al. The landscape of cancer genes and mutational processes in breast cancer. Nature. 2012; 486(7403):400-4. [DOI:10.1038/nature11017] [PMID] [PMCID]
2.Shah R, Rosso K, Nathanson SD. Pathogenesis, prevention, diagnosis and treatment of breast cancer. World J Clin Oncol. 2014; 5(3):283-98. [DOI:10.5306/wjco.v5.i3.283] [PMID] [PMCID]
3.Huarte M. The emerging role of lncRNAs in cancer. Nature Medicine. 2015; 21(11):1253-61. [DOI:10.1038/nm.3981] [PMID]
4.Mercer TR, Dinger ME, Mattick JS. Long non-coding RNAs: Insights into functions. Nature reviews genetics. 2009; 10(3):155-9. [DOI:10.1038/nrg2521] [PMID]
5.Cao J. The functional role of long non-coding RNAs and epigenetics. Biological procedures online. 2014; 16:11. [DOI:10.1186/1480-9222-16-11] [PMID] [PMCID]
6.Yu AD, Wang Z, Morris KV. Long noncoding RNAs: A potent source of regulation in immunity and disease. Immunology and cell biology. 2015; 93(3):277-83. [DOI:10.1038/icb.2015.2] [PMID]
7.Spurlock CF 3rd, Crooke PS 3rd, Aune TM. Biogenesis and Transcriptional Regulation of Long Noncoding RNAs in the Human Immune System. J Immunol. 2016; 197(12):4509-17. [DOI:10.4049/jimmunol.1600970] [PMID] [PMCID]
8.Mattick JS. The genetic signatures of noncoding RNAs. PLoS Genet. 2009; 5(4):e1000459. [DOI:10.1371/journal.pgen.1000459] [PMID] [PMCID]
9.Batista PJ, Chang HY. Long noncoding RNAs: Cellular address codes in development and disease. Cell. 2013; 152(6):1298-307. [DOI:10.1016/j.cell.2013.02.012] [PMID] [PMCID]
10.Wojcik SE, Rossi S, Shimizu M, Nicoloso MS, Cimmino A, Alder H, et al. Non-codingRNA sequence variations in human chronic lymphocytic leukemia and colorectal cancer. Carcinogenesis. 2009; 31(2):208-15. [DOI:10.1093/carcin/bgp209] [PMID] [PMCID]
11.Jendrzejewski J, He H, Radomska HS, Li W, Tomsic J, Liyanarachchi S, et al. The polymorphism rs944289 predisposes to papillary thyroid carcinoma through a large intergenic noncoding RNA gene of tumor suppressor type. Proceedings of the National Academy of Sciences. 2012; 109(22):8646-51. [DOI:10.1073/pnas.1205654109] [PMID] [PMCID]
12.Chen K, Song F, Calin GA, Wei Q, Hao X, Zhang W. Polymorphisms in microRNA targets: A gold mine for molecular epidemiology. Carcinogenesis. 2008; 29(7):1306-11. [DOI:10.1093/carcin/bgn116] [PMID]
13.Gao Y, He Y, Ding J, Wu K, Hu B, Liu Y, et al. An insertion/deletion polymorphism at miRNA-122-binding site in the interleukin-1α 3′ untranslated region confers risk for hepatocellular carcinoma. Carcinogenesis. 2009; 30(12):2064-9. [DOI:10.1093/carcin/bgp283] [PMID]
14.Wu H, Zheng J, Deng J, Hu M, You Y, Li N, et al. A genetic polymorphism in lincRNA-uc003opf. 1 is associated with susceptibility to esophageal squamous cell carcinoma in Chinese populations. Carcinogenesis. 2013; 34(12):2908-17. [DOI:10.1093/carcin/bgt252] [PMID]
15.Li N, Zhou P, Zheng J, Deng J, Wu H, Li W, et al. A polymorphism rs12325489C> T in the lincRNA-ENST00000515084 exon was found to modulate breast cancer risk via GWAS-based association analyses. PloS one. 2014; 9(5):e98251. [DOI:10.1371/journal.pone.0098251] [PMID] [PMCID]
16.Fan Q-H, Yu R, Huang W-X, Cui X-X, Luo B-H, Zhang L-Y. The has-miR-526b binding-site rs8506G> a polymorphism in the lincRNA-NR_024015 exon identified by GWASs predispose to non-cardia gastric cancer risk. PloS one. 2014; 9(3):e90008. [DOI:10.1371/journal.pone.0090008] [PMID] [PMCID]
17.Han L, Liu S, Liang J, Guo Y, Shen S, Guo X, et al. A genetic polymorphism at miR‐526b binding‐site in the lincRNA‐NR_024015 exon confers risk of esophageal squamous cell carcinoma in a population of North China. Mol Carcinog. 2017; 56(3):960-71. [DOI:10.1002/mc.22549] [PMID]
18.Barjui SP, Reiisi S, Ebrahimi S, Shekari B. Study of correlation between genetic variants in three microRNA genes (hsa-miR-146a, hsa-miR-502 binding site, hsa-miR-27a) and breast cancer risk. Current research in translational medicine. 2017; 65(4):141-7. [DOI:10.1016/j.retram.2017.10.001] [PMID]
19.Rodriguez S, Gaunt TR, Day IN. Hardy-Weinberg equilibrium testing of biological ascertainment for Mendelian randomization studies. American journal of epidemiology. 2009; 169(4):505-14. [DOI:10.1093/aje/kwn359] [PMID] [PMCID]
20.Saunders MA, Liang H, Li W-H. Human polymorphism at microRNAs and microRNA target sites. Proceedings of the National Academy of Sciences. 2007; 104(9):3300-5. [DOI:10.1073/pnas.0611347104] [PMID] [PMCID]
21.Kotake Y, Nakagawa T, Kitagawa K, Suzuki S, Liu N, Kitagawa M, et al. Long non-coding RNA ANRIL is required for the PRC2 recruitment to and silencing of p15p15 INK4B tumor suppressor gene. Oncogene. 2011; 30(16):1956-62. [DOI:10.1038/onc.2010.568] [PMID] [PMCID]
22.Morrison LE, Jewell SS, Usha L, Blondin BA, Rao RD, Tabesh B, et al. Effects of ERBB2 amplicon size and genomic alterations of chromosomes 1, 3, and 10 on patient response to trastuzumab in metastatic breast cancer. Genes, Chromosomes and Cancer. 2007; 46(4):397-405. [DOI:10.1002/gcc.20419] [PMID]
23.Michailidou K, Lindström S, Dennis J, Beesley J, Hui S, Kar S, et al. Association analysis identifies 65 new breast cancer risk loci. Nature. 2017; 551(7678):92-4. [DOI:10.1038/nature24284] [PMID] [PMCID]
24.Bayram S, Sümbül AT, Batmacı CY, Genç A. Effect of HOTAIR rs920778 polymorphism on breast cancer susceptibility and clinicopathologic features in a Turkish population. Tumor Biology. 2015; 36(5):3863-70. [DOI:10.1007/s13277-014-3028-0] [PMID]
25.Zhang Z-y, Fu S-l, Xu S-q, Zhou X, Liu X-s, Xu Y-j, et al. By downregulating Ku80, hsa-miR-526b suppresses non-small cell lung cancer. Oncotarget. 2015; 6(3):1462-77. [DOI:10.18632/oncotarget.2808] [PMID] [PMCID]
| بازنشر اطلاعات | |
 |
این مقاله تحت شرایط Creative Commons Attribution-NonCommercial 4.0 International License قابل بازنشر است. |