BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:
URL: http://jams.arakmu.ac.ir/article-1-6123-fa.html


 ، ناصر سنچولی2
، ناصر سنچولی2 

 ، نیما سندگل1
، نیما سندگل1 


2- گروه زیستشناسی، دانشکده علوم پایه، دانشگاه زابل، زابل، زاهدان، ایران. ،
مقدمه
بیماری آلزایمر یک اولویت بهداشت جهانی شناخته شده است، زیرا هزینه زیادی را بر سیستمهای مراقبتهای بهداشتی و اقتصادی تحمیل میکند [1]. آلزایمر بیماری تحلیلبرنده عصبی است که به عنوان شایعترین شکل جنون شناخته میشود و نزدیک به حدود ۴۶ میلیون نفر در سراسر دنیا به آن مبتلا هستند. اختلالات شناختی و رفتاری از علائم آن است و از نشانههای مهم پاتولوژی آن میتوان به تخریب و تحلیل رفتن نورونها اشاره کرد [2]. یکی از مهمترین فاکتورهای بیماری آلزایمر سن است و بر اساس سن به دو گروه بیماری آلزایمر زودرس و بیماری آلزایمر دیررس تقسیمبندی میشود.
در بیماری آلزایمر زودرس، ژنهای پروتئین پیشساز آمیلوئیدبتا، پرسنیلین 1 و پرسینیلین ۲ نقش دارند [3]. همچنین از نشانگرهای پاتولوژیک بیماری آلزایمر میتوان به وجود پلاکهای آمیلوئیدی حاوی آمیلوئیدبتا خارجسلولی و فیبرهای نورونی بههمریخته که به وسیله پروتئین تائو در درون نورونها ایجاد میشوند، اشاره کرد [4]. این بیماری میتواند مسیرهای سلولی از قبیل سیستم یوبی کوئیتینه پروتئاز، اتوفاژی، چاپرونها و هر سیستمی را که با تجمع پروتئینی درگیر است، فعال کند [5].
مورفولوژی شبکه آندوپلاسمی شامل ورقها، لولهها و ماده زمینهای است [6]. شبکه آندوپلاسمی در تاخوردن و حمل کردن پروتئینها، تولید انرژی و آپوپتوز نقش دارد [7]. هومئوستاز پروتئینهای تاخورده در شبکه آندوپلاسمی برای عملکرد سلول بسیار حیاتی است و وجود اختلالات خارجسلولی و داخلسلولی مثل مواد شیمیایی یا شرایط فیزیولوژیکی باعث تجمع پروتئینهای تانخورده و پروتئینهای اشتباه تاخورده در شبکه آندوپلاسمی میشود [8]. اختلال عملکرد در شبکه آندوپلاسمی در بیماریهای مختلف انسانی مانند بیماری آلزایمر، پارکینسون و هانتینگتون دیده شده است [9].
پاسخ پروتئینهای تانخورده پاسخی به استرسهای ناشی از شبکه آندوپلاسمی به علت اختلال در پروتئینهای تاخورده است. تجمع پروتئینهای اشتباه تاخورده و تجمعات پروتئینی ویژگی مشترک بین بیماریهای تحلیلبرنده عصبی مثل بیماری آلزایمر، پارکینسون و پریون است [10]. زمانی که شبکه آندوپلاسمی تحت استرس قرار میگیرد، سیگنالهای موجود در شبکه آندوپلاسمی تحریک میشوند و درنتیجه پاسخ پروتئینهای تانخورده در شبکه آندوپلاسمی ایجاد میشود [11]. در بسیاری از بیماریهای تحلیلبرنده عصبی ازجمله ام اس پاسخ پروتئینهای تانخورده دچار مشکل میشود [12]. سه مسیر پاسخ پروتئینهای تانخورده را فعال میکند که این سه مسیر شامل فعالکننده فاکتور ۶ رونویسی، اینوزیتول متصل به پروتئین ۱ و پروتئین کیناز شبه R شبکه آندوپلاسمی کیناز است [13].
عملکرد شبکه آندوپلاسمی و مورفولوژی آن با اتوفاژی ارتباط دارد [6]. به تازگی نشان داده شده است که استرس شبکه آندوپلاسمی باعث اتوفاژی میشود [14]. اتوفاژی برای حفظ هومئوستاز بافت ضروری است و از شروع و پیشرفت بسیاری از بیماریها مانند پیری، بیماریهای تحلیلبرنده عصبی و سرطان جلوگیری میکند [15]. اتوفاژی فرایندی است که در اکثر سلولها وجود دارد و مسئول تخریب پروتئینهای اشتباه تاخورده و ارگانهای آسیبدیده از طریق دستگاههای لیزوزومی است [16]. ثابت شده که اتوفاژی با بیماریهای تحلیلبرنده عصبی ازجمله بیماری آلزایمر مرتبط است [17] و همچنین دیده شده است که در بیماری آلزایمر فرآیند اتوفاژی دچار اختلال میشود [18]. در اتوفاژی زمانی که استرس وجود دارد، اتوفاگوزوم تشکیل میشود که حاوی بخشهایی ازپروتئینهای شبکه آندوپلاسمی است. اتوفاژی که در شبکه آندوپلاسمی اتفاق میافتد به عنوان خودخواری شبکه آندوپلاسمی یا رتیکولوفاژی شناخته شده است و انعطافپذیری شبکه آندوپلاسمی را در طول پاسخ پروتئینهای تانخورده متعادل کرده و به هومئوستاز سلول کمک میکند [19]. بر اساس محرکهای خاص مثل استرس شبکه آندوپلاسمی، محرومیت مواد مغذی، تجمع پروتئینها و یا حمله پاتوژنها، ER-phagy افزایش پیدا میکند [20]. ER-phagy شامل تجزیه پروتئینهای ناحیه سیترونی شبکه آندوپلاسمی داخل اتوفاگوزومها است که در مخمر و پستانداران دیده شده است. در پستانداران، چهار گیرنده برای ER-phagy وجود دارد که این گیرندهها اساساً پروتئینهای ساکن شبکه آندوپلاسمی یا غشای انتقالی هستند که دارای دامینهای سیتوزولی و لومنی هستند یا دامینهایی روی غشای شبکه آندوپلاسمی دارند؛ بااینحال وجود یک موتیف ناحیه متصلشونده به LC3 در سیتوزول ضروری است، زیرا موتیف LIR برای تجزیه شبکه آندوپلاسمی در اتوفاگوزوم مورد نیاز است. پروتئینها وابسته به شبکه آندوپلاسمی شامل تنظیمکننده خودخواری شبکه آندوپلاسمی ۱، پیشرفت چرخه سلولی 1، همولوگ SEC62 و رتیکولون ۳ است [21].
مولکول FAM134B در لبههای ورقهای شبکه آندوپلاسمی قرار گرفته است و نقش آن به ساب دامینهایی که دارد، وابسته است. FAm13B به وسیله دامین همولوگ Rel (RHD) خود شبکه آندوپلاسمی را تجزیه و سپس از طریق ناحیه LIR خود، قطعات شبکه آندوپلاسمی را به غشاهای اتوفاژیک تبدیل میکند. تنظیم مقادیر FAM134B یا جهش در LIR آن موجب گسترش شبکه آندوپلاسمی میشود، درحالیکه بیان بیشازحد FAM134B باعث تجزیه شبکه آندوپلاسمی و تخریب لیزوزومی ورقهای شبکه آندوپلاسمی وپروتئینهای ساکن آن مثل پروتئین ۴ وابسته به اسکلت سلولی (CLIMP63) میشود. FAM134B نقش مهمی در تخریب شبکه آندوپلاسمی در اثر پاسخ به گرسنگی یا شرایط استرس شبکه آندوپلاسمی دارد؛ علاوهبراین بهتازگی منتقلکننده کلسترول داخلسلولی NPC1 اشتباه تاخورده که یک گلیکوپروتئین است و در شبکه آندوپلاسمی ساخته میشود، به عنوان یک بستر آندوژنی برای ER-phagy وابسته به FAM134B شناخته شده است؛ اما اینکه چگونه FAM134B میتواند NPC1 اشتباه تاخورده را تشخیص دهد، هنوز مشخص نیست. این یافته نشان میدهد که ER-phagy ممکن است پروتئینهای اشتباه تاخورده خاص دیگری را نیز هدف بگیرد [20].
مولکول Ccpg1 به عنوان یک عامل مؤثر بر استرس وابسته به ژن پروتئین مرتبط با گیرنده گاماآمینو بوتیریک اسید و پروتئینهای مرتبط با B1/A1 میکروتوبول زنجیره سبک ۳ بتا شناخته شده است. این پروتئین یک پروتئین نوع II شبکه آندوپلاسمی است که به طور مستقیم با پروتئین زنجیره ۱ القا شده ۱ RB (FIP200) و به طور غیرمستقیم با اجزای کمپلکس Unc ۵۱ کیناز مانند (UIK) که شامل پروتئین ۱۰۱ وابسته به اتوفاژی (ATG101)، پروتئین ۱۳ وابسته به اتوفاژی (ATG13) و پروتئین سرین/تیروزین کیناز (ULK1) است، باعث میشود که بیوژنز اتوفاگوزوم انجام شود. بیان غیرعادی CCPG 1 باعث کاهش اندازه شبکه آندوپلاسمی محیطی و محتوای سلولی از پروتئین RTN3 لولههای شبکه آندوپلاسمی محیطی میشود. مولکول CCPG1 برای انجام ER-phagy نیاز به پروتئین ۵ اتوفاژی (ATG5) و FIP200 دارد [22].
مولکول RTN3 یک پروتئین حاوی دامین RHD است که در لولههای شبکه آندوپلاسمی قرار دارد و پس از گرسنگی فعال میشود. این پروتئین به عنوان یکی از اعضای خانواده رتیکولون شناخته شده است که در شکلگیری لولههای شبکه آندوپلاسمی دخالت دارد. RTN3 دارای ایزوفرمهای مختلف است و طولانیترین ایزوفرم RTN3 دارای شش دامین LIR فعال در ناحیه انتهایی N است که برای اتصال با LC3 و GABARAP مهم است. گزارش شده است که RTN3 در بیماریهای تحلیلبرنده عصبی بهویژه بیماری آلزایمر دخالت دارد؛ بااینحال بیشتر تجزیهوتحلیلهای مربوط روی ایزوفرمهای کوچک RTN3 بوده است؛ البته غیر از ایزوفرمهای بزرگ که در حمل موتیف LIR نقش دارد؛ بنابراین اثر بالقوه آن بر ER-phagy هنوز باقی مانده است [6].
مولکول Sec62 قسمتی از انتقال غشایی وابسته به شبکه آندوپلاسمی است که در دستگاه انتقالی sec 61/62/63 قرار دارد و نقش آن، انتقال پروتئینهای تازهتولیدشده به لومن شبکه آندوپلاسمی است. sec62 به عنوان گیرنده ER-phagy شناخته شده است که پس از تمام شدن استرس شبکه آندوپلاسمی، باعث تنظیم شبکه آندوپلاسمی میشود. فرآیند کاتابولیکی تنظیمشده توسط sec62، مقدار و حجم شبکه آندوپلاسمی قبل از استرس را بازسازی و درنتیجه آن را به عنوان ER-phagy ترمیمی معرفی میکند. منطقه LIR که در sec62 وجود دارد، برای RecoER-phagy موردنیاز است، اما برای نقش دیگر آن یعنی انتقال پروتئینها موردنیاز نیست. تاکنون گزارش شده است که RecoER-phagy با استفاده از sec62 به پروتئینهای اتوفاژی مثل LC3, ATG5 و آنزیم ATG7 فعال اصلاحکننده یوبی کوئیتینه مانند ATG7 نیاز دارد [23].
مواد و روشها
تهیه و بررسی دادههای ریزآرایه
در این پژوهش از پایگاه داده GEO استفاده شد [24]. کلیدواژه بیماری آلزایمر برای پیدا کردن مطالعات مرتبط در نظر گرفته شد. برای این کلیدواژه از میان مطالعات ریزآرایه، مطالعاتی انتخاب شد که روی موشهای تراریخته انجام شده بود؛ سپس مطالعات غیرمرتبط و مطالعات دارویی حذف شد. برای آنالیز این دادهها از نرمافزار آنلاین GEO2R استفاده شد که معیار آماری آن Benjamin & Hochberg است و میزان معنیداری برای تغییرات بیان ۰۵/۰ در نظر گرفته شد [25].
بررسی ارتباطات پروتئینی
برای بررسی ارتباطات پروتئینی از پایگاه داده STRING استفاده شد. در این رابطه برای ژنهای NPC1 FAM134B, Sec62, Rtn3 و Ccpg1 ارتباطات پروتئینی بررسی و رسم شد و ارتباطات پروتئینی جداگانهای برای ژنهای FAM134B و NPC1 نیز بررسی و رسم شد [25].
یافتهها
با بررسی کلیدواژه بیماری آلزایمر، 267 مطالعه به دست آمد و از میان این مطالعاتT 109 مورد انتخاب شد که روی موش انجام شده بود؛ سپس ۹ مطالعه انتخاب شد که به مقایسه بیان ژن روی موشهای غیرتراریخته (وحشی) و مدل آلزایمر پرداخته بود. دادههای مطالعات با نرمافزار آنلاین GEO2R آنالیز و تغییرات بیان ژنها ثبت شد که این مطالعات شامل: 1- مطالعه GSE31624 که در ناحیه هیپوکامپ موشهای دارای جهش Tg2576 (ژن APP) است؛ ۲- مطالعه GSE104249 که در ناحیه مغز موشهای دارای جهش APPPS1 (ژنهای APP و PSEN1) است؛ 3- مطالعه GSE109055 که در دو ناحیه هیپوکامپ و کورتکس موشهای دارای جهش 3xTg (ژنهای APP، PSEN1 و MAPT) است. در ناحیه هیپوکامپ، ژن Sec62 با معناداری 0.02 و تغییر بیان 0.258- و ژن Ccpg1 در همین ناحیه مغز با تغییر بیان 0.145- و با 0.009 میزان معناداری دیده شد و در ناحیه کورتکس همین مطالعه نیز ژن Ccpg1 با معناداری 0.01 و با تغییر بیان 0.192- مشاهده شد؛ 4- مطالعه GSE92926 که در ناحیه کورتکس موشهای دارای جهش 3xTg (ژنهای APP ،PSEN1 و MAPT) است؛ ۵- مطالعه GSE53480 که در ناحیه هیپوکامپ موشهای دارای جهش rTg (tauP301L) 4510 (ژن MAPT) است که ژن FAM134B با تغییر بیان 0.195 و میزان معناداری 0.005 دیده شد؛ 6- مطالعه GSE31372 که در ناحیه مغز قدامی موشهای دارای جهش Tg2756 (ژن APP) است که ژن FAM134B با تغییر بیان 0.210 و میزان معناداری 0.02 مشاهده شد؛ ۷- مطالعه GSE60911 که در ناحیه قشر پیش پیشانی مغز موشهای دارای جهش 3xTg (ژنهای APP ،PSEN1 و MAPT) است؛ ۸- مطالعه GSE60460 که در ناحیه زیربطنی مغز موشهای دارای جهش 3xTg (ژنهای APP ،PSEN1 و MAPT) است که ژن NPC1 با تغییر بیان 0.551 و میزان معناداری 0.006 دیده شد؛ ۹- مطالعه GSE36981 که در ناحیه هیپوکامپ موشهای دارای جهش 3xTg (ژنهای APP ،PSEN1 و MAPT) است که ژن NPC1 با تغییر بیان 0.120 و میزان معناداری 0.03 مشاهده شد (تصویر شماره 1 و جدول شماره 1) [26].
ارتباطات پروتئینی ژنهای دارای افزایش بیان
برای ژنهای NPC1 ،FAM134B ،Sec62، Rtn3 و Ccpg1 ارتباطات پروتئینی بررسی و رسم شد (تصویر شماره 2).
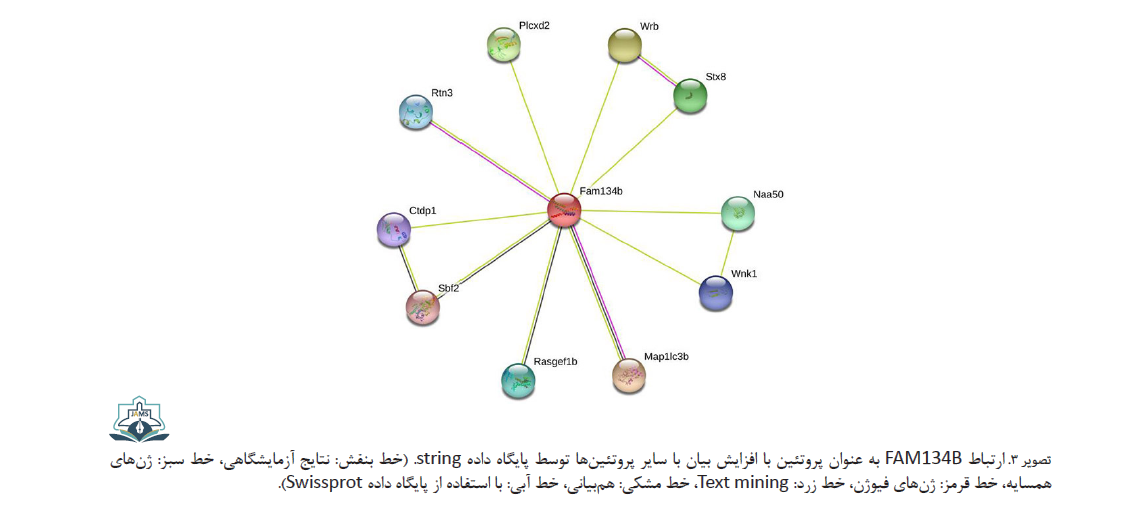
پروتئین FAM134B با پروتئینهای دیگری نیز ارتباط دارد؛ ازجمله این پروتئینها میتوان به پروتئین پایدار غنی از تریپتوفان سینتاکسین ۸ و RTN3 اشاره کرد که در انتقال پروتئینها نقش دارند. پروتئین N-آلفا-استیل ترانسفراز05 استیلاسیون پروتئینها را وساطت میکند. پروتئین سرین / ترئونین پروتئین کیناز در هومئوستاز الکترولیتهای سلولی، مسیر سیگنالینگ، بقا و تکثیر سلولی نقش دارد. LC3 یا Map1lc3b در یوبی کوئیتینه کردن پروتئینها و حذف پروتئینها نقش دارد. پروتئینهای عضو خانواده 1B حاوی دامنه Ras-GEF (Rasgef1b) و پروتئین ۱۳ مرتبط با میوتوبولین (Sbf2)، فاکتورهای هستهای تبادل گوانین هستند. زیرواحد RNA پلیمراز A II دامنه فسفاتاز ناحیه C (Ctdp1) در دفسفوریلاسیون انتهای C پروتئین RNA پلیمراز II نقش دارد (تصویر شماره 3).
پروتئین منتقلکننده داخلسلولی ۲ NPC یک منتقلکننده درونسلولی کلسترول است که همراه با پروتئین منتقلکننده داخلسلولی ۱ NPC در خروج کلسترول از لیزوزوم فعالیت دارد. پروتئین ۵ مرتبط با پروتئین متصلشونده به اکسیسترول (Osbpl5) یک منتقلکننده کلسترول بین شبکه آندوپلاسمی و غشای سلولی است. پروتئین ۱ متصلشونده به عنصر تنظیمی استرول (Srebf1) و پروتئین ۲ متصلشونده به عنصر تنظیمی استرول (Srebf2) فعالکنندههای رونویسی با هدف هومئوستاز لیپیدها هستند و همچنین در تنظیم فعالیت ژنهای گیرنده LDL و سنتز کلسترول نقش دارند. پروتئین جزء ۱ زیرخانواده کستل متصلشونده به ATP در خروج کلسترول و فسفولیپیدها و انتقال آن به آپولیپوپروتئین HDL فعالیت دارد. پروتئین Abca1 در انتقال و هومئوستاز لیپدها در ماکروفاژها نقش دارد. پروتئین اسفنگومیلین فسفودی ترانسفراز تبدیل اسفنگومیلین به سرآمید را وساطت میکند. پروتئین ۱ متصلشونده به GTP یک پروتئین GTPase است که در تنظیم پایداری mRNA نقش دارد. نیوفریکین یک پروتئین متصلشونده به هم Hem است (تصویر شماره 4) [27].
آنالیز بیان ژنهای پروتئینهای مرتبط با ژن FAM134B
در دو مطالعه GSE53480 و GSE31372، ژن FAM134B افزایش بیان پیدا کرده و ژنهای پروتئینهای مرتبط با FAM134B نیز بررسی شده بود. در مطالعه GSE53480 ژن Wnk1 با تغییر بیان 0.212 و میزان معناداری 0.01، افزایش بیان و در ژن Sbf2 با تغییر بیان 0.350- و میزان معناداری 0.01 در آن، کاهش بیان دیده شد. در مطالعه GSE31372 ژن Map1lc3b با تغییر بیان 0.167 و میزان معناداری 0.04 افزایش بیان را نشان داد (جدول شماره ۲).
آنالیز بیان ژنهای پروتئینهای مرتبط با ژن NPC1
در دو مطالعه GSE60460 و GSE36981 که ژن NPC1 افزایش بیان داشت، ژنهای پروتئینهای مربوط NPC1 نیز بررسی شد. در مطالعه GSE60460 ژن عضو ۱ زیرخانواده G کستل متصل به ATP (Abcg1) با تغییر بیان 0.529 و میزان معناداری 0.002 افزایش بیان و ژن Srebf2 با تغییر بیان 0.384- و میزان معناداری 0.03 کاهش بیان را نشان داد و در مطالعه GSE36981 ژن Osbpl5 با تغییر بیان 0.177- و میزان معناداری 0.04 کاهش بیان مشاهده شد (جدول شماره ۳).
شبکه آندوپلاسمی یکی از پیچیدهترین اندامکها در سلولهای یوکاریوتی است. تحقیقات اخیر نشان میدهد که فرایند ER-phagy بیشترین نقش را در شکل و عملکرد شبکه آندوپلاسمی ایفا میکند [20]. یکی از وظایف شبکه آندوپلاسمی، کنترل کیفیت پروتئینها است، اما اخیراً دیده شده که در شرایط استرس، قسمتی از غشای شبکه آندوپلاسمی همراه پروتئینهای آن به لیزوزوم منتقل و حذف میشود و این استرس میتواند شامل استرس به وسیله دارو یا استرس کلی سلول و یا ناشی از گرسنگی باشد [28]. نشان داده شده است که فرایند ER-phagy وابسته به فرایند پاسخ به پروتئینهای تانخورده است [29]. بر پایه گزارشهای پزشکی، استرس شبکه آندوپلاسمی در بیماران آلزایمر اتفاق میافتد. این مورد در مطالعات حیوانی و در (in vitro) نیز دیده شده است. همچنین ثابت شده است که بیماریهای تحلیلبرنده عصبی مانند بیماری آلزایمر، بیماری پارکینسون و غیره با فرایند پاسخ به پروتئینهای تانخورده مرتبط هستند [30]؛ ازاینرو ما در این پژوهش به بررسی ارتباط بین بیان ژنهای دخیل در ER-phagy در بیماری آلزایمر پرداختیم. ازآنجاکه ژنهای FAM134B ،Npc1 ،SEC62، Ccpg1 و Rtn3 از ژنهای مهم ER-phagy هستند، میزان بیان آنها را در مدلهای آلزایمر بررسی کردیم، اما فقط در چهار مطالعه از 9 مطالعه انتخابشده، افزایش بیان فقط در دو ژن FAM134B و NPC1 دیده شد. این دادهها نشان میدهد که در بیماری آلزایمر بیان ژنهای ER-phagy تغییر زیادی نمیکند. ازآنجاکه ژنهای FAM134B، Npc1 افزایش بیان نشان دادند و ژنهای دیگر ER-phagy افزایش بیان نداشتند، به نظر آمد که این ژنها عملکردی غیر از ER-phagy داشته باشند (تصویر شماره 3 و 4)؛ ازاینرو تغییر بیان ژنهای پروتئینهایی که با این دو پروتئین (Npc1 و FAM134B) در ارتباط هستند، نیز بررسی شد. ژن FAM134Bدر دو مطالعه افزایش بیان داشت؛ یکی در ناحیه هیپوکامپ که همراه با آن ژن Wnk1 افزایش بیان را نشان داد و نیز در ناحیه مغز قدامی، ژن Map1lc3b همراه با ژن FAM134B افزایش بیان مشاهده شد (جدول شماره ۲). همراه با افزایش بیان NPC1 در ناحیه زیربطنی، در ژن Abcg1 نیز افزایش بیان دیده شد (جدول شماره ۳).
نتیجهگیری
به نظر میرسد که جهشهای مربوط به APP و MAPT میتوانند بیان ژنهای FAM134B و NPC1 را افزایش دهند و همچنین جهش در PSEN1 هم میتواند بیان ژن NPC1 را افزایش دهد. ژنهای Wnk1 در ناحیه هیپوکامپ و ژن Map1lc3b در ناحیه مغز قدامی همراه با ژن FAM134B افزایش بیان داشتند؛ ازاینرو پیشبینی میشود که ژن FAM134B تأثیر خود را از طریق بقا، تکثیر سلولی، تشکیل فاگوزوم و حذف از طریق یوبی کوئیتینه کردن نقش خود را ایفا کند. در ناحیه زیربطنی مغز که ژن NPC1 افزایش بیان داشت، ژن Abcg1 افزایش بیان پیدا کرد؛ ازاینرو احتمال میرود که ژن NPC1 از طریق هومئوستاز لیپیدها نقش خود را انجام دهد. به طور خلاصه نتایج بیان ژن نشان میدهد که ER-phagy نقش پررنگی در بیماری آلزایمر ایفا نمیکند و احتمالاً ژنهای FAM134B و NPC1 از مسیرهای دیگر تأثیر خود را بر آن میگذارند. پیشنهاد میشود که مطالعات آزمایشگاهی بیشتری روی این موضوع انجام شود.
ملاحظات اخلاقی
پیروی از اصول اخلاق پژوهش
تمامی ملاحظات اخلاقی در این پژوهش رعایت شدهاند.
حامی مالی
این مقاله توسط دانشگاه زابل حمایت مالی شده است.
مشارکت نویسندگان
مفهوم سازی: تمام نویسندگان؛ تحقیق و بررسی: ذکیه قریب؛ ویراستاری و نهایی سازی نوشته: ناصر سنچولی و نیما سندگل.
تعارض منافع
نویسندگان هیچ تعارض منافعی را گزارش نکردهاند.
تشکر و قدردانی
نویسندگان مایلند از دانشگاه زابل بابت حمایت مالی این پژوهش تشکر نمایند.
References
1.Haapasalo A, Pikkarainen M, Soininen H. Alzheimer’s disease. A report from the 7th Kuopio Alzheimer symposium. Neurodegener Dis Manag. 2015; 5(5):379-82. [DOI:10.2217/nmt.15.31] [PMID]
2.Zhu JB, Tan CC, Tan L, Yu JT. State of play in Alzheimer’s disease genetics. J Alzheimers Dis. 2017; 58(3):631-59. [DOI:10.3233/JAD-170062] [PMID]
3.Giau VV, Senanarong V, Bagyinszky E, An SSA, Kim S. Analysis of 50 Neurodegenerative genes in clinically diagnosed early-onset Alzheimer’s disease. Int J Mol Sci. 2019; 20(6):1514. [DOI:10.3390/ijms20061514] [PMID] [PMCID]
4.Khan A, Corbett A, Ballard C. Emerging amyloid and tau targeting treatments for Alzheimer’s disease. Expert Rev Neurother. 2017; 17(7): 697-711. [DOI:10.1080/14737175.2017.1326819]
5.Zhang Y, Chen X, Zhao Y, Ponnusamy M, Liu Y. The role of ubiquitin proteasomal system and autophagy-lysosome pathway in Alzheimer’s disease. Rev Neurosci. 2017; 28(8): 861-8. [DOI:10.1515/revneuro-2017-0013] [PMID]
6.Grumati P, Dikic I, Stolz A. ER-phagy at a glance. J Cell Sci. 2018; 131(17):217364. [DOI:10.1242/jcs.217364] [PMID]
7.Ahmadian N, Hejazi S, Mahmoudi J, Talebi M. Tau pathology of alzheimer disease: Possible role of sleep deprivation. Basic Clin Neurosci. 2018; 9(5):307. [DOI:10.32598/bcn.9.5.307] [PMID] [PMCID]
8.Poothong J, Sopha P, Kaufman RJ, Tirasophon W. IRE1α nucleotide sequence cleavage specificity in the unfolded protein response. FEBS lett. 2017; 591(2): 406-414. [DOI:10.1002/1873-3468.12546] [PMID] [PMCID]
9.Muneer A, Khan S, Mozammil R. Endoplasmic reticulum stress: Implications for neuropsychiatric disorders. Chonnam Med J. 2019; 55(1):8-19. [DOI:10.4068/cmj.2019.55.1.8] [PMID] [PMCID]
10.Scheper W, Hoozemans JJ. The unfolded protein response in neurodegenerative diseases: A neuropathological perspective. Acta Neuropathol. 2015; 130(3): 315-31. [DOI:10.1007/s00401-015-1462-8] [PMID] [PMCID]
11.Dong Z, Cui H. The autophagy-lysosomal pathways and their emerging roles in modulating proteostasis in tumors. Cells. 2019; 8(1):4. [DOI:10.3390/cells8010004] [PMID] [PMCID]
12.Sanadgol N, Maleki P. Study of the effects of ellagic acid on population and activity of central nervous system neuroglia cells in the cuprizone-induced multiple sclerosis. J Arak Uni Med Sci. 2018; 21(6):34-46. http://jams.arakmu.ac.ir/article-1-5743-en.html
13.Castillo-Carranza DL, Zhang Y, Guerrero-Munoz MJ, Kayed R, Rincon-Limas DE, Fernandez-Funez P. Differential activation of the ER stress factor XBP1 by oligomeric assemblies. Neurochem Res. 2012; 37(8):1707-17. [DOI:10.1007/s11064-012-0780-7] [PMID] [PMCID]
14.Yorimitsu T, Klionsky DJ. Endoplasmic reticulum stress: A new pathway to induce autophagy. Autophagy. 2007; 3(2):160-2. [DOI:10.4161/auto.3653] [PMID]
15.Forrester A, De Leonibus C, Grumati P, Fasana E, Piemontese M, Staiano L, et al. A selective ER‐phagy exerts procollagen quality control via a Calnexin‐FAM134B complex. EMBO J. 2019; 38(2):e99847. [DOI:10.15252/embj.201899847] [PMID] [PMCID]
16.Correia SC, Resende R, Moreira PI, Pereira CM. Alzheimer’s disease-related misfolded proteins and dysfunctional organelles on autophagy menu. DNA Cell Biol. 2015; 34(4):261-73. [DOI:10.1089/dna.2014.2757] [PMID]
17.Uddin M, Stachowiak A, Mamun AA, Tzvetkov NT, Takeda S, Atanasov AG, et al. Autophagy and Alzheimer’s disease: From molecular mechanisms to therapeutic implications. Front Aging Neurosci. 2018; 10:4. [DOI:10.3389/fnagi.2018.00004] [PMID] [PMCID]
18.Salminen A, Kaarniranta K, Kauppinen A, Ojala J, Haapasalo A, Soininen H, et al. Impaired autophagy and APP processing in Alzheimer’s disease: The potential role of Beclin 1 interactome. Prog Neurobiol. 2013; 106:33-54. [DOI:10.1016/j.pneurobio.2013.06.002] [PMID]
19.Anding AL, Baehrecke EH. Cleaning house: Selective autophagy of organelles. Dev Cell. 2017; 41(1):10-22. [DOI:10.1016/j.devcel.2017.02.016] [PMID] [PMCID]
20.Dikic I. Open questions: Why should we care about ER-phagy and ER remodelling? BMC biol. 2018; 16(1):131. [DOI:10.1186/s12915-018-0603-7] [PMID] [PMCID]
21.Smith M, Wilkinson S. ER homeostasis and autophagy. Essays Biochem. 2017; 61(6):625-35. [DOI:10.1042/EBC20170092] [PMID] [PMCID]
22.Fregno I, Molinari M. Endoplasmic reticulum turnover: ER-phagy and other flavors in selective and non-selective ER clearance. F1000 Res. 2018; 7:454. [DOI:10.12688/f1000research.13968.1] [PMID] [PMCID]
23.Loi M, Fregno I, Guerra C, Molinari M. Eat it right: ER-phagy and recover-phagy. Biochem Soc Trans. 2018; 46(3):699-706. [DOI:10.1042/BST20170354] [PMID] [PMCID]
24.Edgar R, Michael D, Alex EL. Gene Expression Omnibus: NCBI gene expression and hybridization array data repository. Nucleic Acids Res. 2002; 30(1):207-10. [DOI:10.1093/nar/30.1.207] [PMID] [PMCID]
25.Amini J, Sanchooli N, Sanadgol N. [Evaluation of microarray-derived gene expression patterns in transgenic mouse models of Alzheimer’s disease (Tau and Amyloid beta) using bioinformatics tools (Persian)]. J Cell Tissue. 2019; 10 (1): 1-11. http://jct.araku.ac.ir/article_35313_en.html
26.Kinoshita J, Clark T. Alzforum. Methods Mol Biol. 2007; 365-81. [DOI:10.1007/978-1-59745-520-6_19] [PMID]
27.UniProt Consortium. UniProt: A hub for protein information. Nucleic Acids Res. 2014; 43(Database issue):D204-12. [DOI:10.1093/nar/gku989] [PMID] [PMCID]
28.Lipatova Z, Segev N. A role for macro-ER-phagy in ER quality control. PLoS Genet. 2015; 11(7):e1005390. [DOI:10.1371/journal.pgen.1005390] [PMID] [PMCID]
29.Song S, Tan J, Miao Y, Zhang Q. Crosstalk of ER stress‐mediated autophagy and ER‐phagy: Involvement of UPR and the core autophagy machinery. J Cell Physiol. 2018; 233(5):3867-74. [DOI:10.1002/jcp.26137] [PMID]
30.Li JQ, Yu JT, Jiang T, Tan L. Endoplasmic reticulum dysfunction in Alzheimer’s disease. Mol Neurobiol. 2015; 51(1):383-95. [DOI:10.1007/s12035-014-8695-8] [PMID]
| بازنشر اطلاعات | |
 |
این مقاله تحت شرایط Creative Commons Attribution-NonCommercial 4.0 International License قابل بازنشر است. |