دوره 23، شماره 5 - ( آذر و دی - شماره ویژه کووید 19 1399 )
جلد 23 شماره 5 صفحات 631-614 |
برگشت به فهرست نسخه ها
Download citation:
BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:



BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:
Hojabr Rajeooni A, Mehrbod P. Molecular Structure of SARS-CoV-2 Virus and the Proposed Related Drugs. J Arak Uni Med Sci 2020; 23 (5) :614-631
URL: http://jams.arakmu.ac.ir/article-1-6323-fa.html
URL: http://jams.arakmu.ac.ir/article-1-6323-fa.html
هژبر راجعونی علی، مهربد پروانه. بررسی ساختار مولکولی ویروس SARS-CoV-2 و داروهای مرتبط. مجله دانشگاه علوم پزشكي اراك. 1399; 23 (5) :614-631
1- گروه ویروسشناسی، دانشکده دامپزشکی، دانشگاه تهران، تهران، ایران.
2- بخش آنفلوانزا و ویروسهای تنفسی شایع، انستیتو پاستور ایران، تهران، ایران. ،mehrbode@yahoo.com
2- بخش آنفلوانزا و ویروسهای تنفسی شایع، انستیتو پاستور ایران، تهران، ایران. ،
واژههای کلیدی: کووید 19، کروناویروس تیپ 2، سندرم حاد تنفسی، روند تکاملی، بیولوژی مولکولی، پیشگیری، درمان
متن کامل [PDF 9801 kb]
(4607 دریافت)
| چکیده (HTML) (5985 مشاهده)
متن کامل: (26980 مشاهده)
مقدمه
بیماری کووید 19 از بیماریهای مهم ویروسی در دهه حاضر محسوب میشود که پاندمی گستردهای در دنیا ایجاد کرده است. این بیماری اولینبار در 17 آذر 1398 از شهر ووهان استان هوبی چین از بیماران با علائم پنومونی شدید گزارش شد. در 17 دی 1398 مرکز کنترل و پیشگیری از بیماریها در چین، ویروس را شناسایی نمود و سازمان بهداشت جهانی آن را nCoV-2019 نامید. سپس نام بیماری به کووید 19 تغییر یافت. بر اساس دادههای کمیته بینالمللی طبقهبندی ویروسها این ویروس SARS-CoV-2 نامیده میشود که از خانواده کروناویروسها محسوب میشود. ویروسهای موجود در این خانواده در سالهای 1381 (بیماری سارس) و 1391 (بیماری مرس) همهگیری وسیعی در چندین کشور مختلف داشتهاند و منجر به مرگومیر و زیان اقتصادی شدهاند. در مقاله حاضر این بیماری از جنبههای مختلف روند تکاملی و بیولوژی مولکولی ویروس بررسی شده است تا در جهت کنترل، پیشگیری و درمان بیماری مؤثر واقع شود.
تاکسونومی و آنالیز فیلوژنتیکی ویروس
ویروس SARS-CoV-2 از خانواده ویروسهای غشادار با RNA پلاریته مثبت است که مهرهداران را مبتلا میکند. در طبقهبندی کروناویروسها، 39 گونه در 27 تحت جنس شناسایی شده است. پنج جنس و دو تحت خانواده متعلق به خانواده کروناویریده، تحت راسته کروناویرینه، راسته نیدو وایرالز، قلمرو ریبو ویریا وجود دارد [2 ،1].
ویروس SARS-CoV-2 متعلق به گونه کروناویروس مرتبط با سندرم فوق حاد تنفسی، تحت جنس ساربکوویروس، جنس بتا کروناویروس، تحت خانواده ارتوکروناویرینه است [2]. مطالعات تکاملی نشان میدهند این ویروس شباهت نزدیکی با کروناویروسهای شناساییشده در خفاش (خفاش نعل بینی) و مورچهخوار پولکدار (پانگولین) دارد (تصویر شماره 1).
بیماری کووید 19 از بیماریهای مهم ویروسی در دهه حاضر محسوب میشود که پاندمی گستردهای در دنیا ایجاد کرده است. این بیماری اولینبار در 17 آذر 1398 از شهر ووهان استان هوبی چین از بیماران با علائم پنومونی شدید گزارش شد. در 17 دی 1398 مرکز کنترل و پیشگیری از بیماریها در چین، ویروس را شناسایی نمود و سازمان بهداشت جهانی آن را nCoV-2019 نامید. سپس نام بیماری به کووید 19 تغییر یافت. بر اساس دادههای کمیته بینالمللی طبقهبندی ویروسها این ویروس SARS-CoV-2 نامیده میشود که از خانواده کروناویروسها محسوب میشود. ویروسهای موجود در این خانواده در سالهای 1381 (بیماری سارس) و 1391 (بیماری مرس) همهگیری وسیعی در چندین کشور مختلف داشتهاند و منجر به مرگومیر و زیان اقتصادی شدهاند. در مقاله حاضر این بیماری از جنبههای مختلف روند تکاملی و بیولوژی مولکولی ویروس بررسی شده است تا در جهت کنترل، پیشگیری و درمان بیماری مؤثر واقع شود.
تاکسونومی و آنالیز فیلوژنتیکی ویروس
ویروس SARS-CoV-2 از خانواده ویروسهای غشادار با RNA پلاریته مثبت است که مهرهداران را مبتلا میکند. در طبقهبندی کروناویروسها، 39 گونه در 27 تحت جنس شناسایی شده است. پنج جنس و دو تحت خانواده متعلق به خانواده کروناویریده، تحت راسته کروناویرینه، راسته نیدو وایرالز، قلمرو ریبو ویریا وجود دارد [2 ،1].
ویروس SARS-CoV-2 متعلق به گونه کروناویروس مرتبط با سندرم فوق حاد تنفسی، تحت جنس ساربکوویروس، جنس بتا کروناویروس، تحت خانواده ارتوکروناویرینه است [2]. مطالعات تکاملی نشان میدهند این ویروس شباهت نزدیکی با کروناویروسهای شناساییشده در خفاش (خفاش نعل بینی) و مورچهخوار پولکدار (پانگولین) دارد (تصویر شماره 1).
این مطالعات نشان دادند در سطح نوکلئوتیدی، این ویروس حدود 79 درصد با ویروس SARS-CoV شباهت دارد و حدود 72 درصد با توالی ژن پروتئین Spike (S) ویروس بیماری سارس شباهت دارد [3]. مطالعات تکاملی کروناویروسهای شناختهشده در خفاشها نشان دادند از نظر توالی نوکلئوتیدی ویروس SARS-CoV در خفاش نعل بینی (RaTG13) حدود 96 درصد و در ناحیه دُمین اتصال به گیرنده (RBD) حدود 85 درصد با ویروس بیماری کووید 19 شباهت دارد. ژن S1 در ویروس کووید 19، 70 درصد شباهت توالی با ویروس بتاکرونا دارد [4]. علیرغم این شباهت از نظر ویژگیهای کلیدی ژنومی متفاوت است، بهگونهای که ژن Spike (S) ویروس SARS-CoV-2 در محل شکاف حاوی چهار اسیدآمینه بازی است که در بیماریزایی ویروس نقش مهمی دارند [5]. در مطالعه دیگری از کروناویروس خفاش (RmYN02) نشان داده شد، ویروس SARS-CoV-2 در حدود 97 درصد با ژن Replicase بتاکروناویروسهای خفاش شباهت دارد. مطالعات روی کروناویروسهای مورچهخوار پولکدار نشان داد ویروس SARS-CoV-2 در ناحیه دُمین اتصال به گیرنده (RBD) پروتئین Spike حدود 97 درصد با کروناویروسهای مورچهخوار شباهت آمینواسیدی دارد [6]. در این راستا، مطالعه دیگری برای شناخت روند تکاملی ویروس در جمعیت انسانی صورت گرفت و سه واریانت مرکزی با تجزیه و تحلیل شبکه فیلوژنتیکی ژنوم ویروس به دست آمد. بر اساس جهشهای ژنومی سه واریانت A ،B و C برای این ویروس شناسایی شدهاند [8 ،7]. واریانت A به عنوان تیپ اجدادی ویروس با شباهت 96/2 درصد با SARS-CoV خفاش در آسیای شرقی شناسایی شد. واریانت A در افراد ساکن کشورهای شرق آسیا، بر اساس جهش مترادف T29095C با دو تحت کلاستر آلل T و C شناسایی شد.
واریانت B براثر دو جهش مترادف T8782C و جهش غیرمترادف تبدیل لوسین به سرین C28144T بیشتر در کشورهای آسیای شرقی رایج است. این واریانت علاوه بر کشورهای شرق آسیا، در کشورهای آسیایی مجاور، ایالات متحده و کشورهای اروپایی نیز شناسایی شد. واریانت C بیشترین تیپ ویروس در کشورهای اروپایی است. این واریانت در ایالات متحده، برزیل، سنگاپور، تایوان، هنگکنگ و کره جنوبی نیز شناسایی شده است، اما در چین این تیپ مشاهده نشد. این واریانت حاصل جهش غیرمترادف تبدیل گلایسین به والین G26144T از واریانت B است (تصویر شماره 2).
واریانت B براثر دو جهش مترادف T8782C و جهش غیرمترادف تبدیل لوسین به سرین C28144T بیشتر در کشورهای آسیای شرقی رایج است. این واریانت علاوه بر کشورهای شرق آسیا، در کشورهای آسیایی مجاور، ایالات متحده و کشورهای اروپایی نیز شناسایی شد. واریانت C بیشترین تیپ ویروس در کشورهای اروپایی است. این واریانت در ایالات متحده، برزیل، سنگاپور، تایوان، هنگکنگ و کره جنوبی نیز شناسایی شده است، اما در چین این تیپ مشاهده نشد. این واریانت حاصل جهش غیرمترادف تبدیل گلایسین به والین G26144T از واریانت B است (تصویر شماره 2).
بیولوژی مولکولی ویروس
ویروس SARS-CoV-2 دارای ژنوم RNA با پلاریته مثبت با آرایشی است که در تصویر شماره 3 نشان داده شده است.
ویروس SARS-CoV-2 دارای ژنوم RNA با پلاریته مثبت با آرایشی است که در تصویر شماره 3 نشان داده شده است.
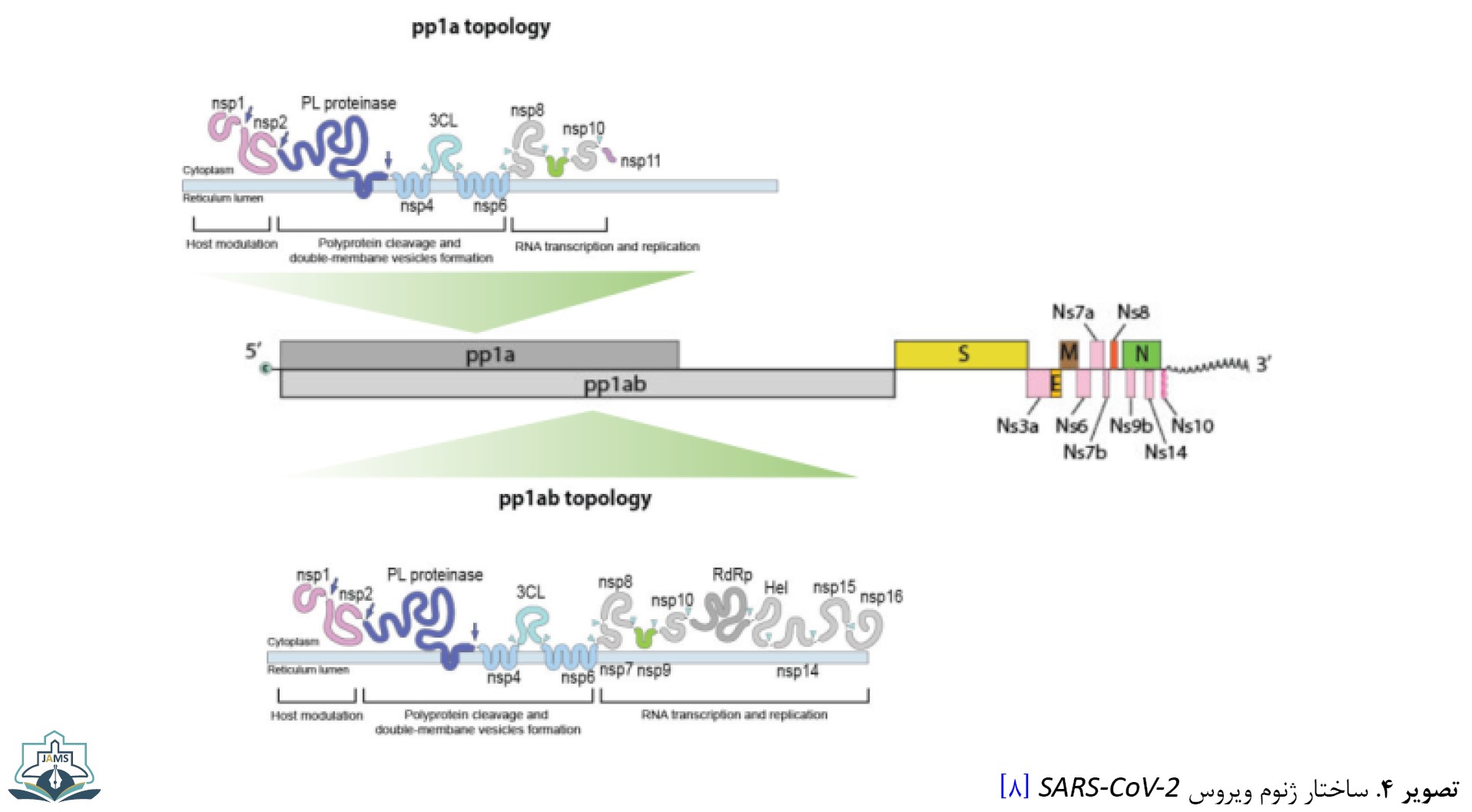
اندازه ژن طولانی Replicase (ژن ORF1ab) بیش از 21Kb است و شانزده پروتئین غیرساختاری را کد میکند (NSP 1->16) که بهصورت پلیپروتئین pp1ab ترجمه میشود. در کنار این ژن، چهارده پروتئین غیرساختاری توسط mRNAهای تحت ژنومی (NS 3a->14) نیز کد میشوند. تصویر شماره 4 ساختار ژنوم ویروس SARS-CoV-2 را نشان میدهد
پروتئین nsp1 با اتصال به زیرواحد 40S ریبوزوم در سلول منجر به مهار ترجمه در سلول میزبان میشود. این کمپلکس سبب القای شکاف اندونوکلئولیتیک ناحیه 5’UTR در mRNAهای میزبان و درنهایت موجب تجزیه آنها میشود. mRNAهای ویروسی به دلیل وجود توالی هدایتکننده انتهایی در ناحیه 5’ در برابر این شکاف اندونوکلئولیتیک محافظت میشوند. با سرکوب بیان ژن در سلول میزبان، پروتئین nsp1، بیان ژنهای ویروسی در سلولهای مبتلا و فرار از پاسخ سیستم ایمنی میزبان را تسهیل میکند [9]. پروتئین nsp2 در تنظیم مسیر انتقال پیام بقای سلولها با واکنش بین مولکولهای PHB و PHB2 میزبان نقش دارد. این دو پروتئین نقش کلیدی در پایداری عملکرد میتوکندری و حفظ سلول از استرسها ایفا میکنند [10]. پروتئین nsp3 در شکاف توالی انتهایی N در پلیپروتئینها نقش دارد. در کنار این پروتئین، PL-PRO دارای فعالیت deubiquitinating یا deISGylating است که در مهار پاسخ ایمنی نقش دارد و زنجیرههای متصل پلییوبیکوتین به Lys63 و 48Lys در سوبستراهای سلولی را هدف قرار میدهد [11]. این پروتئین به همراه پروتئین nsp4 در تشکیل وزیکولهای دوغشایی ضروری برای تکثیر ویروس نیز نقش دارد. پروتئین nsp3 با بلوکه کردن فسفوریلاسیون، دیمریزاسیون و انتقال بین جایگاهی هسته سلولها، سبب مهار القای اینترفرون تیپ 1 از ایمنی ذاتی میشود. این پروتئین در مهار انتقال پیام NF-kappa-B نقش دارد [13 ،12]. در ناحیه ژن کدکننده nsp3 توالی دُمین SUD وجود دارد که فقط در کروناویروسهای تیپ سارس دیده میشود که با اتصال به mRNA G4 در مهار انتقال پیام آپوپتوز و بقای سلولها نقش دارد [14]. پروتئیناز 3CL در شکاف توالی انتهایی C پلیپروتئین replicase در یازده ناحیه نقش دارد (تصویر شماره 5).
سوبستراهای شناختهشده برای این پروتئین حاوی توالی [ILMVF]-Q-|-[SGACN] هستند. این پروتئین همچنین به ADRP نیز متصل میشود [15].
پروتئین nsp6 در القای اولیه اتوفاگوزوم شبکه آندوپلاسمی میزبان و سپس محدود کردن بسط این فاگوزومها و تسهیل در انتقال اجزای ویروسی به لیزوزومها نقش دارد [16]. پروتئین nsp7 با پروتئین nsp8 یک هگزا دکامر تشکیل میدهد که به عنوان یک پریماز در تکثیر ویروس نقش دارد [17]. پروتئین nsp9 نیز به عنوان پروتئین اتصالی به ssRNA در تکثیر ویروس نقش دارد [18]. پروتئین nsp10 با همزمانسازی فعالیتهای 3’-5’ اگزوریبونوکلئازی پروتئین nsp14 و 2’-O-متیل ترانسفراز پروتئین nsp16 نقش حیاتی در سیستم ترجمه ویروس دارد. این پروتئین در متیلاسیون Cap توالی mRNAهای ویروسی نقش دارد [19]. پروتئین RdRp یا nsp12 همراه با کوفاکتورهای nsp7 و nsp8 در تکثیر و ترجمه ژنوم RNA ویروس نقش دارد [20]. پروتئین هلیکاز (Hel) یک پروتئین چندعملکردی با دُمین اتصالی در ناحیه انتهایی N است. این پروتئین در باز کردن مارپیچ دوگانه RNA و DNA از ناحیه 5’به 3’ نقش دارد. فعالیت پروتئین هلیکاز وابسته به یون منیزیم است [22 ،21]. ناحیه S ژنوم ویروس، گلیکوپروتئین spike را کد میکند که در سطح ویروس قرار دارد و نقش مهمی در اتصال ویروس به میزبان و بیماریزایی ویروس بازی میکند. این پروتئین به گیرنده hACE2 متصل میشود. بر اساس مطالعات مولکولی، این گیرنده در اندامهای مختلف بدن نیز شناسایی شده است که در تروپیسم ویروس نقش دارد (تصویر شماره 6) و موجب بروز علائم قلبی، گوارشی و کلیوی در کنار علائم تنفسی میشود [23].
پروتئین nsp6 در القای اولیه اتوفاگوزوم شبکه آندوپلاسمی میزبان و سپس محدود کردن بسط این فاگوزومها و تسهیل در انتقال اجزای ویروسی به لیزوزومها نقش دارد [16]. پروتئین nsp7 با پروتئین nsp8 یک هگزا دکامر تشکیل میدهد که به عنوان یک پریماز در تکثیر ویروس نقش دارد [17]. پروتئین nsp9 نیز به عنوان پروتئین اتصالی به ssRNA در تکثیر ویروس نقش دارد [18]. پروتئین nsp10 با همزمانسازی فعالیتهای 3’-5’ اگزوریبونوکلئازی پروتئین nsp14 و 2’-O-متیل ترانسفراز پروتئین nsp16 نقش حیاتی در سیستم ترجمه ویروس دارد. این پروتئین در متیلاسیون Cap توالی mRNAهای ویروسی نقش دارد [19]. پروتئین RdRp یا nsp12 همراه با کوفاکتورهای nsp7 و nsp8 در تکثیر و ترجمه ژنوم RNA ویروس نقش دارد [20]. پروتئین هلیکاز (Hel) یک پروتئین چندعملکردی با دُمین اتصالی در ناحیه انتهایی N است. این پروتئین در باز کردن مارپیچ دوگانه RNA و DNA از ناحیه 5’به 3’ نقش دارد. فعالیت پروتئین هلیکاز وابسته به یون منیزیم است [22 ،21]. ناحیه S ژنوم ویروس، گلیکوپروتئین spike را کد میکند که در سطح ویروس قرار دارد و نقش مهمی در اتصال ویروس به میزبان و بیماریزایی ویروس بازی میکند. این پروتئین به گیرنده hACE2 متصل میشود. بر اساس مطالعات مولکولی، این گیرنده در اندامهای مختلف بدن نیز شناسایی شده است که در تروپیسم ویروس نقش دارد (تصویر شماره 6) و موجب بروز علائم قلبی، گوارشی و کلیوی در کنار علائم تنفسی میشود [23].
گلیکوپروتئین spike دارای دو زیرواحد S1برای اتصال به گیرنده و پروتئین S2 برای فیوژن یا همجوشی پوشش ویروس با غشای سلول میزبان است. گلیکوپروتئین S در محل شکاف فورین دارای چند اسیدآمینه بازی (PRRA) در محل اتصال زیرواحد S1 وS2 است که عفونتزایی ویروس را افزایش میدهد. پروتئین S1 دارای دُمین اتصال به گیرنده (RBD) است که در اتصال به گیرنده hACE2 و ورود ویروس به سلول میزبان نقش دارد و سبب القای تغییرات ساختاری گلیکوپروتئین S میشود (تصویر شماره 7).
این پروتئین در ریه انسان با استفاده از پروتئاز بین غشایی سرین 2 (TMPRSS2) سبب ورود ویروس به سلولهای ریه میشود. پروتئولیز گلیکوپروتئین S توسط کاتپسین CatB/L سبب فعالیت پپتید فیوژن S2 و فعال شدن ادغام غشایی ویروس درون اندوزومها میشود. مطالعات درباره جهش در توالی RBD نشان دادند N501 جهش ندارد و با اتصال به Y41 توسط پیوند هیدروژنی سبب پایداری اتصال RBD به گیرنده hACE2میشود و این ناحیه به دلیل حالتهای چندگانه اتصال از عملکرد پیچیدهای برخوردار است. در کنار اینها، مطالعات نشان دادند جهشهای N479 ،T487، L455A ،F456A ،Q493A سبب اتصال ضعیف به گیرنده hACE2 خواهند شد [23، 24، 25، 26 ،4].
پروتئین Ns3a در ایجاد ویروپورین و احتمالاً آزادسازی ویروس نقش دارد. این پروتئین سبب بیان بالادست زیرواحدهای فیبرینوژن FGA ،FGB و FGG در سلولهای اپیتلیال ریه میزبان میشود. این پروتئین در کشت سلول سبب القای آپوپتوز میشود. Ns3a با القای فسفوریلاسیون سرین در زیرواحد 1 گیرنده اینترفرون آلفا (IFNAR1) سبب بیان پایین گیرنده تیپ 1 اینترفرون و افزایش ubiquitination در IFNAR1 میشود [27، 28، 29 ،30].
پروتئین E نقش محوری در مورفوژنز و تشکیل ساختار ویروس دارد. این پروتئین به عنوان ویروپورین با منافذ پنتامری لیپیدی پروتئینی امکان عبور یونها را فراهم میکند. این پروتئین در القای آپوپتوز نقش دارد. مطالعات مقایسهای همردیفی، توالیهای ژنومی این پروتئین کروناویروس در خفاش و مورچهخوار پولکدار را نشان میدهند. پروتئین E با یک جانشینی در جایگاه 69 (R69N/D/E)، حذف در جایگاه 70 (70G/C) و جانشینی در جایگاه 55 و 56 (T,V-> S,F) متحمل تغییرات ساختاری شده که سبب شده این پروتئین کوچک نقش مهمی در مراحل عفونت و تکثیر ویروس داشته باشد [31]. گلیکوپروتئین M در مورفوژنز و کنار هم قرار گرفتن اجزای مختلف ویروس نقش دارد و در جوانهزنی ویروس نقش مهمی ایفا میکند (تصویر شماره 8).
پروتئین Ns3a در ایجاد ویروپورین و احتمالاً آزادسازی ویروس نقش دارد. این پروتئین سبب بیان بالادست زیرواحدهای فیبرینوژن FGA ،FGB و FGG در سلولهای اپیتلیال ریه میزبان میشود. این پروتئین در کشت سلول سبب القای آپوپتوز میشود. Ns3a با القای فسفوریلاسیون سرین در زیرواحد 1 گیرنده اینترفرون آلفا (IFNAR1) سبب بیان پایین گیرنده تیپ 1 اینترفرون و افزایش ubiquitination در IFNAR1 میشود [27، 28، 29 ،30].
پروتئین E نقش محوری در مورفوژنز و تشکیل ساختار ویروس دارد. این پروتئین به عنوان ویروپورین با منافذ پنتامری لیپیدی پروتئینی امکان عبور یونها را فراهم میکند. این پروتئین در القای آپوپتوز نقش دارد. مطالعات مقایسهای همردیفی، توالیهای ژنومی این پروتئین کروناویروس در خفاش و مورچهخوار پولکدار را نشان میدهند. پروتئین E با یک جانشینی در جایگاه 69 (R69N/D/E)، حذف در جایگاه 70 (70G/C) و جانشینی در جایگاه 55 و 56 (T,V-> S,F) متحمل تغییرات ساختاری شده که سبب شده این پروتئین کوچک نقش مهمی در مراحل عفونت و تکثیر ویروس داشته باشد [31]. گلیکوپروتئین M در مورفوژنز و کنار هم قرار گرفتن اجزای مختلف ویروس نقش دارد و در جوانهزنی ویروس نقش مهمی ایفا میکند (تصویر شماره 8).
مطالعات دارویی برای مهار ویروس SARS-COV-2
در یافتن درمان بیماری کووید 19، شناسایی ویژگیهای ساختاری ویروس و بیولوژی ویروس امری ضروری است. در کنار مطالعات ویروسشناسی، مطالعات بالینی و درمانی در مقابله با ویروس نیز از اهمیت زیادی برخوردار است. استراتژیهای دارویی متعددی در مقابله با این ویروس و عدم تکثیر آن به کار گرفته شدهاند در این مقاله برخی از آنها بررسی شدهاند.
ایمونوگلوبولیندرمانی
مطالعات در حوزه ایمونوگلوبولیندرمانی درونرگی نشان دادند استفاده از آنتیبادی IgG در مراحل اولیه بیماری با دُز بالا و در بیماران دارای وضعیت وخیم سبب کاهش پاسخهای التهابی میشود که با بهبود عملکرد اندامها همراه است و منجر به کاهش مرگومیر میشود [33].
ترکیبات آمینوکینولین
یکی از اهداف مهم ویروس در اتصال به سلولهای بدن، گیرنده hACE2 است. گلیکوپروتئین S ویروس برای اتصال به گیرنده hACE2 از دو پروتئاز سلولی استفاده میکند. پروتئاز سیستئینی CatB/L از پروتئازهای اندوزومی محسوب میشود که شامل کاتپسین B و L است که در pH پایین فعال میشوند. پروتئاز TMPRRS2 یک پروتئاز سرینی بین غشایی تیپ 2 است که در ریه و پروستات به میزان زیادی بیان میشود [34]. در کنار اینها، مطالعات اخیر نشان میدهند پروتئاز TMPRRS2 و گیرنده hACE2 به میزان زیادی در ملتحمه چشم نیز بیان میشوند که به عنوان یکی از راههای ورود ویروس به بدن در نظر گرفته میشوند[35]. این دو پروتئاز سلولی در پروتئولیز گلیکوپروتئین S و فعال شدن زیرواحد S2 در ادغام غشایی ویروس با سلول میزبان و زیرواحد S1(RBD) در اتصال به گیرنده hACE2 نقش دارند. داروی کلروکین یک ترکیب قلیایی ضعیف است. این دارو از طریق بخش غیرپروتونی از غشای سلول عبور میکند و در ارگانلهای دارای شرایط اسیدی و pH پایین تجمع کرده و پروتونه میشود. این دارو pH وزیکولهای اسیدی را افزایش میدهد. بدینترتیب مانع فعال شدن کاتپسین B و L اندوزومی میشود. در سلولهای طبیعی گیرندههای hACE2 به دو شکل در سلول ساخته میشوند که شامل گیرنده hACE2 تغییریافته در شبکه آندوپلاسمی (ACE2-ER) و گیرنده hACE2 تغییریافته در گلژی (ACEII-Golgi) هستند. این دارو در گلیکوزیلاسیون گیرنده hACE2 اختلال ایجاد میکند و بیشتر با شکل ACEII-ER همراه است. بر اثر اختلال در گلیکوزیلاسیون گیرنده، اتصال مؤثر زیرواحد گلیکوپروتئین S1 ویروس با گیرنده hACE2 شکل نمیگیرد [35]. این دارو برای بیماریهایی نظیر مالاریا، آمیبیوزیس و نیز بیماریهای خودایمن استفاده میشود. میزان حاشیه درمانی و سمی این دارو به هم نزدیک بوده و از عوارض جانبی مهم دارو اختلالات قلبی و عروقی است. مصرف این دارو در ابتدای بیماری توصیه میشود؛ زیرا با اندوسیتوز ویروس به همراه گیرنده سلول کاهشی در میزان گیرنده hACE2 ایجاد میشود که منجر به افزایش میزان AngII در خون میشود که علاوه بر انقباض عروقی، به عنوان سایتوکاین پیشالتهابی همراه با AT1R سبب فعال شدن مسیر NF-kB و آزادسازی بیشتر سایتوکاینهای التهابی میشود [36].
مهارکنندههای پروتئازهای سلولی
یکی از استراتژیهای درمانی بیماری مهار پروتئازهای مؤثر در اتصال ویروس به گیرنده hACE2 است. داروهای متعددی در مهار پروتئازهای سیستئینی و پروتئازهای سرینی شناسایی شدهاند. از داروهای مؤثر در مهار پروتئازهای سرینی به خصوص TMPRSS2 داروی کاموستات را میتوان نام برد که با اتصال به جایگاههای فعال آنزیم (H296 و S441) سبب مهار فعالیت این پروتئاز میشود (تصویر شماره 9 الف).
در یافتن درمان بیماری کووید 19، شناسایی ویژگیهای ساختاری ویروس و بیولوژی ویروس امری ضروری است. در کنار مطالعات ویروسشناسی، مطالعات بالینی و درمانی در مقابله با ویروس نیز از اهمیت زیادی برخوردار است. استراتژیهای دارویی متعددی در مقابله با این ویروس و عدم تکثیر آن به کار گرفته شدهاند در این مقاله برخی از آنها بررسی شدهاند.
ایمونوگلوبولیندرمانی
مطالعات در حوزه ایمونوگلوبولیندرمانی درونرگی نشان دادند استفاده از آنتیبادی IgG در مراحل اولیه بیماری با دُز بالا و در بیماران دارای وضعیت وخیم سبب کاهش پاسخهای التهابی میشود که با بهبود عملکرد اندامها همراه است و منجر به کاهش مرگومیر میشود [33].
ترکیبات آمینوکینولین
یکی از اهداف مهم ویروس در اتصال به سلولهای بدن، گیرنده hACE2 است. گلیکوپروتئین S ویروس برای اتصال به گیرنده hACE2 از دو پروتئاز سلولی استفاده میکند. پروتئاز سیستئینی CatB/L از پروتئازهای اندوزومی محسوب میشود که شامل کاتپسین B و L است که در pH پایین فعال میشوند. پروتئاز TMPRRS2 یک پروتئاز سرینی بین غشایی تیپ 2 است که در ریه و پروستات به میزان زیادی بیان میشود [34]. در کنار اینها، مطالعات اخیر نشان میدهند پروتئاز TMPRRS2 و گیرنده hACE2 به میزان زیادی در ملتحمه چشم نیز بیان میشوند که به عنوان یکی از راههای ورود ویروس به بدن در نظر گرفته میشوند[35]. این دو پروتئاز سلولی در پروتئولیز گلیکوپروتئین S و فعال شدن زیرواحد S2 در ادغام غشایی ویروس با سلول میزبان و زیرواحد S1(RBD) در اتصال به گیرنده hACE2 نقش دارند. داروی کلروکین یک ترکیب قلیایی ضعیف است. این دارو از طریق بخش غیرپروتونی از غشای سلول عبور میکند و در ارگانلهای دارای شرایط اسیدی و pH پایین تجمع کرده و پروتونه میشود. این دارو pH وزیکولهای اسیدی را افزایش میدهد. بدینترتیب مانع فعال شدن کاتپسین B و L اندوزومی میشود. در سلولهای طبیعی گیرندههای hACE2 به دو شکل در سلول ساخته میشوند که شامل گیرنده hACE2 تغییریافته در شبکه آندوپلاسمی (ACE2-ER) و گیرنده hACE2 تغییریافته در گلژی (ACEII-Golgi) هستند. این دارو در گلیکوزیلاسیون گیرنده hACE2 اختلال ایجاد میکند و بیشتر با شکل ACEII-ER همراه است. بر اثر اختلال در گلیکوزیلاسیون گیرنده، اتصال مؤثر زیرواحد گلیکوپروتئین S1 ویروس با گیرنده hACE2 شکل نمیگیرد [35]. این دارو برای بیماریهایی نظیر مالاریا، آمیبیوزیس و نیز بیماریهای خودایمن استفاده میشود. میزان حاشیه درمانی و سمی این دارو به هم نزدیک بوده و از عوارض جانبی مهم دارو اختلالات قلبی و عروقی است. مصرف این دارو در ابتدای بیماری توصیه میشود؛ زیرا با اندوسیتوز ویروس به همراه گیرنده سلول کاهشی در میزان گیرنده hACE2 ایجاد میشود که منجر به افزایش میزان AngII در خون میشود که علاوه بر انقباض عروقی، به عنوان سایتوکاین پیشالتهابی همراه با AT1R سبب فعال شدن مسیر NF-kB و آزادسازی بیشتر سایتوکاینهای التهابی میشود [36].
مهارکنندههای پروتئازهای سلولی
یکی از استراتژیهای درمانی بیماری مهار پروتئازهای مؤثر در اتصال ویروس به گیرنده hACE2 است. داروهای متعددی در مهار پروتئازهای سیستئینی و پروتئازهای سرینی شناسایی شدهاند. از داروهای مؤثر در مهار پروتئازهای سرینی به خصوص TMPRSS2 داروی کاموستات را میتوان نام برد که با اتصال به جایگاههای فعال آنزیم (H296 و S441) سبب مهار فعالیت این پروتئاز میشود (تصویر شماره 9 الف).
از مهارکنندههای پروتئازهای سیستئنی در مهار کاتپسینها داروهای اپوکسی سوسکسینیل پپتید (EST) و آزا-پپتید اپوکسید (AZP) نیز شناسایی شدهاند که به صورت برگشتناپذیر به پروتئازهای سیستئینی متصل شده و سبب مهار فعالیت آنها میشوند [37]. داروی مقلد پپتیدی AZP به عنوان مهارکننده پروتئین3CLpro نیز شناسایی شده است (تصویر شماره 9 ب).
مهارکنندههای پروتئین S
مطالعات دارویی بر اساس آنالیز محل اتصال داروهای مختلف با گلیکوپروتئین S نشان دادند داروی آربیدول با جلوگیری از تریمریزاسیون S2 در پروتئین Spike مانع اتصال ویروس به سلول میزبان میشود [38]. مطالعات بالینی نیز نشان دادند این دارو به عنوان درمان پیشگیریکننده از ابتلا به ویروس مؤثر بوده است [39]. از پپتیدهای سنتتیک، پپتید EK1C4 شناسایی شده است که با اتصال مارپیچهای آلفا 6-HB میتواند در مهار اتصال ویروس به گیرنده hACE2 مؤثر باشد [40] (تصویر شماره 10).
مهارکنندههای پروتئین S
مطالعات دارویی بر اساس آنالیز محل اتصال داروهای مختلف با گلیکوپروتئین S نشان دادند داروی آربیدول با جلوگیری از تریمریزاسیون S2 در پروتئین Spike مانع اتصال ویروس به سلول میزبان میشود [38]. مطالعات بالینی نیز نشان دادند این دارو به عنوان درمان پیشگیریکننده از ابتلا به ویروس مؤثر بوده است [39]. از پپتیدهای سنتتیک، پپتید EK1C4 شناسایی شده است که با اتصال مارپیچهای آلفا 6-HB میتواند در مهار اتصال ویروس به گیرنده hACE2 مؤثر باشد [40] (تصویر شماره 10).
آنالوگهای نوکلئوزیدی
یکی از پروتئینهای مهم در تکثیر و ساخت ژنوم ویروس، پروتئین RdRp یا nsp12 است. این پروتئین دارای فعالیت اگزونوکلئازی است و سنتز RNA را با دو مکانیسم وابسته به پرایمر و غیروابسته به پرایمر با استفاده از الگوهای RNA هوموپلیمریک انجام میدهد. این پروتئین ترجیحاً از الگوهای هوموپلیمریک پیریمیدینی برای ساخت ژنوم و تکثیر ویروس استفاده میکند [20]. از داروهای مهم در مهار این پروتئین میتوان آنالوگهای نوکلئوزیدی را نام برد. داروی رمدسیویر یک آنالوگ آدنوزینی است که با قرار گرفتن در زنجیره ژنوم RNA سبب خاتمه در همانندسازی ژنوم ویروس و ساخته شدن ناقص ژنوم ویروس میشود. داروی فاویپیراویر یک ترکیب ضدویروسی است که به عنوان یک آنالوگ آدنوزینی یا گوانوزینی در خاتمه سنتز ژنوم ویروس نقش دارد. این دارو به دلیل دارا بودن ریبوفورانوزیل تریفسفات اثر جهشزایی بر ژنوم ویروس دارد [41]. از دیگر داروهای مؤثر این دسته در مهار ویروس داروی هیدروکسی سیستیدیل (β-D-N4-hydroxycytidine) است (تصویر شماره 11).
یکی از پروتئینهای مهم در تکثیر و ساخت ژنوم ویروس، پروتئین RdRp یا nsp12 است. این پروتئین دارای فعالیت اگزونوکلئازی است و سنتز RNA را با دو مکانیسم وابسته به پرایمر و غیروابسته به پرایمر با استفاده از الگوهای RNA هوموپلیمریک انجام میدهد. این پروتئین ترجیحاً از الگوهای هوموپلیمریک پیریمیدینی برای ساخت ژنوم و تکثیر ویروس استفاده میکند [20]. از داروهای مهم در مهار این پروتئین میتوان آنالوگهای نوکلئوزیدی را نام برد. داروی رمدسیویر یک آنالوگ آدنوزینی است که با قرار گرفتن در زنجیره ژنوم RNA سبب خاتمه در همانندسازی ژنوم ویروس و ساخته شدن ناقص ژنوم ویروس میشود. داروی فاویپیراویر یک ترکیب ضدویروسی است که به عنوان یک آنالوگ آدنوزینی یا گوانوزینی در خاتمه سنتز ژنوم ویروس نقش دارد. این دارو به دلیل دارا بودن ریبوفورانوزیل تریفسفات اثر جهشزایی بر ژنوم ویروس دارد [41]. از دیگر داروهای مؤثر این دسته در مهار ویروس داروی هیدروکسی سیستیدیل (β-D-N4-hydroxycytidine) است (تصویر شماره 11).
این دارو یک آنالوگ ریبونوکلئوزیدی با فعالیت ضدویروسی گسترده علیه RNA ویروسهای غیرمرتبط نظیر آنفلوانزا، ابولا، سارس و آنسفالیت ونزوئلایی است. این ترکیب از طریق جهشزایی مرگبار با ایجاد جهشهای حذفی در ژنوم ویروس و تجمع آنها در ژنوم سبب مهار تکثیر ویروس میشود [42].
مهارکنندههای پروتئازهای ویروسی
از پروتئازهای ویروسی مهم در شکاف پلیپروتئینهای ویروسی، پروتئین 3CLpro یا Mpro و پروتئین PLpro است. این پروتئینها از خانواده پروتئازهای سیستئینی محسوب میشوند. مطالعات ژنومی نشان دادهاند در پروتئاز اصلی ویروس (Mpro) جایگزینی Thr285 با Ala و Ile286 با Leu سبب تقویت فعالیت کاتالیتیکی این پروتئین شده است. این پروتئین از سه دُمین تشکیل میشود. محل اتصال سوبسترا در این پروتئین بین دُمینهای I و II قرار دارد. این پروتئاز دارای دو پروتومر است. در هرکدام از این پروتومرها، بخشی به نام بخش انتهایی N-Finger بین دُمینهای II و III قرار دارد که همراه با دُمین II از پروتومر دیگر در تنظیم دیمریزاسیون نقش مهمی دارند. این پروتئاز دارای گروههای دوگانه کاتالیتیک His41 و Cys145 است که اسیدآمینه سیستئین به عنوان نوکلئوفیل مشترک در فرآیند پروتئولیتیک پروتئاز عمل میکند (تصویر شماره 12 الف).
مهارکنندههای پروتئازهای ویروسی
از پروتئازهای ویروسی مهم در شکاف پلیپروتئینهای ویروسی، پروتئین 3CLpro یا Mpro و پروتئین PLpro است. این پروتئینها از خانواده پروتئازهای سیستئینی محسوب میشوند. مطالعات ژنومی نشان دادهاند در پروتئاز اصلی ویروس (Mpro) جایگزینی Thr285 با Ala و Ile286 با Leu سبب تقویت فعالیت کاتالیتیکی این پروتئین شده است. این پروتئین از سه دُمین تشکیل میشود. محل اتصال سوبسترا در این پروتئین بین دُمینهای I و II قرار دارد. این پروتئاز دارای دو پروتومر است. در هرکدام از این پروتومرها، بخشی به نام بخش انتهایی N-Finger بین دُمینهای II و III قرار دارد که همراه با دُمین II از پروتومر دیگر در تنظیم دیمریزاسیون نقش مهمی دارند. این پروتئاز دارای گروههای دوگانه کاتالیتیک His41 و Cys145 است که اسیدآمینه سیستئین به عنوان نوکلئوفیل مشترک در فرآیند پروتئولیتیک پروتئاز عمل میکند (تصویر شماره 12 الف).
بیشتر جایگاههای شکاف این پروتئین مانند سایر پروتئازهای 3CLpro کروناویروسها دارای توالی حفاظتشده Gln↓(Ser, Ala, Gly) هستند که تا کنون هیچ پروتئاز انسانی با چنین جایگاه شکاف شناخته نشده است. این پروتئازهای ویروسی دارای گروهای تیولی در جایگاه فعال هستند. داروی لوپیناویر از داروهای مقلد پپتیدی است که با اتصال به جایگاههای نزدیک به جایگاه کاتالیتیکی پروتئاز 3CLpro سبب مهار فرایندهای پروتئولیتیکی ویروس میشود. این دارو از طریق پیوند هیدروژنی به گروه هیدروکسیل اسیدآمینه Ser46 و به اسیدهای آمینه Met49 ،Asn142 و Glu166 از طریق اتصالات هیدروفوبیک متصل میشود (تصویر شماره 12 ب). این دارو به همراه داروی ریتوناویر مورد استفاده قرار میگیرد. داروی ریتوناویر یک پپتید متقارن است و به عنوان داروی مهارکننده پروتئاز ویروسی کاربرد دارد. این دارو به دلیل ساختار متقارن، فراهم زیستی پایینی دارد. ریتوناویر بالقوه قابلیت مهار فعالیت متابولیسمی وابسته به سیتوکروم P450 (CYP3A) را دارد و به عنوان داروی ترکیبی به منظور عدم تجزیه داروهای مهارکننده پروتئازهای ویروسی و بالا رفتن میزان دارو در خون به کار برده میشود [43].
داروی نلفیناویر یک داروی غیرپپتیدی ضدویروسی است. این دارو به واسطه ایجاد پیوندهای هیدروژنی زیاد سبب مهار فعالیت پروتئازی ویروس میشود. این دارو نسبت به لوپیناویر اثر بیشتری در مهار ویروس دارد. این دارو از طریق پیوند هیدروژنی با اسیدهای آمینه Glu166 ،ASN142 (گروه آمیدی) و Thr26 (گروه هیدروکسیل) و از طریق پیوند هیدروژنی N-H…N با ایمیدازول اسید آمینه His41 سبب مهار این پروتئاز میشود. در مطالعات بالینی استفاده از این دارو به همراه سفارانتین در مهار ویروس و بیماری مؤثر بوده است (تصویر شماره 12 ج). این دارو در مهار سایتوکاینهای التهابی حاصل از بیماری نقش مؤثری دارد [44، 45].
پروتئازهای ویروس دارای گروههای تیول هستند. یکی از استراتژیهای دارویی در مهار ویروس، TOS II است. دارو با اتصال غیرکوالانسی فعالیت گروههای تیولی پروتئینهای سیتوزولی را مهار میکند. با اکسیداسیون تیول / تیولات به دیسولفید فعالیت پروتئازی ویروس مهار میشود. از مهارکنندههای آلفا کتوآمید ترکیب دارویی با نام 13b شناسایی شده است که با اتصال به محل سوبسترا در سطح پروتومرهای بین دُمینهای I و II و تهاجم نوکلئوفیلیک به Cys145 با گروه آلفا کتو (تشکیل ساختار همیکتال) و His41 با گروه اکسی آنیون (هیدروکسیل) سبب مهار پروتئاز ویروس و شکلگیری ویریون عفونی ویروس میشود (تصویر شماره 12 ب). این ترکیب با مرکز کاتالیتیک پروتئاز ویروس، دو پیوند هیدروژنی برقرار میکند. این ترکیب به دلیل داشتن گروه ترت بوتیل اکسیکربونیل (Boc) از غشای سلول عبور میکند و به خوبی در بافتهای بدن از جمله ریه منتشر میشود. این ترکیب علاوه بر استفاده خوراکی، بهصورت استنشاقی نیز قابل مصرف است [43].
پروتئینهای سنتزی ACE2
گیرنده ACE2 از پروتئینهای مهم در ورود ویروس به سلول میزبان است. پروتئینهای سنتزی ACE2 به صورت محلول (sACE2) هستند. در این پروتئینها بخش بین غشایی پروتئین وجود ندارد. این پروتئین قابلیت اتصال با FC ایمونوگلوبولین انسانی را دارد که میتواند اویدیتی مناسبی در زمان فراخوانی سلولهای فعال ایمنی فراهم کند و پایداری ایمونوگلوبولین را در سرم افزایش میدهد. از پروتئینهای نوترکیب، پروتئین sACE2.v2.4 بر اساس جهشهای جایگزینی T27Y ،L79T و N330Y در ساختار پروتئین ساخته شده است که به دلیل شباهت زیاد به پروتئین اصلی، ایمنیزایی کمتری ایجاد میکند و مزیت آن نسبت به آنتیبادیهای مونوکلونال، عدم ایجاد تشدید عفونت به واسطه آنتیبادی (ADE) است [46، 47].
مهارکنندههای فاکتورهای میزبانی
شناسایی عوامل میزبان در تکثیر ویروس و تشدید عفونت در کنترل عفونتهای ویروسی از اهمیت زیادی برخوردار است. با تجزیه و تحلیل دادههای حاصل از آزمایشات AP-MS و DIA-MS به ترتیب شبکه برهمکنش پروتئینهای میزبان (PPI) با پروتئینهای ویروس و نیز فسفریلاسیون پروتئینهای ویروس توسط میزبان مشخص شد. در دادههای حاصل از PPI، 332 پروتئین مؤثر میزبانی شناسایی شدند. در این مطالعه دو کلاس از داروهای مولکولی شناسایی شد که با مهار عوامل میزبانی سبب اختلال در روند تکثیر ویروس در سلول میشوند. مهارکنندههای بیوژنز پروتئینی (zotatifin ،ternatin-4 و PS3061) با مهار فاکتور elF4H سبب اختلال در ترجمه mRNA ویروسی میشوند و درنتیجه سطح مناسبی از پروتئینهای ویروسی ساخته نمیشود. از دیگر داروهای مولکولی، لیگاندهای گیرنده Sigma-1 و Sigma-2 هستند که با جلوگیری از میانکنش این گیرندهها با پروتئینهای ویروسی (ORF9c و NSP6) مانع تکثیر ویروس میشوند. این لیگاندها شامل haloperidol ،PB28 و hydroxychloroquine هستند [48]. بررسی بیشتر دادههای PPI نشان داد پروتئین ویروسی NSP13 نقش مؤثری در تنظیم شرایط متابولیسمی مناسب برای تکثیر ویروس دارد. در مطالعه دیگری، نقش نمکهای بیسموت در مهار پروتئین NSP13 بررسی شد. این مطالعه نشان داد نمکهای بیسموت پتاسیم سیترات (BPC) و رانیتیدین بیسموت سیترات (RBC) که به عنوان داروهای بیماریهای گوارشی مصرف میشود نسبت به بیسموت سیترات (BC) در مهار فعالیت NTPase ،ATPase و مهار فعالیت باز کردن پیچش ژنومی (Unwinding) پروتئین NSP13 اثر بیشتری دارد [49]. آزمایش روی فسفریلاسیون پروتئینهای ویروسی، 49 جایگاه فسفریلاسیون روی هفت پروتئین ویروسی M ،N (بیشترین تعداد)، NSP14 ،NSP13 و NSP9 نشان داد. در این مطالعه، فعالیت کازئین کیناز 2 (CK2)، فعالسازی مسیر p38 MAPK و سرکوب کینازهای میتوزی با فسفریلاسیون پروتئینهای ویروس، در توقف چرخه سلولی نقش دارند. پروتئین N ویروس در تنظیم فعالیت CK2 و سازماندهی اسکلت سلولی و جوانهزنی ذرات ویروس نقش مؤثری دارد. در این مطالعه داروهای مولکولی Silmitasertib (مهار CK2، فاز 2 بالینی)، gilteritinib (مهار AXL) ARRY-797 (مهار p38، فاز 2 یا 3 بالینی) شناسایی شدند [50].
مهارکنندههای مولکولی نوکلئیک اسیدی
از ویژگیهای مهم ژنوم ویروس SARS-CoV-2 میانکنشهای RNA-RNA با فواصل بلند و کوتاه در ژنوم ویروس و میانکنش ژنوم RNA ویروسی با RNAهای کوچک هستهای (snRNA) و بلند سلول میزبان است که در تکثیر و رونویسی ناپیوسته ژنوم ویروس نقش دارند. میانکنش snRNAها بیشتر با ژنوم ویروس (gRNA) صورت میگیرد که از طریق محلهای اختصاصی اتصال (ssb) در U2 ،U1 و U4 به نواحی ORF1a و ORF1b ژنوم ویروس متصل میشوند. رونوشتهای ژنوم ویروس (sgmRNA) از نواحی N و 3’UTR نیز به میزان زیادی با محلهای اختصاصی اتصال U1 و U2 میانکنش دارند. از میانکنش ژنوم ویروس با RNAهای بلند سلول میزبان، RNAase MRP از سلول میزبان شناسایی شد که در تجزیه RNA ویروسی نقش دارد و جهش در آن سبب بیماریهای انسانی از جمله بیماری هیپوپلازی مو غضروف میشود. از میانکنشهای RNA-RNA ژنوم ویروس و رونوشت آن در ناحیه 5’UTR پنج ناحیه SL1-SL5 و در 3’UTR سه ناحیه BSL ،hairpin-type pseudoknot و مارپیچ سهگانه اتصال شناسایی شد که در تکثیر و رونویسی ناپیوسته نقش دارند. با میانکنش RNA-RNA بین ناحیه مارپیچ سهگانه اتصال در 3’-UTR و ناحیه SL-3 در 5’-UTR، ژنوم ویروس، حلقوی میشود که در تنظیم رونوشتبرداری به صورت ناپیوسته نقش دارد [51]. ناحیه SL1 5’UTR به همراه NSP1، در ترجمه پروتئینهای ویروسی نقش اساسی دارد و در درمان با الیگونوکلئوتیدهای آنتیسنس (ASO) به عنوان پاشنه آشیل مهار ترجمه پروتئینهای ویروسی در نظر گرفته میشود [52]. آنالیزهای دارویی نشان دادند گلیسیرریزین، لوباریک اسید، گارسینولیک اسید و تریلازاد با اتصال به پروتئین NSP1 سبب مهار تشکیل کمپلکس NSP1/SL1 میشوند [53]. ناحیه s2m 3’UTR در ژنوم ویروس یک ناحیه حفاظتشده است که میانکنش آن با ORF1a شناسایی شده است. استفاده از الیگونوکلئوتیدهای LAN-Gapmer نشان داد اتصال مولکولهای Gapmer به ناحیه s2m سبب تجزیه مولکولهای mRNA ویروس توسط RNase H سلول میزبان میشود [54]. کاربرد درمانی این مولکولها (LAN-Gapmer) در کنار پروتئینهای نوترکیب hACE2 به صورت استنشاقی نیز مطرح شده است [55]. مطالعات روی مدلهای حیوانی نشان دادند از عوامل تداخل RNA (RNAi) ژنهای مختلف ویروس، مولکولهای siRNA طراحیشده بر اساس ژن (siRNA-N14 R7) RdRp در مهار تکثیر ویروس مؤثر است [56]. در کنار پایداری پایین مولکولهای siRNA، از محدودیت این مولکولها، سرکوب توسط پروتئین نوکلئوکپسید ویروس است که به عنوان سرکوبکننده ویروسی عوامل تداخل RNA (VSR) شناسایی شده است [57]. از مولکولهای RNA کاتالیتیک (ریبوزیم)، ریبوزیم نوترکیب DNA-RNA Hammerhead در بیماری سارس شناسایی شده است که به طور اختصاصی با اتصال به ناحیه ORF1ab(15460 GUC) سبب تجزیه ژنوم ویروس بیماری سارس میشود. این مولکولها به طور اختصاصی به مولکول RNA هدف متصل و سبب تجزیه آن میشوند، اما در ASO و RNAi تجزیه RNA هدف توسط RNase میزبان صورت میگیرد [57].
نتیجهگیری
مطالعه حاضر نشان میدهد جهت کنترل این ویروس، بررسی ساختار ویروس و بیولوژی آن در بدن از اهمیت زیادی برخوردار است. شناسایی داروهای مؤثر بر ویروس با توجه به ساختمان بیولوژیک ویروس ضروری است. با توجه به تغییرات ساختاری ویروس و جهشهای پیدرپی در ژنوم ویروس و به وجود آمدن سویههای مقاوم یا سویههای با قابلیت واگیری زیاد، ردگیری مطالعات در حوزه ساختار ویروس و تغییرات آن در طراحی استراتژیهای دارویی و درمانی بسیار مؤثر است. مطالعه حاضر نشان میدهد استفاده از استراتژیهای درمانی و دارویی با توجه به مرحله بیماری متفاوت است، بهگونهای که برخی از داروها در مراحل اولیه بیماری از ورود ویروس به سلولهای هدف ممانعت میکنند یا در ترکیب با گلیکوپروتئینهای سطحی ویروس، از اتصال آنتیژن ویروس با گیرندههای موجود در سلولهای میزبان جلوگیری مینمایند. در مراحل پیشرفت بیماری، داروهای ضدویروسی از جمله مهارکنندههای پروتئازی و آنالوگهای نوکلئوزیدی باعث اختلال در تکثیر و تشکیل ساختمان ویروس میشوند. به دلیل تغییرات متناوب ویروس و ایجاد ویروسهای مقاوم به نمایش دارویی، بررسی مداوم مطالعات ویروسشناسی و بالینی و عملکرد داروهای موجود علیه ویروس حائز اهمیت است.
ملاحظات اخلاقی
پیروی از اصول اخلاق پژوهش
در این مطالعه، تمامی اصول اخلاق در پژوهش رعایت شده است.
حامی مالی
این مقاله یک مقاله مروری است و هیچگونه حمایت مالی از هیچ نهادی دریافت نکرده است.
مشارکت نویسندگان
مفهومسازی، تحلیل دادهها و نگارش متن و بازبینی:علی هژبر راجعونی، پروانه مهربد؛ انجام مطالعه:علی هژبر راجعونی.
تعارض منافع
طبق نظر نویسندگان هیچگونه تضاد منافعی در پژوهش حاضر وجود ندارد.
داروی نلفیناویر یک داروی غیرپپتیدی ضدویروسی است. این دارو به واسطه ایجاد پیوندهای هیدروژنی زیاد سبب مهار فعالیت پروتئازی ویروس میشود. این دارو نسبت به لوپیناویر اثر بیشتری در مهار ویروس دارد. این دارو از طریق پیوند هیدروژنی با اسیدهای آمینه Glu166 ،ASN142 (گروه آمیدی) و Thr26 (گروه هیدروکسیل) و از طریق پیوند هیدروژنی N-H…N با ایمیدازول اسید آمینه His41 سبب مهار این پروتئاز میشود. در مطالعات بالینی استفاده از این دارو به همراه سفارانتین در مهار ویروس و بیماری مؤثر بوده است (تصویر شماره 12 ج). این دارو در مهار سایتوکاینهای التهابی حاصل از بیماری نقش مؤثری دارد [44، 45].
پروتئازهای ویروس دارای گروههای تیول هستند. یکی از استراتژیهای دارویی در مهار ویروس، TOS II است. دارو با اتصال غیرکوالانسی فعالیت گروههای تیولی پروتئینهای سیتوزولی را مهار میکند. با اکسیداسیون تیول / تیولات به دیسولفید فعالیت پروتئازی ویروس مهار میشود. از مهارکنندههای آلفا کتوآمید ترکیب دارویی با نام 13b شناسایی شده است که با اتصال به محل سوبسترا در سطح پروتومرهای بین دُمینهای I و II و تهاجم نوکلئوفیلیک به Cys145 با گروه آلفا کتو (تشکیل ساختار همیکتال) و His41 با گروه اکسی آنیون (هیدروکسیل) سبب مهار پروتئاز ویروس و شکلگیری ویریون عفونی ویروس میشود (تصویر شماره 12 ب). این ترکیب با مرکز کاتالیتیک پروتئاز ویروس، دو پیوند هیدروژنی برقرار میکند. این ترکیب به دلیل داشتن گروه ترت بوتیل اکسیکربونیل (Boc) از غشای سلول عبور میکند و به خوبی در بافتهای بدن از جمله ریه منتشر میشود. این ترکیب علاوه بر استفاده خوراکی، بهصورت استنشاقی نیز قابل مصرف است [43].
پروتئینهای سنتزی ACE2
گیرنده ACE2 از پروتئینهای مهم در ورود ویروس به سلول میزبان است. پروتئینهای سنتزی ACE2 به صورت محلول (sACE2) هستند. در این پروتئینها بخش بین غشایی پروتئین وجود ندارد. این پروتئین قابلیت اتصال با FC ایمونوگلوبولین انسانی را دارد که میتواند اویدیتی مناسبی در زمان فراخوانی سلولهای فعال ایمنی فراهم کند و پایداری ایمونوگلوبولین را در سرم افزایش میدهد. از پروتئینهای نوترکیب، پروتئین sACE2.v2.4 بر اساس جهشهای جایگزینی T27Y ،L79T و N330Y در ساختار پروتئین ساخته شده است که به دلیل شباهت زیاد به پروتئین اصلی، ایمنیزایی کمتری ایجاد میکند و مزیت آن نسبت به آنتیبادیهای مونوکلونال، عدم ایجاد تشدید عفونت به واسطه آنتیبادی (ADE) است [46، 47].
مهارکنندههای فاکتورهای میزبانی
شناسایی عوامل میزبان در تکثیر ویروس و تشدید عفونت در کنترل عفونتهای ویروسی از اهمیت زیادی برخوردار است. با تجزیه و تحلیل دادههای حاصل از آزمایشات AP-MS و DIA-MS به ترتیب شبکه برهمکنش پروتئینهای میزبان (PPI) با پروتئینهای ویروس و نیز فسفریلاسیون پروتئینهای ویروس توسط میزبان مشخص شد. در دادههای حاصل از PPI، 332 پروتئین مؤثر میزبانی شناسایی شدند. در این مطالعه دو کلاس از داروهای مولکولی شناسایی شد که با مهار عوامل میزبانی سبب اختلال در روند تکثیر ویروس در سلول میشوند. مهارکنندههای بیوژنز پروتئینی (zotatifin ،ternatin-4 و PS3061) با مهار فاکتور elF4H سبب اختلال در ترجمه mRNA ویروسی میشوند و درنتیجه سطح مناسبی از پروتئینهای ویروسی ساخته نمیشود. از دیگر داروهای مولکولی، لیگاندهای گیرنده Sigma-1 و Sigma-2 هستند که با جلوگیری از میانکنش این گیرندهها با پروتئینهای ویروسی (ORF9c و NSP6) مانع تکثیر ویروس میشوند. این لیگاندها شامل haloperidol ،PB28 و hydroxychloroquine هستند [48]. بررسی بیشتر دادههای PPI نشان داد پروتئین ویروسی NSP13 نقش مؤثری در تنظیم شرایط متابولیسمی مناسب برای تکثیر ویروس دارد. در مطالعه دیگری، نقش نمکهای بیسموت در مهار پروتئین NSP13 بررسی شد. این مطالعه نشان داد نمکهای بیسموت پتاسیم سیترات (BPC) و رانیتیدین بیسموت سیترات (RBC) که به عنوان داروهای بیماریهای گوارشی مصرف میشود نسبت به بیسموت سیترات (BC) در مهار فعالیت NTPase ،ATPase و مهار فعالیت باز کردن پیچش ژنومی (Unwinding) پروتئین NSP13 اثر بیشتری دارد [49]. آزمایش روی فسفریلاسیون پروتئینهای ویروسی، 49 جایگاه فسفریلاسیون روی هفت پروتئین ویروسی M ،N (بیشترین تعداد)، NSP14 ،NSP13 و NSP9 نشان داد. در این مطالعه، فعالیت کازئین کیناز 2 (CK2)، فعالسازی مسیر p38 MAPK و سرکوب کینازهای میتوزی با فسفریلاسیون پروتئینهای ویروس، در توقف چرخه سلولی نقش دارند. پروتئین N ویروس در تنظیم فعالیت CK2 و سازماندهی اسکلت سلولی و جوانهزنی ذرات ویروس نقش مؤثری دارد. در این مطالعه داروهای مولکولی Silmitasertib (مهار CK2، فاز 2 بالینی)، gilteritinib (مهار AXL) ARRY-797 (مهار p38، فاز 2 یا 3 بالینی) شناسایی شدند [50].
مهارکنندههای مولکولی نوکلئیک اسیدی
از ویژگیهای مهم ژنوم ویروس SARS-CoV-2 میانکنشهای RNA-RNA با فواصل بلند و کوتاه در ژنوم ویروس و میانکنش ژنوم RNA ویروسی با RNAهای کوچک هستهای (snRNA) و بلند سلول میزبان است که در تکثیر و رونویسی ناپیوسته ژنوم ویروس نقش دارند. میانکنش snRNAها بیشتر با ژنوم ویروس (gRNA) صورت میگیرد که از طریق محلهای اختصاصی اتصال (ssb) در U2 ،U1 و U4 به نواحی ORF1a و ORF1b ژنوم ویروس متصل میشوند. رونوشتهای ژنوم ویروس (sgmRNA) از نواحی N و 3’UTR نیز به میزان زیادی با محلهای اختصاصی اتصال U1 و U2 میانکنش دارند. از میانکنش ژنوم ویروس با RNAهای بلند سلول میزبان، RNAase MRP از سلول میزبان شناسایی شد که در تجزیه RNA ویروسی نقش دارد و جهش در آن سبب بیماریهای انسانی از جمله بیماری هیپوپلازی مو غضروف میشود. از میانکنشهای RNA-RNA ژنوم ویروس و رونوشت آن در ناحیه 5’UTR پنج ناحیه SL1-SL5 و در 3’UTR سه ناحیه BSL ،hairpin-type pseudoknot و مارپیچ سهگانه اتصال شناسایی شد که در تکثیر و رونویسی ناپیوسته نقش دارند. با میانکنش RNA-RNA بین ناحیه مارپیچ سهگانه اتصال در 3’-UTR و ناحیه SL-3 در 5’-UTR، ژنوم ویروس، حلقوی میشود که در تنظیم رونوشتبرداری به صورت ناپیوسته نقش دارد [51]. ناحیه SL1 5’UTR به همراه NSP1، در ترجمه پروتئینهای ویروسی نقش اساسی دارد و در درمان با الیگونوکلئوتیدهای آنتیسنس (ASO) به عنوان پاشنه آشیل مهار ترجمه پروتئینهای ویروسی در نظر گرفته میشود [52]. آنالیزهای دارویی نشان دادند گلیسیرریزین، لوباریک اسید، گارسینولیک اسید و تریلازاد با اتصال به پروتئین NSP1 سبب مهار تشکیل کمپلکس NSP1/SL1 میشوند [53]. ناحیه s2m 3’UTR در ژنوم ویروس یک ناحیه حفاظتشده است که میانکنش آن با ORF1a شناسایی شده است. استفاده از الیگونوکلئوتیدهای LAN-Gapmer نشان داد اتصال مولکولهای Gapmer به ناحیه s2m سبب تجزیه مولکولهای mRNA ویروس توسط RNase H سلول میزبان میشود [54]. کاربرد درمانی این مولکولها (LAN-Gapmer) در کنار پروتئینهای نوترکیب hACE2 به صورت استنشاقی نیز مطرح شده است [55]. مطالعات روی مدلهای حیوانی نشان دادند از عوامل تداخل RNA (RNAi) ژنهای مختلف ویروس، مولکولهای siRNA طراحیشده بر اساس ژن (siRNA-N14 R7) RdRp در مهار تکثیر ویروس مؤثر است [56]. در کنار پایداری پایین مولکولهای siRNA، از محدودیت این مولکولها، سرکوب توسط پروتئین نوکلئوکپسید ویروس است که به عنوان سرکوبکننده ویروسی عوامل تداخل RNA (VSR) شناسایی شده است [57]. از مولکولهای RNA کاتالیتیک (ریبوزیم)، ریبوزیم نوترکیب DNA-RNA Hammerhead در بیماری سارس شناسایی شده است که به طور اختصاصی با اتصال به ناحیه ORF1ab(15460 GUC) سبب تجزیه ژنوم ویروس بیماری سارس میشود. این مولکولها به طور اختصاصی به مولکول RNA هدف متصل و سبب تجزیه آن میشوند، اما در ASO و RNAi تجزیه RNA هدف توسط RNase میزبان صورت میگیرد [57].
نتیجهگیری
مطالعه حاضر نشان میدهد جهت کنترل این ویروس، بررسی ساختار ویروس و بیولوژی آن در بدن از اهمیت زیادی برخوردار است. شناسایی داروهای مؤثر بر ویروس با توجه به ساختمان بیولوژیک ویروس ضروری است. با توجه به تغییرات ساختاری ویروس و جهشهای پیدرپی در ژنوم ویروس و به وجود آمدن سویههای مقاوم یا سویههای با قابلیت واگیری زیاد، ردگیری مطالعات در حوزه ساختار ویروس و تغییرات آن در طراحی استراتژیهای دارویی و درمانی بسیار مؤثر است. مطالعه حاضر نشان میدهد استفاده از استراتژیهای درمانی و دارویی با توجه به مرحله بیماری متفاوت است، بهگونهای که برخی از داروها در مراحل اولیه بیماری از ورود ویروس به سلولهای هدف ممانعت میکنند یا در ترکیب با گلیکوپروتئینهای سطحی ویروس، از اتصال آنتیژن ویروس با گیرندههای موجود در سلولهای میزبان جلوگیری مینمایند. در مراحل پیشرفت بیماری، داروهای ضدویروسی از جمله مهارکنندههای پروتئازی و آنالوگهای نوکلئوزیدی باعث اختلال در تکثیر و تشکیل ساختمان ویروس میشوند. به دلیل تغییرات متناوب ویروس و ایجاد ویروسهای مقاوم به نمایش دارویی، بررسی مداوم مطالعات ویروسشناسی و بالینی و عملکرد داروهای موجود علیه ویروس حائز اهمیت است.
ملاحظات اخلاقی
پیروی از اصول اخلاق پژوهش
در این مطالعه، تمامی اصول اخلاق در پژوهش رعایت شده است.
حامی مالی
این مقاله یک مقاله مروری است و هیچگونه حمایت مالی از هیچ نهادی دریافت نکرده است.
مشارکت نویسندگان
مفهومسازی، تحلیل دادهها و نگارش متن و بازبینی:علی هژبر راجعونی، پروانه مهربد؛ انجام مطالعه:علی هژبر راجعونی.
تعارض منافع
طبق نظر نویسندگان هیچگونه تضاد منافعی در پژوهش حاضر وجود ندارد.
References
- Siddell SG, Walker PJ, Lefkowitz EJ, Mushegian AR, Adams MJ, Dutilh BE, et al. Additional changes to taxonomy ratified in a special vote by the international committee on taxonomy of viruses (october 2018). Arch Virol. 2019; 164(3):943-6. [DOI:10.1007/s00705-018-04136-2]
- Gorbalenya AE, Baker SC, Baric RS, de Groot RJ, Drosten C, Gulyaeva AA, et al. The species severe acute respiratory syndrome-related coronavirus:Classifying 2019-nCoV and naming it SARS-CoV-2. Nat Microbiol. 2020; 5(4):536-44. [DOI:10.1038/s41564-020-0695-z] [PMCID]
- Zhou P, Yang X-L, Wang X-G, Hu B, Zhang L, Zhang W, et al. A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature. 2020; 579(7798):270-3. [DOI:10.1038/s41586-020-2012-7] [PMCID]
- Wrapp D, Wang N, Corbett KS, Goldsmith JA, Hsieh C-L, Abiona O, et al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. Science. 2020; 367(6483):1260-3. [DOI:10.1126/science.abb2507] [PMCID]
- Coutard B, Valle C, de Lamballerie X, Canard B, Seidah N, Decroly E. The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade. Antiviral Res. 2020; 176:104742. [DOI:10.1016/j.antiviral.2020.104742] [PMCID]
- Zhang YZ, Holmes EC. A genomic perspective on the origin and emergence of sars-cov-2. Cell. 2020; 181(2):223-7. [DOI:10.1016/j.cell.2020.03.035]
- Forster P, Forster L, Renfrew C, Forster M. Phylogenetic network analysis of SARS-CoV-2 genomes. Proc Natl Acad Sci USA. 2020; 117(17):9241-3. [DOI:10.1073/pnas.2004999117]
- Hulo C, de Castro E, Masson P, Bougueleret L, Bairoch A, Xenarios I, et al. ViralZone:A knowledge resource to understand virus diversity. Nucleic Acids Res. 2011; 39:D576-82. [DOI:10.1093/nar/gkq901]
- Lokugamage KG, Narayanan K, Huang C, Makino S. Severe acute respiratory syndrome coronavirus protein nsp1 is a novel eukaryotic translation inhibitor that represses multiple steps of translation initiation. J Virol. 2012; 86(24):13598-608. [DOI:10.1128/JVI.01958-12] [PMCID]
- Cornillez-Ty CT, Liao L, Yates 3rd JR, Kuhn P, Buchmeier MJ. Severe acute respiratory syndrome coronavirus nonstructural protein 2 interacts with a host protein complex involved in mitochondrial biogenesis and intracellular signaling. J Virol. 2009; 83(19):10314-8. [DOI:10.1128/JVI.00842-09] [PMCID]
- Lindner HA, Lytvyn V, Qi H, Lachance P, Ziomek E, Menard R. Selectivity in ISG15 and ubiquitin recognition by the SARS coronavirus papain-like protease. Arch Biochem Biophys. 2007; 466(1):8-14. [DOI:10.1016/j.abb.2007.07.006]
- Frieman M, Ratia K, Johnston RE, Mesecar AD, Baric RS. Severe acute respiratory syndrome coronavirus papain-like protease ubiquitin-like domain and catalytic domain regulate antagonism of IRF3 and NF-kappaB signaling. J Virol. 2009; 83(13):6689-705. [DOI:10.1128/JVI.02220-08] [PMCID]
- Saikatendu KS, Joseph JS, Subramanian V, Clayton T, Griffith M, Moy K, et al. Structural basis of severe acute respiratory syndrome coronavirus ADP-ribose-1’’-phosphate dephosphorylation by a conserved domain of nsP3. Structure. 2005; 13(11):1665-75. [DOI:10.1016/j.str.2005.07.022]
- Hognon C, Miclot T, Iriepa CG, France-Monerris A, Grandemange S, Terenzi A, et al. Role of RNA Guanine quadruplexes in favoring the dimerization of SARS unique domain in coronaviruses. J Phys Chem Lett. 2020; 11(14):5661-7. [DOI:10.1021/acs.jpclett.0c01097]
- Wang M, Cao R, Zhang L, Yang X, Liu J, Xu M, et al. Remdesivir and chloroquine effectively inhibit the recently emerged novel coronavirus (2019-nCoV) in vitro. Cell Res. 2020; 30(3):269-71. [DOI:10.1038/s41422-020-0282-0] [PMCID]
- Cottam EM, Whelband MC, Wileman T. Coronavirus NSP6 restricts autophagosome expansion. Autophagy. 2014; 10(8):1426-41. [DOI:10.4161/auto.29309]
- te Velthuis AJW, van den Worm SHE, Snijder EJ. The SARS-coronavirus nsp7+nsp8 complex is a unique multimeric RNA polymerase capable of both de novo initiation and primer extension. Nucleic Acids Res. 2012; 40(4):1737-47. [DOI:10.1093/nar/gkr893]
- Miknis ZJ, Donaldson EF, Umland TC, Rimmer RA, Baric RS, Schultz LW. Severe acute respiratory syndrome coronavirus nsp9 dimerization is essential for efficient viral growth. J Virol. 2009; 83(7):3007-18. [DOI:10.1128/JVI.01505-08] [PMCID]
- Bouvet M, Imbert I, Subissi L, Gluais L, Canard B, Decroly E. RNA 3’-end mismatch excision by the severe acute respiratory syndrome coronavirus nonstructural protein nsp10/nsp14 exoribonuclease complex. Proc Natl Acad Sci USA. 2012; 109(24):9372-7. [DOI:10.1073/pnas.1201130109]
- Ahn D-G, Choi J-K, Taylor DR, Oh J-W. Biochemical characterization of a recombinant SARS coronavirus nsp12 RNA-dependent RNA polymerase capable of copying viral RNA templates. Arch Virol. 2012; 157(11):2095-104. [DOI:10.1007/s00705-012-1404-x]
- Adedeji AO, Marchand B, Te Velthuis AJW, Snijder EJ, Weiss S, Eoff RL, et al. Mechanism of nucleic acid unwinding by SARS-CoV helicase. PloS One. 2012; 7(5):e36521. [DOI:10.1371/journal.pone.0036521] [PMCID]
- Tanner JA, Watt RM, Chai Y-B, Lu L-Y, Lin MC, Peiris JS, et al. The severe acute respiratory syndrome (SARS) coronavirus NTPase/helicase belongs to a distinct class of 5’ to 3’ viral helicases. J Biol Chem. 2003; 278(41):39578-82. [DOI:10.1074/jbc.C300328200]
- Zou X, Chen K, Zou J, Han P, Hao J, Han Z. Single-cell RNA-seq data analysis on the receptor ACE2 expression reveals the potential risk of different human organs vulnerable to 2019-nCoV infection. Front Med. 2020; 14(2):185-92. [DOI:10.1007/s11684-020-0754-0]
- Hoffmann M, Kleine-Weber H, Schroeder S, Krüger N, Herrler T, Erichsen S, et al. SARS-CoV-2 cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically proven protease inhibitor. Cell. 2020; 181(2):271-80.e8. [DOI:10.1016/j.cell.2020.02.052] [PMCID]
- Walls AC, Park Y-J, Tortorici MA, Wall A, McGuire AT, Veesler D. Structure, function, and antigenicity of the SARS-CoV-2 spike glycoprotein. Cell. 2020; 181(2):281-92.e6. [DOI:10.1016/j.cell.2020.02.058]
- Zou J, Yin J, Fang L, Yang M, Wang T, Wu W, et al. Computational prediction of mutational effects on the SARS-CoV-2 binding by relative free energy calculations. J Chem Inf Model. 2020; 60(12):5794-802. [DOI:10.1021/acs.jcim.0c00679]
- Minakshi R, Padhan K, Rani M, Khan N, Ahmad F, Jameel S. The SARS Coronavirus 3a protein causes endoplasmic reticulum stress and induces ligand-independent downregulation of the type 1 interferon receptor. PloS One. 2009; 4(12):e8342. [DOI:10.1371/journal.pone.0008342] [PMCID]
- Tan YJ, Tham PY, Chan DZL, Chou CF, Shen S, Fielding BC, et al. The severe acute respiratory syndrome coronavirus 3a protein up-regulates expression of fibrinogen in lung epithelial cells. J Virol. 2005; 79(15):10083-7. [DOI:10.1128/JVI.79.15.10083-10087.2005]
- Lu W, Zheng BJ, Xu K, Schwarz W, Du L, Wong CKL, et al. Severe acute respiratory syndrome-associated coronavirus 3a protein forms an ion channel and modulates virus release. Proc Natl Acad Sci USA. 2006; 103(33):12540-5. [DOI:10.1073/pnas.0605402103] [PMCID]
- Law PTW, Wong CH, Au TCC, Chuck CP, Kong SK, Chan PKS, et al. The 3a protein of severe acute respiratory syndrome-associated coronavirus induces apoptosis in Vero E6 cells. J Gen Virol. 2005; 86(Pt 7):1921-30. [DOI:10.1099/vir.0.80813-0]
- Bianchi M, Benvenuto D, Giovanetti M, Angeletti S, Ciccozzi M, Pascarella S. SARS-CoV-2 envelope and membrane proteins:Differences from closely related proteins linked to cross-species transmission?. Biomed Res Int. 2020; 2020:4389089. [DOI:10.1155/2020/4389089]
- Cao W, Liu X, Bai T, Fan H, Hong K, Song H, et al. High-dose intravenous immunoglobulin as a therapeutic option for deteriorating patients with coronavirus disease 2019. Open Forum Infect Dis. 2020; 7(3):ofaa102. [DOI:10.1093/ofid/ofaa102]
- Vaarala MH, Porvari KS, Kellokumpu S, Kyllönen AP, Vihko PT. Expression of transmembrane serine protease TMPRSS2 in mouse and human tissues. J Pathol. 2001; 193(1):134-40. [DOI:10.1002/1096-9896(2000)9999:9999<::AID-PATH743>3.0.CO;2-T]
- Zhang BN, Wang Q, Liu T, Dou SQ, Qi X, Jiang H, et al. Expression analysis of 2019-nCoV related ACE2 and TMPRSS2 in eye tissues. Zhonghua Yan Ke Za Zhi. 2020; 56(6):438-46. [DOI:10.3760/cma.j.cn112142-20200310-00170]
- Vincent MJ, Bergeron E, Benjannet S, Erickson BR, Rollin PE, Ksiazek TG, et al. Chloroquine is a potent inhibitor of SARS coronavirus infection and spread. Virol J. 2005; 2:69. [DOI:10.1186/1743-422X-2-69] [PMCID]
- Hirano T, Murakami M. COVID-19:A new virus, but a familiar receptor and cytokine release syndrome. Immunity. 2020; 52(5):731-3. [DOI:10.1016/j.immuni.2020.04.003]
- Shanker AK, Bhanu D, Alluri A, Gupta S. Whole genome sequence analysis and homology modelling of a 3C Like Peptidase and 1 a non-structural protein 3 of the SARS-CoV-2 shows protein ligand interaction with an Aza-Peptide and a noncovalent lead inhibitor with possible antiviral properties. New J Chem. 2020; 44(22):9202-12. [DOI:10.1039/D0NJ00974A]
- Vankadari N. Arbidol:A potential antiviral drug for the treatment of SARS-CoV-2 by blocking trimerization of the spike glycoprotein. Int J Antimicrob Agents. 2020; 56(2):105998. [DOI:10.1016/j.ijantimicag.2020.105998]
- Yang C, Ke C, Yue D, Li W, Hu Z, Liu W, et al. Effectiveness of arbidol for COVID-19 prevention in health professionals. Front Pub Health. 2020; 8:249. [DOI:10.3389/fpubh.2020.00249] [PMCID]
- Xia S, Liu M, Wang C, Xu W, Lan Q, Feng S, et al. Inhibition of SARS-CoV-2 (previously 2019-nCoV) infection by a highly potent pan-coronavirus fusion inhibitor targeting its spike protein that harbors a high capacity to mediate membrane fusion. Cell Res. 2020; 30(4):343-55. [DOI:10.1038/s41422-020-0305-x] [PMCID]
- Sheahan TP, Sims AC, Zhou S, Graham RL, Pruijssers AJ, Agostini ML, et al. An orally bioavailable broad-spectrum antiviral inhibits SARS-CoV-2 in human airway epithelial cell cultures and multiple coronaviruses in mice. Sci Transl Med. 2020; 12(541):eabb5883. [DOI:10.1126/scitranslmed.abb5883]
- Pillaiyar T, Manickam M, Namasivayam V, Hayashi Y, Jung S-H. An overview of severe acute respiratory syndrome–Ccoronavirus (SARS-CoV) 3CL protease inhibitors:Peptidomimetics and small molecule chemotherapy. J Med Chem. 2016; 59(14):6595-628. [DOI:10.1021/acs.jmedchem.5b01461]
- Ohashi H, Watashi K, Saso W, Shionoya K, Iwanami S, Hirokawa T, et al. Multidrug treatment with nelfinavir and cepharanthine against COVID-19. BioRxiv. Preprint. 2020. [DOI:10.1101/2020.04.14.039925]
- Xu Z, Yao H, Shen J, Wu N, Xu Y, Lu X, et al. Nelfinavir is active against SARS-CoV-2 in Vero E6 cells. ChemRxiv. Preprint. 2020. [DOI: 10.26434/chemrxiv.12039888.v1]
- Yang H, Yang M, Ding Y, Liu Y, Lou Z, Zhou Z, et al. The crystal structures of severe acute respiratory syndrome virus main protease and its complex with an inhibitor. Proc Nat Acad Sci USA. 2003; 100(23):13190-5. [DOI:10.1073/pnas.1835675100]
- Procko E. The sequence of human ACE2 is suboptimal for binding the S spike protein of SARS coronavirus 2. BioRxiv. 2020; 2020.03.16.994236. [DOI:10.1101/2020.03.16.994236]
- Gordon DE, Jang GM, Bouhaddou M, Xu J, Obernier K, O’Meara MJ, et al. A SARS-CoV-2-human protein-protein interaction map reveals targets for drug repurposing. BioRxiv. 2020; 2020.03.22.002386. [DOI:10.1101/2020.03.22.002386]
- Shu T, Huang M, Wu D, Ren Y, Zhang X, Han Y, et al. SARS-coronavirus-2 Nsp13 possesses NTPase and RNA helicase activities that can be inhibited by bismuth salts. Virol Sin. 2020; 35(3):321-9. [DOI:10.1007/s12250-020-00242-1] [PMCID]
- Bouhaddou M, Memon D, Meyer B, White KM, Rezelj VV, Correa Marrero M, et al. The global phosphorylation landscape of SARS-CoV-2 infection. Cell. 2020; 182(3):685-712.e19. [DOI:10.1016/j.cell.2020.06.034]
- Ziv O, Price J, Shalamova L, Kamenova T, Goodfellow I, Weber F, et al. The short- and long-range RNA-RNA Interactome of SARS-CoV-2. Mol Cell. 2020; 80(6):1067-77.e5. [DOI:10.1016/j.molcel.2020.11.004]
- Tidu A, Janvier A, Schaeffer L, Sosnowski P, Kuhn L, Hammann P, et al. The viral protein NSP1 acts as a ribosome gatekeeper for shutting down host translation and fostering SARS-CoV-2 translation. RNA. 2020 rna.078121.120. [DOI:10.1261/rna.078121.120]
- Vankadari N, Jeyasankar NN, Lopes WJ. Structure of the SARS-CoV-2 Nsp1/5′-Untranslated region complex and implications for potential therapeutic targets, a vaccine, and virulence. J Physic Chem Lett. 2020; 11(22):9659-68. [DOI:10.1021/acs.jpclett.0c02818] [PMCID]
- Lulla V, Wandel MP, Bandyra KJ, Dendooven T, Yang X, Doyle N, et al. Antisense oligonucleotides target a nearly invariant structural element from the SARS-CoV-2 genome and drive RNA degradation. BioRxiv. Preprint. 2020. [DOI:10.101/2020.09.18.304139]
- Verma NK, Fazil MHUT, Duggan SP, Kelleher D. Combination therapy using inhalable gapmeR and recombinant ACE2 for COVID-19. Front Mol Biosci. 2020; 7:197. [DOI:10.3389/fmolb.2020.00197]
- Gu SH, Yu CH, Song Y, Kim NY, Sim E, Choi JY, et al. A Small interfering RNA lead targeting RNA-dependent RNA-polymerase effectively inhibit the SARS-CoV-2 infection in Golden Syrian hamster and Rhesus macaque. BioRxiv. Preprint. 2020. [DOI:10.101/2020.07.07.190967]
- Mu J, Xu J, Zhang L, Shu T, Wu D, Huang M, et al. SARS-CoV-2-encoded nucleocapsid protein acts as a viral suppressor of RNA interference in cells. Sci China Life Sci. 2020; 63(9):1-4. [DOI:10.1007/s11427-020-1692-1] [PMCID]
- Fukushima A, Fukuda N, Lai Y, Ueno T, Moriyama M, Taguchi F, et al. Development of a chimeric DNA-RNA hammerhead ribozyme targeting SARS virus. Intervirology. 2009; 52(2):92-9. [DOI:10.1159/000215946]
ارسال پیام به نویسنده مسئول
| بازنشر اطلاعات | |
 |
این مقاله تحت شرایط Creative Commons Attribution-NonCommercial 4.0 International License قابل بازنشر است. |









.jpg)
.jpg)
.jpg)
.jpg)
.jpg)
.jpg)





