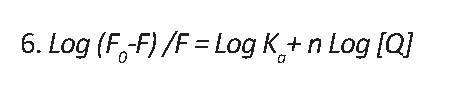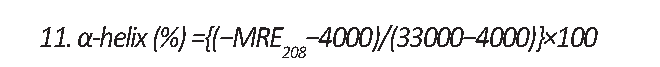دوره 22، شماره 6 - ( بهمن و اسفند 1398 )
جلد 22 شماره 6 صفحات 169-136 |
برگشت به فهرست نسخه ها
Download citation:
BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:



BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:
Hadi Chegni1 S, Taghizadeh M, Goliaei B. A Systematic Review of The Biophysical Aspect of The Effect of Pesticides on The Structural Changes in HSA Protein: The Analysis of Experimental and Computational Studies. J Arak Uni Med Sci 2020; 22 (6) :136-169
URL: http://jams.arakmu.ac.ir/article-1-6080-fa.html
URL: http://jams.arakmu.ac.ir/article-1-6080-fa.html
هادی چگنی شهرزاد، تقی زاده محمد، گلیایی بهرام. بررسی نظاممند مطالعات بیوفیزیکی اثر آفتکشها بر تغییرات ساختاری پروتئین HSA به روش تجربی و محاسباتی. مجله دانشگاه علوم پزشكي اراك. 1398; 22 (6) :136-169
1- گروه بیوفیزیک، دانشگاه آزاد اسلامی، واحد علوم و تحقیقات، تهران، ایران.
2- گروه بیوتکنولوژی، دانشکده علوم و فناوری پیشرفته، دانشکده علوم پزشکی، دانشگاه آزاد اسلامی، تهران، ایران
3- گروه بیوفیزیک، موسسه بیوشیمی و بیوفیزیک (IBB)، دانشگاه تهران، تهران. ،goliaei@ibb.ut.ac.ir
2- گروه بیوتکنولوژی، دانشکده علوم و فناوری پیشرفته، دانشکده علوم پزشکی، دانشگاه آزاد اسلامی، تهران، ایران
3- گروه بیوفیزیک، موسسه بیوشیمی و بیوفیزیک (IBB)، دانشگاه تهران، تهران. ،
واژههای کلیدی: آلبومین سرم انسانی(HSA)، آفتکشها، تعامل الکترواستاتیک، تعامل هیدروفوبیک، تبدیل فوریه ـ مادون قرمز، دورنگنمایی دورانی
متن کامل [PDF 9223 kb]
(2381 دریافت)
| چکیده (HTML) (6238 مشاهده)
A0 مقدار جذب پروتئین بدون حضور آفتکش، A جذب کمپلکسهای مختلف در حضور آفتکش است، و ضریب مولی پروتئین و آفتکش است مسیر نور کوت است. درنهایت تفاوت مقادیر جذب A و A0 یعنی A را محاسبه میکنیم. با رسم نمودار در نرمافزار اکسل، محور Y عکس تفاوت جذب و محور X عکس غلظت اولیه لیگاندها (Q) برحسب مولار است و با تقسیم عرض از مبدأ به شیب معادله نمودار مقدار ثابت تشکیل یا ثابت پیوندی (Ka) به دست میآید [50 ،49]. در تکنیک فلورسانس نیز برای به دست آوردن ثابت پیوندی از رابطه هیل (فرمول شماره 6) استفاده شده است:
F نشر پروتئین با حضور آفتکش، F0 نشر پروتئین بدون حضور آفتکش، [Q] غلظت آفتکش و Ka ثابت تجمع یا اتصال پروتئین ـ آفتکش است که با رسم نمودار Log (F0–F) /F علیه Log [Q] معادله نمودار به دست میآید؛ عرض از مبدأ آن، همان Log Ka و شیب آن، تعداد جایگاه اتصال (n) است [51].
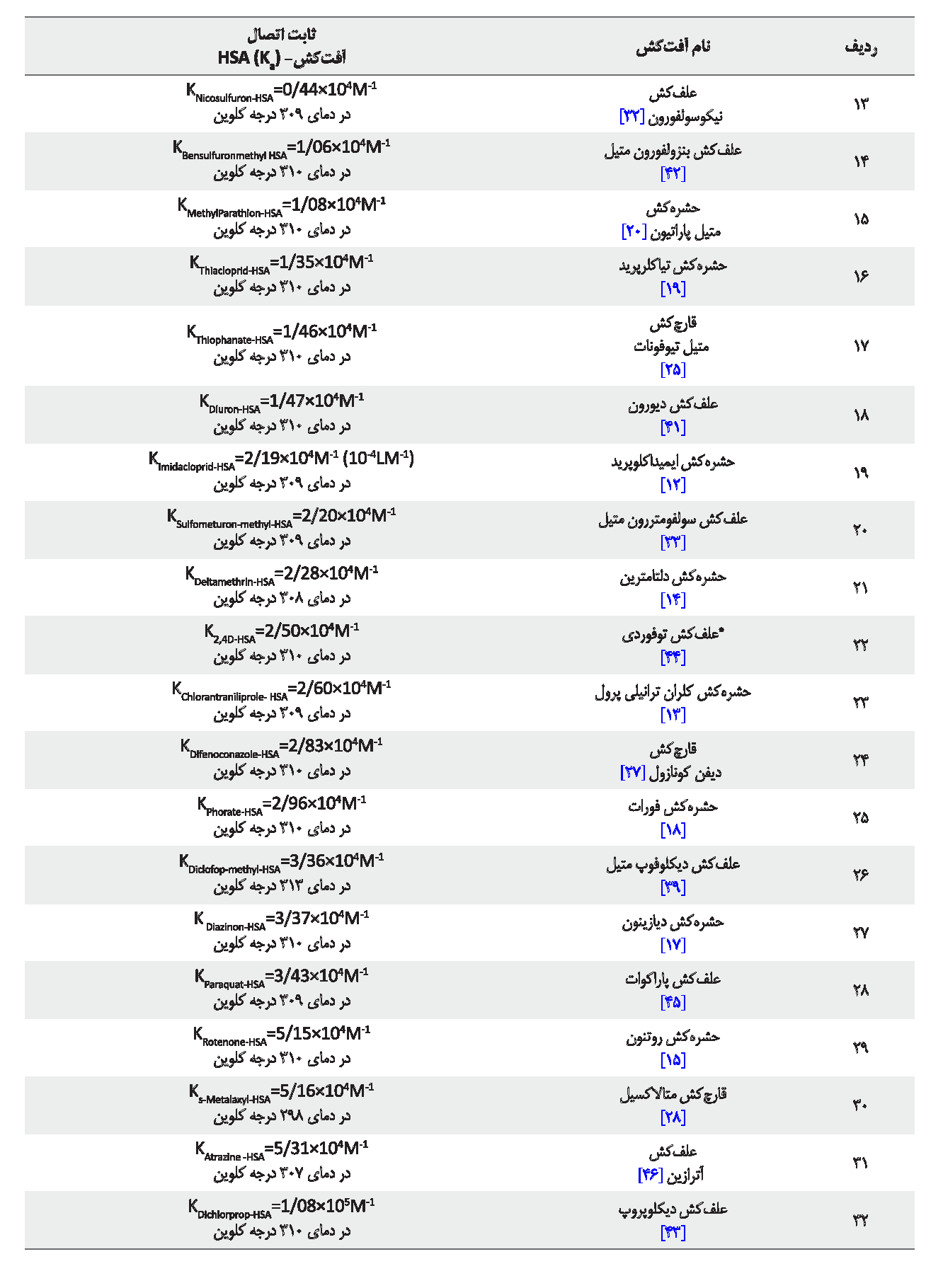
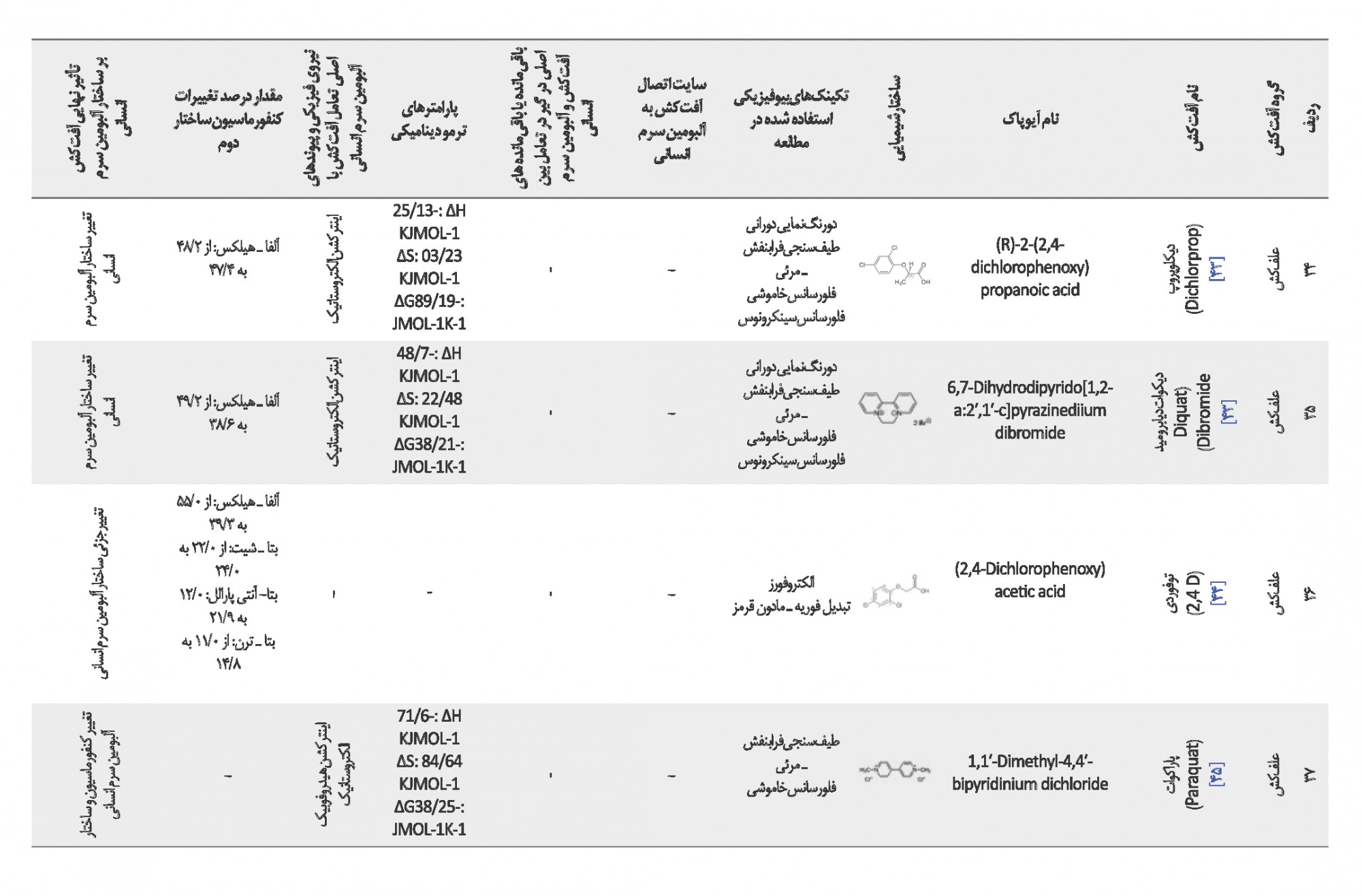
بر اساس دادههای محاسبات طیفسنجی فرابنفش ـ مرئی و فلورسانس به دو روش بالا، ثابتهای اتصال آفتکشها به آلبومین سرم انسانی طبق جدول شماره 1 به دست آمدهاند. اکثر ثابتهای اتصال گزارش شده در جدول شماره 1 بر مبنای دمای فیزیولوژی بدن (37 درجه سانتیگراد "310 درجه کلوین") درج شده است و آفتکشهایی که در این دما گزارشی نداشتهاند دامنه دمایی ترجیحاً نزدیک به آن انتخاب شده است. این موارد شامل آفتکشهای آترازین در دمای 34 درجه سانتیگراد (307 درجه کلوین)؛ پندی متالین و دلتامترین در دمای 35 درجه سانتیگراد (308 درجه کلوین)؛ ایمیداکلوپرید، پاراکوات، کلران ترانیلی پرول، نیکوسولفورون، سولفومترون متیل، متسولفورون متیل و پریزوسولفورون اتیل در دمای 36 درجه سانتیگراد (309 درجه کلوین)؛ کاربندازیم، دیکلوفوپ متیل و دیکلوفوپ در دمای 40 درجه سانتیگراد (313 درجه کلوین) هستند. متالاکسیل، ادیفنفوس و تیوکونازول هم تنها در دمای 25 درجه سانتیگراد (298 درجه کلوین) گزارش داشتهاند.
ثابتهای اتصال آفتکش ـ HSA با توجه به دامنههای دمایی ذکر شده برای 39 آفتکش طبق جدول شماره 1 در دو محدوده 1-M103 تا 106 گزارش شدهاند. با یک بررسی کلی «مقدار عددی ثابت اتصال» آفتکشهایی که در دمای 37 درجه سانتیگراد گزارش داشتهاند، به ترتیب شامل آفتکشهای گلیفوزات، تریادیمفون، کربوفوران، دیکوات دیابرومید، ایمازالیل، پنکونازول، میکلوبوتانیل، پرمترین، پروپیکونازول، بنزولفورون متیل، متیل پاراتیون، تیاکلرپرید، متیل تیوفونات، دیورون، توفوردی، دیفن کونازول، فورات، دیازینون، روتنون، دیکلوپروپ، کلرپیریفوس و پنتا کلروفنل هستند که برای دستیابی آسانتر و مشاهده مقدار عددی ثابت اتصال آنها در جدول شماره 1 به صورت رنگی نمایش داده شدهاند. ( استخراج اطلاعات بر اساس منابع 12 تا 46 بوده است).
پیوندها و نیروهای فیزیکی تعامل آفتکش ـ HSA
تجزیه، تحلیل و قضاوت در مورد ماهیت و نوع نیروهای فیزیکی و پیوندهای درگیر در تعامل آفتکش با پروتئین HSA بر اساس پارامترهای مهم ترمودینامیکی شامل تغییرات آنتالپی(ΔH)، تغییرات آنتروپی(ΔS) و تغییرات انرژی آزاد گیبس(ΔG°)که از روش طیفسنجی فلورسانس محاسبه میشوند، انجام میگیرد. عامل برقراری تعامل بین پروتئین و لیگاندها اساساً میانکنشهای هیدروفوبیک، الکترواستاتیک، نیروی واندروالسی و حضور پیوندهای هیدروژنی است. بزرگی و علامت پارامترهای ترمودینامیکی، نوع اینترکشنها، نیروها و پیوندهای دخالتکننده در تعامل آفتکش ـ پروتئین را مشخص میکند [53 ،52]. چنانچه تغییرات آنتروپی و تغییرات آنتالپی محاسبهشده هر دو مثبت باشند، اینترکشن از نوع هیدروفوبیک و اگر هر دو منفی باشند اتصال به شکل پیوند هیدروژنی و نیروی واندروالسی است و چنانچه تغییرات آنتالپی تقریباً برابر صفر و تغییرات آنتروپی مثبت باشند، اینترکشن از نوع الکتروستاتیک است [55 ،54]. توابع ترمودینامیکی لیگاند اتصالشده به پروتئین به کمک فرمولهلی 7 تا 9 تعیین میشود:
پس از بررسی، نوع اینترکشن آفتکش ـ HSA در هر مقاله به کمک پارامترهای ترمودینامیکی در طیفسنجی فلورسانس نتایج بدین شرح استخراج شدند: برای آفتکشهای کلرپیریفوس، دیازینون، تریادیمفون، پنکونازول، ایمازالیل، میکلوبوتانیل، پروپیکونازول، دیفن کونازول و پاراکوات اینترکشن الکتروستاتیکی و هیدروفوبیکی، آفتکشهای گلیکوفوزات، پرمترین، پندی متالین، دیکلوفوب متیل، دیکلوفوب، متیل تیوفونات و نیکوسولفورون اینترکشن هیدروفوبیکی و پیوند هیدروژنی، آفتکشهای کلران ترانیلی پرول، دلتامترین، کربوفوران پریزوسولفورون اتیل نیروی واندروالسی و پیوند هیدروژنی، آفتکشهای دیکلوپروب، دیکوات دیابرمید و آترازین اینترکشن الکتروستاتیک، آفتکشهای؛ فوران، متیل پاراتیون، پنتاکلروفنل، ادیفنفوس، تیوکونازول اینترکشن هیدروفوبیکی، آفتکشهای متسولفورون متیل، دیورون، بنزولفورون متیل و تیاکلروپرید اینترکشن هیدروفوبیک، الکتروستاتیک و پیوند هیدروژنی، آفتکشهای کاربندازیم و امیداکلروپرید اینترکشن هیدروفوبیک، پیوند هیدروژنی و π-π، آفتکش متالاکسیل اینترکشن هیدروفوبیک و پیوند، آفتکش روتنون پیوند هیدروژنی و سولفومترون متیل پیوند هیدروژنی، اینترکشن واندروالسی و هیدروفوبیک به عنوان اینترکشنها و پیوندهای قالب در تعامل با پروتئین HSA گزارش شده است. همانطور که مشخص است به ترتیب اولویت تعامل هیدروفوبیک، پیوند هیدروژنی و تعامل الکتروستاتیک بیشترین سهم را در اینترکشن آفتکشها با پروتئین HSA داشتهاند (استخراج اطلاعات بر اساس منابع 12 تا 46 بوده است).
θobs؛ CD در millidegree، n تعداد باقیماندههای آمینواسید (585)، l طول سل (یک سانتیمتر)، Cp غلظت مولار HSA.
همچنین آلفا ـ هیلکس برای پروتئین آزاد و ترکیب با آفتکش، از ارزش MRE در 208 نانومتر با استفاده از فرمول شماره 11 محاسبه میشود:
از مجموع 35 مقاله، 26 مقاله ساختار دوم پروتئین را با طیفسنجی CD آنالیز کردند که آفتکشهای تیوکونازول، افزایش ساختار آلفا ـ هلیکس از 55 درصد به 60 درصد و پریزاسولفورون، از 37/45 درصد به 27/51 درصد داشتند. همچنین آترازین کاهش ساختار بتا ـ ترن از 2/28 درصد به 3/ 26 درصد و دیکلوفوپ متیل کاهش درصد بتا ـ شیت در ساختار پروتئین HSA داشتند، در بقیه 23 مورد دیگر از 26 مقاله که از روش طیفسنجی CD برای بررسی تغییرات ساختار پروتئین HSA استفاده کرده بودند کاهش در ساختار آلفا ـ هلیکس و افزایش در سایر ساختارها مانند بتا ـ شیت و بتا ـ ترن گزارش شده است ( استخراج اطلاعات بر اساس منابع 12 تا 46 بوده است).
سایتهای مورد علاقه آفتکشها در پروتئین HSA
برای تعیین جایگاه اتصال مورد هدف آفتکش در تعامل با پروتئین HSA، ریشههای آمینواسید درگیر در پیوند، نوع اینترکشن و نوع پیوند، از روش مدلسازی مولکولی رایانهای در مقالهها استفاده شده است. مدلسازی مولکولی بهترین جایگاه برای اتصال لیگاند به پروتئین HSA، با کمترین انرژی پیوند را تعیین و پیشگویی میکند. پس از دریافت ساختار کریستاله پروتئین سرم آلبومین و آفتکش از بانکهای اطلاعاتی چون Data Base PDB، برای محاسبات Docking میتوان از مجموعه نرمافزاری متداول AutoDock Tools و AutoDock vina، weblab viewer جهت نمایش نتایج و Pose صحیح لیگاند و رسپتور استفاده کرد [17]. با بررسی جایگاه اتصال آفتکشها روی پروتئین HSA در 35 مقاله مطالعهشده از بین حشرهکشها هشت مورد ایمیداکلوپرید، دلتامترین، روتنون، کلرپیریفوس، دیازینون، فورات، تیاکلرپرید و متیل پاراتیون جایگاه یا سایت یک را در زیردامنه IIA، کلران ترانیلی پرول زیردامنه IIIA در سایت دو و پنتا کلروفنل سایت دیگری بین دامنه (دُمین) یک و سه را انتخاب کردهاند. تاکنون هم جایگاه اتصال حشرهکش کربوفوران مورد مطالعه قرار نگرفته است. از بین 11 قارچکش، 10 مورد تریادیمفون، پنکونازول، ایمازالیل، میکلوبوتانیل، پروپیکونازول، متیل تیوفونات، دیفن کونازول، متالاکسیل، ادیفنفوس وکاربندازیم زیردامنه IIA در سایت یک را انتخاب کرده و برای تیوبوکونازول مطالعه بررسی جایگاه اتصال تاکنون صورت نگرفته است.
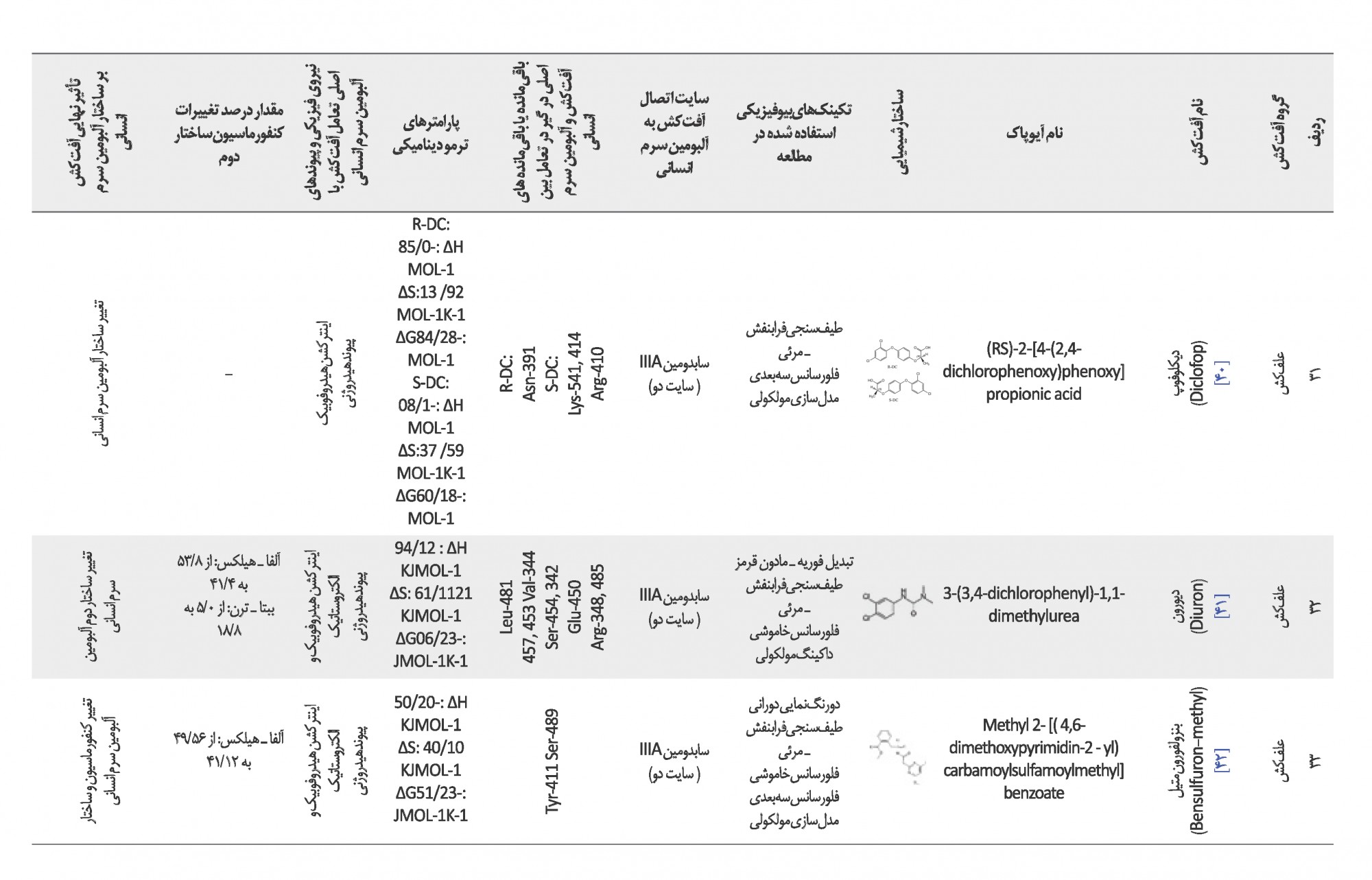
از 17 علفکش، هفت مورد نیکوسولفورون، سولفومتررون متیل، گلیفوزات، پرمترین، پندی متالین، متسولفورون متیل، پریزوسولفورون اتیل زیردامنه IIA در سایت یک و شش مورد دیکلوفوپ متیل، دیکلوفوپ، دیورون، پریزوسولفورون اتیل، آترازین و بنزولفورون متیل زیردامنه IIIA در سایت دو را برای اتصال به پروتئین HSA انتخاب کردهاند. برای چهار مورد دیکلوپروپ، دیکوات دیابرومید، توفوردی، پاراکوات نیز مطالعه جایگاه اتصال انجام نشده است و سم آمیترول نیز تعاملی با آلبومین سرم انسانی نداشته است. درمجموع از 39 آفتکش، جایگاه اتصال 32 مورد بررسی شده که 25 آفتکش به پروتئین HSA در زیردامنه IIA پیوند شدهاند (استخراج اطلاعات بر اساس منابع 12 تا 46 بوده است).
بحث
طبق تحقیقات مختلف، آفتکشها تأثیر عمدهای بر سلامت عمومی مردم بهویژه آنزیمها، کانالهای یونی و گیرندهها، پروتئینهای حامل از جمله پروتئینهای پلاسما مانند پروتئین HSA که در عملکردهای مهم بیولوژیکی زیستی مانند فرایند سمزدایی شرکت میکنند، دارند و آفتکشها در ایجاد بیماری مختلف از جمله پارکینسون میتوانند دخالت داشته باشند [63-65]. این مطالعه مروری نظاممند نیز بر اساس گزارشات 35 مقاله علمی ـ پژوهشی بیوفیزیکی بیان میدارد آفتکشها توانستهاند باعث تغییر نشر و جذب طبیعی پروتئین پس از اینترکشن با آن شوند و میزان تغییرات در نشر و جذب پروتئین HSA وابسته به غلظت آفتکشهاست. این تغییر در نشر و جذب نشان از تغییرات ساختاری پروتئین HSA ناشی از اثر آفتکشهاست که میتواند در اثر قرارگرفتن آمینواسیدهای تریپتوفان در محیط آبگریز یا آبدوست و یا تغییر موقعیت بار باشد که منجر به افزایش یا کاهش در جذب، خاموشکردن وکاهش نشر پروتئین، تغییر ساختار فضایی، ناپایداری پروتئین و درنهایت تغییردرصدی از ساختار سوم پروتئین HSA متناسب با نوع آفتکش میشوند [66]. از طرفی با بررسی مطالعات طیفسنجی تبدیل فوریه ـ مادون قرمز و دورنگنمایی دورانی مقالههای بررسیشده مشخص شد حضور اکثر آفتکشها باعث کاهش ساختار آلفا ـ هلیکس، افزایش ساختارهای بتا (ورقهای) و ساختارهای نامنظم پروتئین میشوند.
از آنجایی که ساختار دوم پروتئین HSA رابطه نزدیکی با فعالیتهای بیولوژیکی آن دارد [17]، ایجاد تغییرات در ساختار دوم پروتئین هرچند به صورت جزئی، توسط آفتکشها منجر به کاهش فعالیت بیولوژیکی پروتئین HSA میشود. حال باید دید آفتکشها با چه ثابت اتصالی این تغییرات را بر ساختار پروتئین اعمال کردهاند؟ همانطور که میدانیم به طور معمول مقدار عددی اتصال لیگاند به پروتئین بین دامنه 104 تا 106 معکوس مولار (M-1) است [52 ،8]. در تحقیقات دیگری ثابت اتصال ترکیبات خارجی در دمای 25 تا 37 درجه سانتیگراد بین دو دامنه -1M103 تا 105 بیان شده است [16]. با بررسی 35 مقاله درباره اثر آفتکشها بر پروتئین HSA نیز ثابت اتصال آفتکشها به پروتئین، در دو محدوده -1M103 تا 106 گزارش شدهاند که نشان از تعامل به نسبت خوب آفتکشها با پروتئین HSA دارد که برای اعمال اهداف خود و یا تعیین سرنوشت آنها توسط این پروتئین میتواند حائز اهمیت باشد.
به نظر میرسد اتصال آفتکشها به HSA در بیشتر موارد، از طریق اینترکشنهای هیدروفوبیکی و الکتروستاتیکی با حضور پیوندهای هیدروژنی است و اکثر این تعاملها در زیردامنه مهم IIA در سایت یک پروتئین HSA اتفاق میافتند. آفتکشها با دسترسی به Trp-214 پروتئین در زیردامنه IIA باعث تغییر قطبیت محیط اطراف آن و اعمال تغییرات ساختاری در پروتئین میشوند. هرچند زیردامنه IIA ظرفیت اتصال همزمان به چند لیگاند را دارد، اما میتوان نگران جنبه رقابتی آفتکشها نسبت به لیگاندهای مؤثر در بدن بود، مانند داروها و هورمونها که بیشتر آنها اتصالشان در زیردامنه IIA اتفاق میافتد. گزارشاتی مبنی بر اختلال در اتصال داروها و لیگاندهای ضروری به خاطر درگیرشدن پروتئین HSA در توزیع و حذف آفتکشها گزارش شده است که منجر به کاهش غلظت پروتئین آزاد شدهاند [5].
در کل همه آفتکشهای بررسیشده به جز آمیترول، به گونهای بر ساختار پروتئین HSA تغییرات خود را اعمال کردهاند و میزان و درصد این تغییرات بستگی مستقیم به غلظت آفتکش و زمان ماندگاری آنها در جریان خون دارد. این مطالعه فقط 39 مورد از آفتکشها از صدها آفتکش استفادهشده درکشاورزی را بررسی کرده است و اطلاعات کاملی از بین مقالههای موجود در نمایههای معتبر علمی از تأثیرات سایرآفتکشها بر تغییر ساختار پروتئین HSA به روش بیوفیزیکی هنوز گزارش نشده است. وجود مطالعههای بیشتر در مورد بررسی بیوفیزیکی تغییرات ساختار پروتئین HSA در حضور آفتکشها میتواند به ارزیابی جامعتری از تأثیر آفتکشها بر ساختار پروتئین HSA کمک کند. از طرفی این آزمایشات و تفسیر نتایج بر اساس مقالههای علمی ـ پژوهشی بوده که در محیط آزمایشگاهی خارج از بدن انسان انجام شدهاند و نیازمند بررسی و انجام آزمایشات تکمیلی در محیط زنده با رعایت اصول اخلاقی در پژوهشهای آتی است.
نتیجهگیری
به طور کلی این بررسی نشان داد که حضور آفتکشها در جریان خون بدن انسان میتوانند روی HSA (پروتئین مهم پلاسما) در کنار کاهش غلظت آزاد آن، تغییرات ساختاری ـ هرچند به صورت جزئی ـ متناسب با زمان حضور در بدن اعمال کنند و با تغییر کنفورماسیون ساختار دوم و سوم پروتئین HSAدر عملکرد بیولوژیکی آن اختلال ایجاد کنند. با توجه به اینکه HSA یک پروتئین بسیار مهم و پرکاربرد در عملکردهای زیستی بدن انسان به حساب میآید، خطرات آفتکشها و تأثیر آنها بر این پروتئین را نبایستی نادیده گرفت.
ملاحظات اخلاقی
پیروی از اصول اخلاق پژوهش
در این مطالعه، تمامی اصول اخلاق در پژوهش رعایت شده است.
حامی مالی
؟؟
مشارکت نویسندگان
تمامی نویسندگان در نگارش مقاله به یک اندازه مشارکت داشتهاند.
تعارض منافع
نویسندگان این مقاله تصریح میکنند که هیچگونه تضاد منافعی در خصوص پژوهش حاضر وجود ندارد.
References
García FP, Cortés Ascencio SY, Gaytan Oyarzun JC, Hernández AC, Alavarado PV. Pesticides: Classification, uses and toxicity. Measures of exposure and genotoxic risks. J Res Environ Sci Toxicol. 2012; 1(11):279-93.
Commission FWCA. Maximum residue limits for pesticides. FAO/WHO: Rome, Italy. 2001.
Křivánkova L, Boček P, Tekel J, Kovačičová J. Isotachophoretic determination of herbicides prometryne, desmetryne, terbutryne and hydroxy‐derivatives of atrazine and simazine in extracts of milk. Electrophoresis. 1989; 10(10):731-4. [DOI:10.1002/elps.1150101015] [PMID]
Aktar W, Sengupta D, Chowdhury A. Impact of pesticides use in agriculture: their benefits and hazards. Interdiscip Toxicol. 2009; 2(1):1-12. [DOI:10.2478/v10102-009-0001-7] [PMID] [PMCID]
Kragh-Hansen U. Structure and ligand binding properties of human serum albumin. Dan Med Bull. 1990; 37(1):57-84.
Cserháti T, Forgács E. Charge transfer chromatographic study of the binding of commercial pesticides to various albumins. J Chromatogr. 1995; 699(1-2):285-90. [DOI:10.1016/0021-9673(95)00144-C]
Buttar D, Colclough N, Gerhardt S, MacFaul PA, Phillips SD, Plowright A, et al. A combined spectroscopic and crystallographic approach to probing drug-human serum albumin interactions. Bioorg Med Chem. 2010; 18(21):7486-96. [DOI:10.1016/j.bmc.2010.08.052] [PMID]
Zsila F, Bikadi Z, Malik D, Hari P, Pechan I, Berces A, et al. Evaluation of drug-human serum albumin binding interactions with support vector machine aided online automated docking. Bioinformatics. 2011; 27(13):1806-13. [DOI:10.1093/bioinformatics/btr284] [PMID]
Bhattacharya AA, Curry S, Franks NP. Binding of the general anesthetics propofol and halothane to human serum albumin high resolution crystal structures. J Biol Chem. 2000; 275(49):38731-8. [DOI:10.1074/jbc.M005460200] [PMID]
Ghuman J, Zunszain PA, Petitpas I, Bhattacharya AA, Otagiri M, Curry S. Structural basis of the drug-binding specificity of human serum albumin. J Mol Biol. 2005; 353(1):38-52. [DOI:10.1016/j.jmb.2005.07.075] [PMID]
Sahoo BK, Ghosh KS, Dasgupta S. Molecular interactions of isoxazolcurcumin with human serum albumin: Spectroscopic and molecular modeling studies. Biopolymers. 2009; 91(2):108-19. [DOI:10.1002/bip.21092] [PMID]
Wang YQ, Tang BP, Zhang HM, Zhou QH, Zhang GC. Studies on the interaction between imidacloprid and human serum albumin: Spectroscopic approach. J Photochem Photobiol B Biol. 2009; 94(3):183-90. [DOI:10.1016/j.jphotobiol.2008.11.013] [PMID]
Ding F, Liu W, Diao JX, Yin B, Zhang L, Sun Y. Complexation of insecticide chlorantraniliprole with human serum albumin: Biophysical aspects. J Lumin. 2011; 131(7):1327-35. [DOI:10.1016/j.jlumin.2011.03.007]
Wang J, Ma L, Zhang Y, Jiang T. Investigation of the interaction of Deltamethrin (DM) with human serum albumin by multi-spectroscopic method. J Mol Struct. 2017; 1129:160-8. [DOI:10.1016/j.molstruc.2016.09.061]
Fan XY, Zhang Y, Wang J, Yang LY, Jiang FL, Liu Y. Exploring the interaction between rotenone and human serum albumin. J Chem Thermodyn. 2014; 69:186-92. [DOI:10.1016/j.jct.2013.10.021]
Han XL, Tian FF, Ge YS, Jiang FL, Lai L, Li DW, et al. Spectroscopic, structural and thermodynamic properties of chlorpyrifos bound to serum albumin: A comparative study between BSA and HSA. J Photochem Photobiol B biology. 2012; 109:1-11. [DOI:10.1016/j.jphotobiol.2011.12.010] [PMID]
Hadichegeni S, Goliaei B, Taghizadeh M, Davoodmanesh S, Taghavi F, Hashemi M. Characterization of the interaction between human serum albumin and diazinon via spectroscopic and molecular docking methods. Hum Exp Toxicol. 2018; 37(9):959-71. [DOI:10.1177/0960327117741752] [PMID]
Saquib Q, Al-Khedhairy AA, Siddiqui MA, Roy AS, Dasgupta S, Musarrat J. Preferential binding of insecticide phorate with sub-domain IIA of human serum albumin induces protein damage and its toxicological significance. Food Chem Toxicol. 2011; 49(8):1787-95. [DOI:10.1016/j.fct.2011.04.028] [PMID]
Wang C, Chu Q, Chen C, Bo Z. Investigation of the mechanism of binding of thiacloprid to human serum albumin using spectroscopic techniques and molecular modeling methods. J Spectrosc. 2011; 25(2):113-22. [DOI:10.1155/2011/195489]
Silva Dl, Cortez CM, Cunha-Bastos J, Louro SR. Methyl parathion interaction with human and bovine serum albumin. Toxicol Lett. 2004; 147(1):53-61. [DOI:10.1016/j.toxlet.2003.10.014] [PMID]
Wang YQ, Zhang HM, Zhou QH. Investigation of the interaction between pentachlorophenol and human serum albumin using spectral methods. J Mol Struct. 2009; 932(1-3):31-7. [DOI:10.1016/j.molstruc.2009.05.036]
Tunç S, Duman O, Soylu İ, Bozoğlan BK. Spectroscopic investigation of the interactions of carbofuran and amitrol herbicides with human serum albumin. J Lumin. 2014; 151:22-8. [DOI:10.1016/j.jlumin.2014.02.004]
Zhang J, Zhuang S, Tong C, Liu W. Probing the molecular interaction of triazole fungicides with human serum albumin by multispectroscopic techniques and molecular modeling. J Agric Food Chem. 2013; 61(30):7203-11. [DOI:10.1021/jf401095n] [PMID]
Wang C, Li Y. Study on the binding of propiconazole to protein by molecular modeling and a multispectroscopic method. J Agric Food Chem. 2011; 59(15):8507-12. [DOI:10.1021/jf200970s] [PMID]
Li J, Liu X, Ren C, Li J, Sheng F, Hu Z. In vitro study on the interaction between thiophanate methyl and human serum albumin. J Photochem Photobiol B Biol. 2009; 94(3):158-63. [DOI:10.1016/j.jphotobiol.2008.10.001] [PMID]
Saquib Q, Al-Khedhairy AA, Alarifi SA, Dwivedi S, Mustafa J, Musarrat J. Fungicide methyl thiophanate binding at sub-domain IIA of human serum albumin triggers conformational change and protein damage. Int J Biol Macromol. 2010; 47(1):60-7. [DOI:10.1016/j.ijbiomac.2010.03.020] [PMID]
Li Y, Ma X, Lu G. Systematic investigation of the toxic mechanism of difenoconazole on protein by spectroscopic and molecular modeling. Pestic Biochem Physiol. 2013; 105(3):155-60. [DOI:10.1016/j.pestbp.2012.12.010]
Ding F, Li XN, Diao JX, Sun Y, Zhang L, Sun Y. Chiral recognition of metalaxyl enantiomers by human serum albumin: Evidence from molecular modeling and photophysical approach. Chirality. 2012; 24(6):471-80. [DOI:10.1002/chir.22024] [PMID]
Ahmad A, Ahmad M. Understanding the fate of human serum albumin upon interaction with edifenphos: Biophysical and biochemical approaches. Pestic Biochem Physiol. 2018; 145:46-55. [DOI:10.1016/j.pestbp.2018.01.006] [PMID]
Siddiqui MF, Khan MS, Husain FM, Bano B. Deciphering the binding of carbendazim (fungicide) with human serum albumin: A multi-spectroscopic and molecular modelling studies. J Biomol Struct Dyn. 2019; 37(9):2230-41. [DOI:10.1080/07391102.2018.1481768] [PMID]
Staničová J, Želonková K, Verebová V, Holečková B, Dianovský J. Interaction of the fungicide tebuconazole with human serum albumin: A preliminary study. Folia Vet. 2018; 62(2):85-91. [DOI:10.2478/fv-2018-0020]
Ding F, Liu W, Li N, Zhang L, Sun Y. Complex of nicosulfuron with human serum albumin: A biophysical study. J Mol Struct. 2010; 975(1-3):256-64. [DOI:10.1016/j.molstruc.2010.04.033]
Ding F, Liu W, Zhang L, Yin B, Sun Y. Sulfometuron-methyl binding to human serum albumin: Evidence that sulfometuron-methyl binds at the Sudlow’s site I. J Mol Struct. 2010; 968(1-3):59-66. [DOI:10.1016/j.molstruc.2010.01.020]
Yue Y, Zhang Y, Zhou L, Qin J, Chen X. In vitro study on the binding of herbicide glyphosate to human serum albumin by optical spectroscopy and molecular modeling. J Photochem Photobiol B Biol. 2008; 90(1):26-32. [DOI:10.1016/j.jphotobiol.2007.10.003] [PMID]
Wang Y, Zhang G, Wang L. Interaction of prometryn to human serum albumin: Insights from spectroscopic and molecular docking studies. Pestic Biochem Physiol. 2014; 108:66-73. [DOI:10.1016/j.pestbp.2013.12.006] [PMID]
Lee WQ, Affandi ISM, Feroz SR, Mohamad SB, Tayyab S. Evaluation of pendimethalin binding to human serum albumin: Insights from spectroscopic and molecular modeling approach. J Biochem Mol Toxicol. 2017; 31(2):e21839. [DOI:10.1002/jbt.21839] [PMID]
Ding F, Liu W, Zhang X, Zhang L, Sun Y. Fluorescence and circular dichroism studies of conjugates between metsulfuron-methyl and human serum albumin. Colloids Surf B Biointerfaces. 2010; 76(2):441-8. [DOI:10.1016/j.colsurfb.2009.12.003] [PMID]
Ding F, Liu W, Zhang X, Wu LJ, Zhang L, Sun Y. Identification of pyrazosulfuron-ethyl binding affinity and binding site subdomain IIA in human serum albumin by spectroscopic methods. Spectrochim Acta A Mol Biomol Spectrosc. 2010; 75(3):1088-94. [DOI:10.1016/j.saa.2009.12.062] [PMID]
Zhang P, Liu D, Li Z, Shen Z, Wang P, Zhou M, et al. Multispectroscopic and molecular modeling approach to investigate the interaction of diclofop-methyl enantiomers with human serum albumin. J Lumin. 2014; 155:231-7. [DOI:10.1016/j.jlumin.2014.06.040]
Zhang P, Li Z, Wang X, Shen Z, Wang Y, Yan J, et al. Study of the enantioselective interaction of diclofop and human serum albumin by spectroscopic and molecular modeling approaches in vitro. Chirality. 2013; 25(11):719-25. [DOI:10.1002/chir.22204] [PMID]
Chen H, Rao H, Yang J, Qiao Y, Wang F, Yao J. Interaction of diuron to human serum albumin: Insights from spectroscopic and molecular docking studies. J Environ Sci Health B. 2016; 51(3):154-9. [DOI:10.1080/03601234.2015.1108800] [PMID]
Ding F, Liu W, Li Y, Zhang L, Sun Y. Determining the binding affinity and binding site of bensulfuron-methyl to human serum albumin by quenching of the intrinsic tryptophan fluorescence. J Lumin. 2010; 130(11):2013-21. [DOI:10.1016/j.jlumin.2010.05.019]
Tunç S, Duman O, Soylu İ, Bozoğlan BK. Study on the bindings of dichlorprop and diquat dibromide herbicides to human serum albumin by spectroscopic methods. J Hazard Mater 2014; 273:36-43. [DOI:10.1016/j.jhazmat.2014.03.022] [PMID]
Purcell M, Neault J, Malonga H, Arakawa H, Carpentier R, Tajmir-Riahi H. Interactions of atrazine and 2, 4-D with human serum albumin studied by gel and capillary electrophoresis, and FTIR spectroscopy. Biochim Biophys Acta (BBA)-Protein Struct Mol Enzymol. 2001; 1548(1):129-38. [DOI:10.1016/S0167-4838(01)00229-1]
Zhang G, Wang Y, Zhang H, Tang S, Tao W. Human serum albumin interaction with paraquat studied using spectroscopic methods. Pestic Biochem Physiol. 2007; 87(1):23-9. [DOI:10.1016/j.pestbp.2006.05.003]
Zhu M, Wang L, Wang Y, Zhou J, Ding J, Li W, et al. Biointeractions of herbicide atrazine with human serum albumin: UV-Vis, fluorescence and circular dichroism approaches. Int J Environ Res Public Health. 2018; 15(1):116. [DOI:10.3390/ijerph15010116] [PMID] [PMCID]
Miller JN. Recent advances in molecular luminescence analysis. Proc Anal Div Chem Soc; 1979; 16(7):203-8.
Li Y, He W, Dong Y, Sheng F, Hu Z. Human serum albumin interaction with formononetin studied using fluorescence anisotropy, FTIR spectroscopy, and molecular modeling methods. Bioorg Med Chem. 2006; 14(5):1431-6. [DOI:10.1016/j.bmc.2005.09.066] [PMID]
Stephanos JJ, Farina SA, Addison AW. Iron ligand recognition by monomeric hemoglobins. Biochim Biophys Acta (BBA)-Protein Struct Mol Enzymol. 1996; 1295(2):209-21. [DOI:10.1016/0167-4838(96)00041-6]
Zhong W, Wang Y, Yu JS, Liang Y, Ni K, Tu S. The interaction of human serum albumin with a novel antidiabetic agent-SU‐118. J Pharm Sci. 2004; 93(4):1039-46. [DOI:10.1002/jps.20005] [PMID]
Paul BK, Ghosh N, Mukherjee S. Binding interaction of a prospective chemotherapeutic antibacterial drug with β-lactoglobulin: Results and challenges. Langmuir. 2014; 30(20):5921-9. [DOI:10.1021/la501252x] [PMID]
Kragh-Hansen U, Chuang VTG, Otagiri M. Practical aspects of the ligand-binding and enzymatic properties of human serum albumin. Biol Pharm Bull. 2002; 25(6):695-704. [DOI:10.1248/bpb.25.695] [PMID]
Sarver Jr RW, Krueger WC. Protein secondary structure from Fourier transform infrared spectroscopy: A data base analysis. Anal Biochem. 1991; 194(1):89-100. [DOI:10.1016/0003-2697(91)90155-M]
Homans SW. Dynamics and thermodynamics of ligand-protein interactions. In: Peters T, editor. Bioactive Conformation I, Topics in Current Chemistry. Vol. 272. Berlin/Heidelberg: Springer; 2006. p. 51-82. [DOI:10.1007/128_2006_090]
Ross PD, Subramanian S. Thermodynamics of protein association reactions: forces contributing to stability. Biochemistry. 1981; 20(11):3096-102. [DOI:10.1021/bi00514a017] [PMID]
Carter DC, Ho JX. Structure of serum albumin. In: Anfinsen CB, Edsall JT, Richards FM, Eisenberg DS, editors. Advances in Protein Chemistry, Lipoproteins, Apolipoproteins, and Lipases. Vol. 45. Cambridge, MA: Academic Press; 1994. p. 153-203. [DOI:10.1016/S0065-3233(08)60640-3]
Gallagher W. FTIR analysis of protein structure. Course manual Chem. 2009.
Chen L, Wu M, Lin X, Xie Z. Study on the interaction between human serum albumin and a novel bioactive acridine derivative using optical spectroscopy. Luminescence. 2011; 26(3):172-7. [DOI:10.1002/bio.1201] [PMID]
Hadichegeni Sh, Goliaei B, Hashemi M. [Investigation of the Human Serum Albumin (HSA) protein structure change caused by remained diazinon toxin on the food materials (Persian)]. J Arak Uni Med Sci. 2015; 18(7):92-101
Glassford SE, Byrne B, Kazarian SG. Recent applications of ATR FTIR spectroscopy and imaging to proteins. Biochim Biophys Acta Proteins Proteom. 2013; 1834(12):2849-58. [DOI:10.1016/j.bbapap.2013.07.015] [PMID]
Andreas B, Christian Z. What vibrations tell us about proteins. Q Rev Biophys. 2002; 35(4):369-430. [DOI:10.1017/S0033583502003815] [PMID]
Pelton JT, McLean LR. Spectroscopic methods for analysis of protein secondary structure. Anal Biochem. 2000; 277(2):167-76. [DOI:10.1006/abio.1999.4320] [PMID]
Marutescu L, Chifiriuc MC. Molecular mechanisms of pesticides toxicity. In: Grumezescu AM, editor. New Pesticides and Soil Sensors. Cambridge, MA: Academic Press; 2017. p. 393-435. [DOI:10.1016/B978-0-12-804299-1.00012-6]
Goldman SM, Musgrove RE, Jewell SA, Di Monte DA. Pesticides and Parkinson’s disease: Current experimental and epidemiological evidence. In: Aschner M, Costa LG, editors. Advances in Neurotoxicology, Environmental Factors in Neurodegenerative Diseases. Vol. 1. Cambridge: MA: Academic Press; 2017. [DOI:10.1016/bs.ant.2017.07.004]
Bigdeli B, Delavari B, Daryan SS, Saboury AA, Goliaei B. Biophysical and molecular investigation of the interaction between enterolactone and human serum albumin. Biophys J. 2016; 110(3 Suppl 1):49A. [DOI:10.1016/j.bpj.2015.11.330]
Gore MG, editor. Spectrophotometry and spectrofluorimetry: A practical approach. Oxford, UK: Oxford University Press; 2000.
Mabuchi H, Nakahashi H. A major inhibitor of phenytoin binding to serum protein in uremia. Nephron. 1988; 48(4):310-4. [DOI:10.1159/000184949] [PMID]
متن کامل: (3153 مشاهده)
مقدمه
آفتکشها امروزه درکشاورزی استفاده وسیعی دارند و اغلب متعلق به خانوادههای مختلف مانند ارگانوکلرهها (OCs)، کربامات (CBs)، ارگانوفسفرهها (OPs) و پریتروئیدهای مصنوعی (SP) هستند که در گروههای قارچکش، علفکش، حشرهکش، باکتریکش و غیره عرضه میشوند [1]. بر اساس اعلام مؤسسه استاندارد و تحقیقات صنعتی، هر ماده غذاییکشتشده دارای یک « مرز بیشینه مانده آفتکش» است، یعنی بالاترین غلظت مانده یک ماده شیمیایی،که میتواند به صورت قابل قبول با کمترین خطر در ماده غذایی وجود داشته باشد [2].
گاهی تعدادی ازکشاورزان برای جلوگیری از ضرر و دفع آفت، سمها را در دفعات و دزهای بیشتر از استاندارد، در زمان غیرمناسب و نزدیک به برداشت استفاده میکنند و سبب ورود آنها به ماده غذایی میشوند. شواهد علمی زیادی مبنی بر ماندگاری بقایای آفتکشها بر روی گیاهان، خاک، آب، تولیدات مستقیم و غیرمستقیم مواد غذایی درکشاورزی است [3]. طبق تحقیقات انجامشده درکشورهای مختلف روی مواد غذایی مانند انگور، موز، شیر و لبنیات، گوجهفرنگی، سیبزمینی، لوبیا، خربزه، فلفل، دانه گندم،گلابی وگل کلم، باقیمانده آفتکشهایی چون کلروتانولیل، دیازینون، کربندازین، کلوپریفوس، اندوسولفان، متالاکسیل، تریائوفوس و پرمترین بیش از اندازه مجاز در این مواد غذایی یافت شدهاند [4]. استفاده گسترده از سمومکشاورزی از جمله آفتکشها، منجر به نفوذ آنها در جریان خون انسان و تعامل آنها با پروتئینهای پلاسما، مانند پروتئین آلبومین سرم انسانی میشوند و بر عملکرد بیولوژیکی آنها تأثیر میگذارند [6 ،5].
پروتئین HSA یکی از مهمترین و غنیترین پروتئینهای حملکننده ترکیبات شیمیایی و مولکولهای داخلی و خارجی بدن از جمله هورمونهای استروئیدی، اسید آمینهها، اسیدهای چرب، داروها و فلزات در سیستم عروق خونی است و در عین حال به حمل داروهایی که حلالیت کمی در آب دارند کمک کرده و ترکیبات سمی مانند بیلی روبین را برای سمزدایی به کبد منتقل میکند [7]. HSA دارای سه دُمین یا دامنه است که هرکدام دو زیردامنه با شمارهگذاری B و A دارند، این پروتئین دو سایت اصلی و مهم برای اتصال به لیگاندهای شامل سایت یک در زیردامنه IIA و سایت دو در زیردامنه IIIA دارد (تصویر شماره 1) که در جذب، توزیع، متابولیسم و دفع مواد مختلف نقش حیاتی دارند. البته سایتهای اتصال دیگری نیز در پروتئین HSA وجود دارد که اهمیت کمتری دارند [8-10].
اکثر تعاملات دارویی مرتبط با پروتئین HSA در زیردامنه IIA (سایت یک) به خاطر داشتن یک حفره بزرگ هیدروفوبی با شش ساختار مارپیچی (هیلکسی) و یک ساختار حلقه ـ مارپیچ اتفاق میافتد؛ بنابراین سایت یک، مهمترین و اصلیترین جایگاه لیگاندهاست و رقابت بین لیگاندهای مختلف در اتصال به این سایت وجود دارد [8]؛ بنابراین تجزیه و تحلیل تغییرات اعمالشده در ساختار پروتئین HSA و جایگاههای درگیر در تعامل با لیگاندهای مختلف، از جمله آفتکشها در سطح مولکولی میتواند حائز اهمیت بیولوژیکی باشد. به همین منظور این پژوهش، به بررسی مطالعات انجامشده اثر آفتکشها بر پروتئین HSA، به کمک تکنیکهای تجربی و محاسباتی بیوفیزیکی پرداخته است و تمرکز خاصی روی مطالعات ساختاری این پروتئین دارد.
آفتکشها امروزه درکشاورزی استفاده وسیعی دارند و اغلب متعلق به خانوادههای مختلف مانند ارگانوکلرهها (OCs)، کربامات (CBs)، ارگانوفسفرهها (OPs) و پریتروئیدهای مصنوعی (SP) هستند که در گروههای قارچکش، علفکش، حشرهکش، باکتریکش و غیره عرضه میشوند [1]. بر اساس اعلام مؤسسه استاندارد و تحقیقات صنعتی، هر ماده غذاییکشتشده دارای یک « مرز بیشینه مانده آفتکش» است، یعنی بالاترین غلظت مانده یک ماده شیمیایی،که میتواند به صورت قابل قبول با کمترین خطر در ماده غذایی وجود داشته باشد [2].
گاهی تعدادی ازکشاورزان برای جلوگیری از ضرر و دفع آفت، سمها را در دفعات و دزهای بیشتر از استاندارد، در زمان غیرمناسب و نزدیک به برداشت استفاده میکنند و سبب ورود آنها به ماده غذایی میشوند. شواهد علمی زیادی مبنی بر ماندگاری بقایای آفتکشها بر روی گیاهان، خاک، آب، تولیدات مستقیم و غیرمستقیم مواد غذایی درکشاورزی است [3]. طبق تحقیقات انجامشده درکشورهای مختلف روی مواد غذایی مانند انگور، موز، شیر و لبنیات، گوجهفرنگی، سیبزمینی، لوبیا، خربزه، فلفل، دانه گندم،گلابی وگل کلم، باقیمانده آفتکشهایی چون کلروتانولیل، دیازینون، کربندازین، کلوپریفوس، اندوسولفان، متالاکسیل، تریائوفوس و پرمترین بیش از اندازه مجاز در این مواد غذایی یافت شدهاند [4]. استفاده گسترده از سمومکشاورزی از جمله آفتکشها، منجر به نفوذ آنها در جریان خون انسان و تعامل آنها با پروتئینهای پلاسما، مانند پروتئین آلبومین سرم انسانی میشوند و بر عملکرد بیولوژیکی آنها تأثیر میگذارند [6 ،5].
پروتئین HSA یکی از مهمترین و غنیترین پروتئینهای حملکننده ترکیبات شیمیایی و مولکولهای داخلی و خارجی بدن از جمله هورمونهای استروئیدی، اسید آمینهها، اسیدهای چرب، داروها و فلزات در سیستم عروق خونی است و در عین حال به حمل داروهایی که حلالیت کمی در آب دارند کمک کرده و ترکیبات سمی مانند بیلی روبین را برای سمزدایی به کبد منتقل میکند [7]. HSA دارای سه دُمین یا دامنه است که هرکدام دو زیردامنه با شمارهگذاری B و A دارند، این پروتئین دو سایت اصلی و مهم برای اتصال به لیگاندهای شامل سایت یک در زیردامنه IIA و سایت دو در زیردامنه IIIA دارد (تصویر شماره 1) که در جذب، توزیع، متابولیسم و دفع مواد مختلف نقش حیاتی دارند. البته سایتهای اتصال دیگری نیز در پروتئین HSA وجود دارد که اهمیت کمتری دارند [8-10].
اکثر تعاملات دارویی مرتبط با پروتئین HSA در زیردامنه IIA (سایت یک) به خاطر داشتن یک حفره بزرگ هیدروفوبی با شش ساختار مارپیچی (هیلکسی) و یک ساختار حلقه ـ مارپیچ اتفاق میافتد؛ بنابراین سایت یک، مهمترین و اصلیترین جایگاه لیگاندهاست و رقابت بین لیگاندهای مختلف در اتصال به این سایت وجود دارد [8]؛ بنابراین تجزیه و تحلیل تغییرات اعمالشده در ساختار پروتئین HSA و جایگاههای درگیر در تعامل با لیگاندهای مختلف، از جمله آفتکشها در سطح مولکولی میتواند حائز اهمیت بیولوژیکی باشد. به همین منظور این پژوهش، به بررسی مطالعات انجامشده اثر آفتکشها بر پروتئین HSA، به کمک تکنیکهای تجربی و محاسباتی بیوفیزیکی پرداخته است و تمرکز خاصی روی مطالعات ساختاری این پروتئین دارد.
مواد و روشها
برای انجام این مطالعه مروری نظاممند با هدف بررسی اثر آفتکشها بر تغییر ساختار پروتئین HSA به روش بیوفیزیکی، ابتدا با کمک منابع معتبر علمی لیستی از اسامی آفتکشها پرکاربرد کشاورزی با تأکید بر سه گروه حشرهکشها، قارچکشها و علفکشها تهیه شد. این لیست شامل 99 آفتکش در قالب 34 حشرهکش، 34 قارچکش و 41 علفکش بود. سپس برای جستوجوی مقالهها یکبار با کمک واژه کلیدی «نام علمی آفتکش » استخراج شده از 99 آفتکش پرکاربرد به همراه کلمه کلیدی «پروتئین HSA» یا «Human Serum Albumin» انجام شد و بار دوم برای جستوجوی کاملتر از واژگان کلیدی «Insecticide» (حشرهکش)، «Herbicide» (علفکش) و «Fungicide» (قارچکش) استفاده شد که از هرکدام به صورت جداگانه با کلمه کلیدی «پروتئین HSA» یا «Human Serum Albumin» در نمایههای معتبر علمی پابمد، اسکوپوس، وبآوساینس، ساینس دایرکت و موتور جستوجوگر گوگل اسکالر برای جستوجوی مقاله استفاده شد.
برای انجام این مطالعه مروری نظاممند با هدف بررسی اثر آفتکشها بر تغییر ساختار پروتئین HSA به روش بیوفیزیکی، ابتدا با کمک منابع معتبر علمی لیستی از اسامی آفتکشها پرکاربرد کشاورزی با تأکید بر سه گروه حشرهکشها، قارچکشها و علفکشها تهیه شد. این لیست شامل 99 آفتکش در قالب 34 حشرهکش، 34 قارچکش و 41 علفکش بود. سپس برای جستوجوی مقالهها یکبار با کمک واژه کلیدی «نام علمی آفتکش » استخراج شده از 99 آفتکش پرکاربرد به همراه کلمه کلیدی «پروتئین HSA» یا «Human Serum Albumin» انجام شد و بار دوم برای جستوجوی کاملتر از واژگان کلیدی «Insecticide» (حشرهکش)، «Herbicide» (علفکش) و «Fungicide» (قارچکش) استفاده شد که از هرکدام به صورت جداگانه با کلمه کلیدی «پروتئین HSA» یا «Human Serum Albumin» در نمایههای معتبر علمی پابمد، اسکوپوس، وبآوساینس، ساینس دایرکت و موتور جستوجوگر گوگل اسکالر برای جستوجوی مقاله استفاده شد.
زمینه مطالعاتی مقالهها بهخصوص به روش بیوفیزیکی در راستای هدف این مطالعه خیلی غنی نبود، اما با بررسی کلی عنوانها، 30 مقاله در مورد حشرهکشها، 50 مقاله در مورد قارچکشها و 40 مقاله در مورد اثر علفکشها بر پروتئین HSA پیدا و انتخاب شد. سپس با بررسی چکیده مقالهها و در نظر گرفتن معیارهای ورود مقالهها، درمجموع 35 مقاله درباره آفتکشها، 11 مقاله درباره حشرهکشها، 9 مقاله درباره قارچکشها و 15 مقاله درباره علفکشها که با ساختار پروتئین HSA مرتبط بودند، انتخاب و متن کامل آنها دریافت و بررسی شده است. معیارهای ورورد مقالات شامل این موارد بود: مقالههای علمی ـ پژوهشی منتشرشده بین سالهای 1980 تا ماه می 2019، مقالات منتشرشده به زبان انگلیسی و از نمایههای معتبر علمی، مقالههایی که حداقل با یک تکنیک بیوفیزیکی اثر یک آفتکش را روی ساختار پروتئین HSA مورد مطالعه قرار دادهاند. معیارهای خروج نیز شامل این موارد بودند: مطالعههای بیوفیزیکی بیوسنسوری و نانویی درباره آفتکشها و ارتباطشان با پروتئین HSA و مقالههایی که یک نوع آفتکش یکسان را بررسی کرده بودند و مشابه آنها وجود داشت و کیفیت لازم برای تکمیل اطلاعات را نداشتند (تصویر شماره 2).
چند نکته دیگر نیز در این مطالعه حائز اهمیت است: 1. برخی مطالعهها چند آفتکش را همزمان در یک مقاله بررسی کردهاند مانند دیکلوپروپ و دیکوات دیابرومید (البته دو مقاله مربوط به آفتکش متیل تیوفونات هستند که تکمیلکننده اطلاعات همدیگرند). 2. برخی از مقالهها دو پروتئین HSA و BSA (آلبومین سرم گاوی) را بررسی کردهاند که بخش مطالعه مربوط به BSA حذف شده است. با توجه به این نکات در کل طیف مطالعه، 11 حشرهکش (ایمیداکلوپرید، دلتامترین، روتنون، کلرپیریفوس، دیازینون، فورات، تیاکلرپرید، متیل پاراتیون، کلران ترانیلی پرول، پنتا کلروفنل و کربوفوران)، 11 قارچکش (تریادیمفون، پنکونازول، ایمازالیل، میکلوبوتانیل، پروپیکونازول، متیل تیوفونات، دیفن کونازول، متالاکسیل، ادیفنفوس، کاربندازیم و تیوکونازول)، 17 علفکش (نیکوسولفورون، سولفومتررون متیل، گلیفوزات، پرمترین، پندی متالین، متسولفورون متیل، پرایزوسولفورون اتیل، دیکلوفوپ متیل، دیکلوفوپ، دیورون، بنزولفورون متیل، دیکلوپروپ، دیکوات دیابرومید، توفوردی، پاراکوات، آترازین و آمیترول و در کل 39 آفتکش بررسی شدند.
چند نکته دیگر نیز در این مطالعه حائز اهمیت است: 1. برخی مطالعهها چند آفتکش را همزمان در یک مقاله بررسی کردهاند مانند دیکلوپروپ و دیکوات دیابرومید (البته دو مقاله مربوط به آفتکش متیل تیوفونات هستند که تکمیلکننده اطلاعات همدیگرند). 2. برخی از مقالهها دو پروتئین HSA و BSA (آلبومین سرم گاوی) را بررسی کردهاند که بخش مطالعه مربوط به BSA حذف شده است. با توجه به این نکات در کل طیف مطالعه، 11 حشرهکش (ایمیداکلوپرید، دلتامترین، روتنون، کلرپیریفوس، دیازینون، فورات، تیاکلرپرید، متیل پاراتیون، کلران ترانیلی پرول، پنتا کلروفنل و کربوفوران)، 11 قارچکش (تریادیمفون، پنکونازول، ایمازالیل، میکلوبوتانیل، پروپیکونازول، متیل تیوفونات، دیفن کونازول، متالاکسیل، ادیفنفوس، کاربندازیم و تیوکونازول)، 17 علفکش (نیکوسولفورون، سولفومتررون متیل، گلیفوزات، پرمترین، پندی متالین، متسولفورون متیل، پرایزوسولفورون اتیل، دیکلوفوپ متیل، دیکلوفوپ، دیورون، بنزولفورون متیل، دیکلوپروپ، دیکوات دیابرومید، توفوردی، پاراکوات، آترازین و آمیترول و در کل 39 آفتکش بررسی شدند.
نتایج حاصل از تکنیکهای بیوفیزیکی رایج این مطالعات شامل طیفسنجی فرابنفش ـ مرئی، فلورسانس، دورنگنمایی دورانی، تبدیل فوریه ـ مادون قرمز و مدلسازی مولکولی برای این 39 آفتکش در مقاله حاضر گزارش شده است. جهت دسترسی آسان به اثر و خصوصیات هریک از آفتکشها بر پروتئین HSA، اطلاعات گردآوریشده به صورت خلاصه در جدول شماره 1 و 2 این مقاله درج شده است (استخراج اطلاعات بر اساس منابع 12 تا 46 مربوط به 39 آفتکش مطالعهشده در قالب 35 مقاله بوده است).
یافتهها
طیفسنجی فرابنفش ـ مرئی پروتئین HSA در حضور آفتکشها
طیفسنجی فرابنفش ـ مرئی روشی کاربردی، ساده و تقریباً مطمئن برای تأیید برقراری تعامل بین پروتئین و آفتکش و بررسی تغییرات ساختاری پروتئین HSA است [11]. میزان جذب نور از طریق تنها ریشه تریپتوفان پروتئین ASH؛ Trp-214 صورت میگیرد. محدوده اصلی این طیف در 250 تا 290 نانومتر است. اکثر گزارشات با افزایش طول موج به 200 تا 400 نانومتر، امکان بررسی سایر طیفها را نیز فراهم کردهاند. با بررسی 35 مطالعه انتخابشده درباره اثر آفتکشها بر پروتئین HSA، 21 مطالعه از روش طیفسنجی فرابنفش ـ مرئی برای بررسی ساختار پروتئین HSA در حضور آفتکشها استفاده کرده بودند و در همه مطالعات در طیفسنجی فرابنفش ـ مرئی، کاهش و یا افزایش در جذب طبیعی پروتئین HSA مشاهده شده است. علت تغییرات جذب، تغییر قطبیت در محیط اطراف Trp-214 پروتئین در اثر پیوند شدن آفتکشها به پروتئین HSA گزارش شده است که با افزایش غلظت آفتکش، پیک جذب تریپتوفان تغییرات بیشتری را به صورت افزایش یا کاهش در جذب نشان داده است (استخراج اطلاعات بر اساس منابع 12-46، مربوط به 39 آفتکش مطالعهشده در قالب 35 مقاله بوده است).
فلورسانس خاموشی پروتئین HSA در حضور آفتکشها
فلورسانس ذاتی پروتئین HSA ناشی از حضور ریشه آمینواسید تریپتوفان در ساختارپروتئین است که در طول موج 280 نانومتر برانگیخته میشود [47]. با اندازهگیری فلوئورسنت تریپتوفان میتوان به مطالعه ساختار پروتئینها پرداخت و اطلاعاتی در مورد مکانیسمهای اتصال، حالت اتصال، ثابت اتصال و غیره به دست آورد. عواملی چون محیط حلال، pH، حضور مولکولهای کوچک و لیگاندهای مختلف و غیره میتواند منجر به کاهش فلوئورسنت پروتئین شود که به صورت خاموشی و کاهش در نشر فلورسانس ظاهر میشود و نشان میدهد که لیگاند به تریپتوفان یا به یکی از گروههای اطراف آن اتصال یافته است [48]. بر این اساس اکثر مطالعات از روشهایهای مختلف فلورسانس برای بررسی اثر آفتکش بر پروتئین HSA استفاده کردهاند و کاهش نشر طبیعی پروتئین در همه گزارشات مشاهده شده است و با افزایش غلظت آفتکش، شدت نشر فلورسانس پروتئین کاهش بیشتری نشان داده است (استخراج اطلاعات بر اساس منابع 12 تا 46 بوده است). نمونه کاهش نشر فلورسانس پروتئین HSA با افزایش غلظت آفتکش دیازینون در تصویر شماره 3 نشان داده شده است.
ثابت اتصال آفتکشها به پروتئین HSA
محاسبه ثابت اتصال پیوند شدن آفتکش به آلبومین سرم انسانی از طریق تکنیک طیفسنجی فرابنفش ـ مرئی یا فلورسانس انجام میگیرد. محاسبه ثابت پیوندی در روش طیفسنجی فرابنفش ـ مرئی به کمک فرمولهای شماره 1 تا 5 صورت میگیرد:
یافتهها
طیفسنجی فرابنفش ـ مرئی پروتئین HSA در حضور آفتکشها
طیفسنجی فرابنفش ـ مرئی روشی کاربردی، ساده و تقریباً مطمئن برای تأیید برقراری تعامل بین پروتئین و آفتکش و بررسی تغییرات ساختاری پروتئین HSA است [11]. میزان جذب نور از طریق تنها ریشه تریپتوفان پروتئین ASH؛ Trp-214 صورت میگیرد. محدوده اصلی این طیف در 250 تا 290 نانومتر است. اکثر گزارشات با افزایش طول موج به 200 تا 400 نانومتر، امکان بررسی سایر طیفها را نیز فراهم کردهاند. با بررسی 35 مطالعه انتخابشده درباره اثر آفتکشها بر پروتئین HSA، 21 مطالعه از روش طیفسنجی فرابنفش ـ مرئی برای بررسی ساختار پروتئین HSA در حضور آفتکشها استفاده کرده بودند و در همه مطالعات در طیفسنجی فرابنفش ـ مرئی، کاهش و یا افزایش در جذب طبیعی پروتئین HSA مشاهده شده است. علت تغییرات جذب، تغییر قطبیت در محیط اطراف Trp-214 پروتئین در اثر پیوند شدن آفتکشها به پروتئین HSA گزارش شده است که با افزایش غلظت آفتکش، پیک جذب تریپتوفان تغییرات بیشتری را به صورت افزایش یا کاهش در جذب نشان داده است (استخراج اطلاعات بر اساس منابع 12-46، مربوط به 39 آفتکش مطالعهشده در قالب 35 مقاله بوده است).
فلورسانس خاموشی پروتئین HSA در حضور آفتکشها
فلورسانس ذاتی پروتئین HSA ناشی از حضور ریشه آمینواسید تریپتوفان در ساختارپروتئین است که در طول موج 280 نانومتر برانگیخته میشود [47]. با اندازهگیری فلوئورسنت تریپتوفان میتوان به مطالعه ساختار پروتئینها پرداخت و اطلاعاتی در مورد مکانیسمهای اتصال، حالت اتصال، ثابت اتصال و غیره به دست آورد. عواملی چون محیط حلال، pH، حضور مولکولهای کوچک و لیگاندهای مختلف و غیره میتواند منجر به کاهش فلوئورسنت پروتئین شود که به صورت خاموشی و کاهش در نشر فلورسانس ظاهر میشود و نشان میدهد که لیگاند به تریپتوفان یا به یکی از گروههای اطراف آن اتصال یافته است [48]. بر این اساس اکثر مطالعات از روشهایهای مختلف فلورسانس برای بررسی اثر آفتکش بر پروتئین HSA استفاده کردهاند و کاهش نشر طبیعی پروتئین در همه گزارشات مشاهده شده است و با افزایش غلظت آفتکش، شدت نشر فلورسانس پروتئین کاهش بیشتری نشان داده است (استخراج اطلاعات بر اساس منابع 12 تا 46 بوده است). نمونه کاهش نشر فلورسانس پروتئین HSA با افزایش غلظت آفتکش دیازینون در تصویر شماره 3 نشان داده شده است.
ثابت اتصال آفتکشها به پروتئین HSA
محاسبه ثابت اتصال پیوند شدن آفتکش به آلبومین سرم انسانی از طریق تکنیک طیفسنجی فرابنفش ـ مرئی یا فلورسانس انجام میگیرد. محاسبه ثابت پیوندی در روش طیفسنجی فرابنفش ـ مرئی به کمک فرمولهای شماره 1 تا 5 صورت میگیرد:
A0 مقدار جذب پروتئین بدون حضور آفتکش، A جذب کمپلکسهای مختلف در حضور آفتکش است، و ضریب مولی پروتئین و آفتکش است مسیر نور کوت است. درنهایت تفاوت مقادیر جذب A و A0 یعنی A را محاسبه میکنیم. با رسم نمودار در نرمافزار اکسل، محور Y عکس تفاوت جذب و محور X عکس غلظت اولیه لیگاندها (Q) برحسب مولار است و با تقسیم عرض از مبدأ به شیب معادله نمودار مقدار ثابت تشکیل یا ثابت پیوندی (Ka) به دست میآید [50 ،49]. در تکنیک فلورسانس نیز برای به دست آوردن ثابت پیوندی از رابطه هیل (فرمول شماره 6) استفاده شده است:
F نشر پروتئین با حضور آفتکش، F0 نشر پروتئین بدون حضور آفتکش، [Q] غلظت آفتکش و Ka ثابت تجمع یا اتصال پروتئین ـ آفتکش است که با رسم نمودار Log (F0–F) /F علیه Log [Q] معادله نمودار به دست میآید؛ عرض از مبدأ آن، همان Log Ka و شیب آن، تعداد جایگاه اتصال (n) است [51].
بر اساس دادههای محاسبات طیفسنجی فرابنفش ـ مرئی و فلورسانس به دو روش بالا، ثابتهای اتصال آفتکشها به آلبومین سرم انسانی طبق جدول شماره 1 به دست آمدهاند. اکثر ثابتهای اتصال گزارش شده در جدول شماره 1 بر مبنای دمای فیزیولوژی بدن (37 درجه سانتیگراد "310 درجه کلوین") درج شده است و آفتکشهایی که در این دما گزارشی نداشتهاند دامنه دمایی ترجیحاً نزدیک به آن انتخاب شده است. این موارد شامل آفتکشهای آترازین در دمای 34 درجه سانتیگراد (307 درجه کلوین)؛ پندی متالین و دلتامترین در دمای 35 درجه سانتیگراد (308 درجه کلوین)؛ ایمیداکلوپرید، پاراکوات، کلران ترانیلی پرول، نیکوسولفورون، سولفومترون متیل، متسولفورون متیل و پریزوسولفورون اتیل در دمای 36 درجه سانتیگراد (309 درجه کلوین)؛ کاربندازیم، دیکلوفوپ متیل و دیکلوفوپ در دمای 40 درجه سانتیگراد (313 درجه کلوین) هستند. متالاکسیل، ادیفنفوس و تیوکونازول هم تنها در دمای 25 درجه سانتیگراد (298 درجه کلوین) گزارش داشتهاند.
ثابتهای اتصال آفتکش ـ HSA با توجه به دامنههای دمایی ذکر شده برای 39 آفتکش طبق جدول شماره 1 در دو محدوده 1-M103 تا 106 گزارش شدهاند. با یک بررسی کلی «مقدار عددی ثابت اتصال» آفتکشهایی که در دمای 37 درجه سانتیگراد گزارش داشتهاند، به ترتیب شامل آفتکشهای گلیفوزات، تریادیمفون، کربوفوران، دیکوات دیابرومید، ایمازالیل، پنکونازول، میکلوبوتانیل، پرمترین، پروپیکونازول، بنزولفورون متیل، متیل پاراتیون، تیاکلرپرید، متیل تیوفونات، دیورون، توفوردی، دیفن کونازول، فورات، دیازینون، روتنون، دیکلوپروپ، کلرپیریفوس و پنتا کلروفنل هستند که برای دستیابی آسانتر و مشاهده مقدار عددی ثابت اتصال آنها در جدول شماره 1 به صورت رنگی نمایش داده شدهاند. ( استخراج اطلاعات بر اساس منابع 12 تا 46 بوده است).
پیوندها و نیروهای فیزیکی تعامل آفتکش ـ HSA
تجزیه، تحلیل و قضاوت در مورد ماهیت و نوع نیروهای فیزیکی و پیوندهای درگیر در تعامل آفتکش با پروتئین HSA بر اساس پارامترهای مهم ترمودینامیکی شامل تغییرات آنتالپی(ΔH)، تغییرات آنتروپی(ΔS) و تغییرات انرژی آزاد گیبس(ΔG°)که از روش طیفسنجی فلورسانس محاسبه میشوند، انجام میگیرد. عامل برقراری تعامل بین پروتئین و لیگاندها اساساً میانکنشهای هیدروفوبیک، الکترواستاتیک، نیروی واندروالسی و حضور پیوندهای هیدروژنی است. بزرگی و علامت پارامترهای ترمودینامیکی، نوع اینترکشنها، نیروها و پیوندهای دخالتکننده در تعامل آفتکش ـ پروتئین را مشخص میکند [53 ،52]. چنانچه تغییرات آنتروپی و تغییرات آنتالپی محاسبهشده هر دو مثبت باشند، اینترکشن از نوع هیدروفوبیک و اگر هر دو منفی باشند اتصال به شکل پیوند هیدروژنی و نیروی واندروالسی است و چنانچه تغییرات آنتالپی تقریباً برابر صفر و تغییرات آنتروپی مثبت باشند، اینترکشن از نوع الکتروستاتیک است [55 ،54]. توابع ترمودینامیکی لیگاند اتصالشده به پروتئین به کمک فرمولهلی 7 تا 9 تعیین میشود:
پس از بررسی، نوع اینترکشن آفتکش ـ HSA در هر مقاله به کمک پارامترهای ترمودینامیکی در طیفسنجی فلورسانس نتایج بدین شرح استخراج شدند: برای آفتکشهای کلرپیریفوس، دیازینون، تریادیمفون، پنکونازول، ایمازالیل، میکلوبوتانیل، پروپیکونازول، دیفن کونازول و پاراکوات اینترکشن الکتروستاتیکی و هیدروفوبیکی، آفتکشهای گلیکوفوزات، پرمترین، پندی متالین، دیکلوفوب متیل، دیکلوفوب، متیل تیوفونات و نیکوسولفورون اینترکشن هیدروفوبیکی و پیوند هیدروژنی، آفتکشهای کلران ترانیلی پرول، دلتامترین، کربوفوران پریزوسولفورون اتیل نیروی واندروالسی و پیوند هیدروژنی، آفتکشهای دیکلوپروب، دیکوات دیابرمید و آترازین اینترکشن الکتروستاتیک، آفتکشهای؛ فوران، متیل پاراتیون، پنتاکلروفنل، ادیفنفوس، تیوکونازول اینترکشن هیدروفوبیکی، آفتکشهای متسولفورون متیل، دیورون، بنزولفورون متیل و تیاکلروپرید اینترکشن هیدروفوبیک، الکتروستاتیک و پیوند هیدروژنی، آفتکشهای کاربندازیم و امیداکلروپرید اینترکشن هیدروفوبیک، پیوند هیدروژنی و π-π، آفتکش متالاکسیل اینترکشن هیدروفوبیک و پیوند، آفتکش روتنون پیوند هیدروژنی و سولفومترون متیل پیوند هیدروژنی، اینترکشن واندروالسی و هیدروفوبیک به عنوان اینترکشنها و پیوندهای قالب در تعامل با پروتئین HSA گزارش شده است. همانطور که مشخص است به ترتیب اولویت تعامل هیدروفوبیک، پیوند هیدروژنی و تعامل الکتروستاتیک بیشترین سهم را در اینترکشن آفتکشها با پروتئین HSA داشتهاند (استخراج اطلاعات بر اساس منابع 12 تا 46 بوده است).
بررسی تغییرات ساختار دوم پروتئین HSA در حضور آفتکشها
ظرفیت اتصال به مواد خارجی و داخلی در پروتئین HSA به ساختار دوم پروتئین مرتبط میشود. ساختارهای دوم رایج HSA شامل مارپیچ آلفا، ساختارهای بتا ورندوم کویل است. 67 درصد ساختارها مارپیچ آلفا، 10 درصد ساختارها بتا یا ورقهای هستند و بقیه ساختارها 19 درصد را شامل میشوند [56]. در اکثر مطالعات، از روش دورنگمایی دورانی و برخی نیز از روش تبدیل فوریه ـ مادون قرمز) برای بررسی تغییرات ساختاری آفتکش ـ HSA و میزان غیر طبیعی شدن پروتئین، استفاده کردهاند. دقت اندازهگیری ساختار آلفا ـ هلیکس در طیفسنجی CD و ساختارهای بتا در طیفسنجی FTIR بیشتر است [57].
طیفسنجی تبدیل فوریه ـ مادون قرمز (FTIR) پروتئین HSA در حضور آفتکشها
طیفسنجی FTIR دو باند اصلی دارد یکی در ناحیه آمید (I) (CM-11700-1600) است و عمدتاً 70-85 درصد مربوط به ارتعاشاتکششی C=O است که در تشکیل پیوند هیدروژنی درونمولکولی اهمیت زیادی دارد و دیگری در ناحیه آمید (II) (CM-11600-1500) است که حدود 40-60 درصد به ارتعاشات خمشی پیوند N-H و حدود 18-40 درصد به ارتعاشات کششی پیوند C-N برای پروتئین مربوط است. جهت مطالعه درصد تغییر کنفورماسیون ساختارهای دوم پروتئین HSA ناحیه آمید I مدنظر است [57-59]. در تصویر شماره 4 نواحی آمید I، II و موقعیت ساختارهای دوم در آمید I نشان داده شده است [61 ،60].
از 39 آفتکش، آفتکش های دیازینون [17]، متیل تیوفونات [26 ،25]، گلیکوفوزات [34]، پرمترین [35]، دیورون [41] و توفوردی [44] ساختارهای دوم آنها به روش FTIR بررسی شده است که در تمامی موارد ذکرشده ساختار آلفا ـ هلیکس کاهش و سایر ساختارها مانند بتا ـ شیت، بتا ـ ترن و رندم کویل افزایش نشان دادهاند. فقط در یک مورد آفتکش متیل تیوفونات، در بتا ـ ترن پروتئین از 9/30 درصد به 6/26 درصد با حضور آفتکش، کاهش نشان داده است [25]. در برخی از گزارشات آفتکش دیازینون با ماندگاری سم در زمان بیشتر، تغییرات ساختاری بیشتری را نسبت روز اول در ساختار پروتئین HSA ایجاد کرده است [17].
دورنگنمایی دورانی (CD) پروتئین HSA در حضور آفتکشها
طیفسنجی CD از جذب کروموفور آمیدی پیوند پپتیدی در 250-200 نانومتر اندازهگیری میشود که معمولاً باند منفی ساختار مارپیچ یا هلیکال به علت پیوند هیدروژنی قوی در 222 نانومتر است. برای آنالیز تغییرات ساختار دوم پروتئین HSA در حضور آفتکشها از فرمول شماره 10 استفاده میشود [62]:
ظرفیت اتصال به مواد خارجی و داخلی در پروتئین HSA به ساختار دوم پروتئین مرتبط میشود. ساختارهای دوم رایج HSA شامل مارپیچ آلفا، ساختارهای بتا ورندوم کویل است. 67 درصد ساختارها مارپیچ آلفا، 10 درصد ساختارها بتا یا ورقهای هستند و بقیه ساختارها 19 درصد را شامل میشوند [56]. در اکثر مطالعات، از روش دورنگمایی دورانی و برخی نیز از روش تبدیل فوریه ـ مادون قرمز) برای بررسی تغییرات ساختاری آفتکش ـ HSA و میزان غیر طبیعی شدن پروتئین، استفاده کردهاند. دقت اندازهگیری ساختار آلفا ـ هلیکس در طیفسنجی CD و ساختارهای بتا در طیفسنجی FTIR بیشتر است [57].
طیفسنجی تبدیل فوریه ـ مادون قرمز (FTIR) پروتئین HSA در حضور آفتکشها
طیفسنجی FTIR دو باند اصلی دارد یکی در ناحیه آمید (I) (CM-11700-1600) است و عمدتاً 70-85 درصد مربوط به ارتعاشاتکششی C=O است که در تشکیل پیوند هیدروژنی درونمولکولی اهمیت زیادی دارد و دیگری در ناحیه آمید (II) (CM-11600-1500) است که حدود 40-60 درصد به ارتعاشات خمشی پیوند N-H و حدود 18-40 درصد به ارتعاشات کششی پیوند C-N برای پروتئین مربوط است. جهت مطالعه درصد تغییر کنفورماسیون ساختارهای دوم پروتئین HSA ناحیه آمید I مدنظر است [57-59]. در تصویر شماره 4 نواحی آمید I، II و موقعیت ساختارهای دوم در آمید I نشان داده شده است [61 ،60].
از 39 آفتکش، آفتکش های دیازینون [17]، متیل تیوفونات [26 ،25]، گلیکوفوزات [34]، پرمترین [35]، دیورون [41] و توفوردی [44] ساختارهای دوم آنها به روش FTIR بررسی شده است که در تمامی موارد ذکرشده ساختار آلفا ـ هلیکس کاهش و سایر ساختارها مانند بتا ـ شیت، بتا ـ ترن و رندم کویل افزایش نشان دادهاند. فقط در یک مورد آفتکش متیل تیوفونات، در بتا ـ ترن پروتئین از 9/30 درصد به 6/26 درصد با حضور آفتکش، کاهش نشان داده است [25]. در برخی از گزارشات آفتکش دیازینون با ماندگاری سم در زمان بیشتر، تغییرات ساختاری بیشتری را نسبت روز اول در ساختار پروتئین HSA ایجاد کرده است [17].
دورنگنمایی دورانی (CD) پروتئین HSA در حضور آفتکشها
طیفسنجی CD از جذب کروموفور آمیدی پیوند پپتیدی در 250-200 نانومتر اندازهگیری میشود که معمولاً باند منفی ساختار مارپیچ یا هلیکال به علت پیوند هیدروژنی قوی در 222 نانومتر است. برای آنالیز تغییرات ساختار دوم پروتئین HSA در حضور آفتکشها از فرمول شماره 10 استفاده میشود [62]:
θobs؛ CD در millidegree، n تعداد باقیماندههای آمینواسید (585)، l طول سل (یک سانتیمتر)، Cp غلظت مولار HSA.
همچنین آلفا ـ هیلکس برای پروتئین آزاد و ترکیب با آفتکش، از ارزش MRE در 208 نانومتر با استفاده از فرمول شماره 11 محاسبه میشود:
از مجموع 35 مقاله، 26 مقاله ساختار دوم پروتئین را با طیفسنجی CD آنالیز کردند که آفتکشهای تیوکونازول، افزایش ساختار آلفا ـ هلیکس از 55 درصد به 60 درصد و پریزاسولفورون، از 37/45 درصد به 27/51 درصد داشتند. همچنین آترازین کاهش ساختار بتا ـ ترن از 2/28 درصد به 3/ 26 درصد و دیکلوفوپ متیل کاهش درصد بتا ـ شیت در ساختار پروتئین HSA داشتند، در بقیه 23 مورد دیگر از 26 مقاله که از روش طیفسنجی CD برای بررسی تغییرات ساختار پروتئین HSA استفاده کرده بودند کاهش در ساختار آلفا ـ هلیکس و افزایش در سایر ساختارها مانند بتا ـ شیت و بتا ـ ترن گزارش شده است ( استخراج اطلاعات بر اساس منابع 12 تا 46 بوده است).
سایتهای مورد علاقه آفتکشها در پروتئین HSA
برای تعیین جایگاه اتصال مورد هدف آفتکش در تعامل با پروتئین HSA، ریشههای آمینواسید درگیر در پیوند، نوع اینترکشن و نوع پیوند، از روش مدلسازی مولکولی رایانهای در مقالهها استفاده شده است. مدلسازی مولکولی بهترین جایگاه برای اتصال لیگاند به پروتئین HSA، با کمترین انرژی پیوند را تعیین و پیشگویی میکند. پس از دریافت ساختار کریستاله پروتئین سرم آلبومین و آفتکش از بانکهای اطلاعاتی چون Data Base PDB، برای محاسبات Docking میتوان از مجموعه نرمافزاری متداول AutoDock Tools و AutoDock vina، weblab viewer جهت نمایش نتایج و Pose صحیح لیگاند و رسپتور استفاده کرد [17]. با بررسی جایگاه اتصال آفتکشها روی پروتئین HSA در 35 مقاله مطالعهشده از بین حشرهکشها هشت مورد ایمیداکلوپرید، دلتامترین، روتنون، کلرپیریفوس، دیازینون، فورات، تیاکلرپرید و متیل پاراتیون جایگاه یا سایت یک را در زیردامنه IIA، کلران ترانیلی پرول زیردامنه IIIA در سایت دو و پنتا کلروفنل سایت دیگری بین دامنه (دُمین) یک و سه را انتخاب کردهاند. تاکنون هم جایگاه اتصال حشرهکش کربوفوران مورد مطالعه قرار نگرفته است. از بین 11 قارچکش، 10 مورد تریادیمفون، پنکونازول، ایمازالیل، میکلوبوتانیل، پروپیکونازول، متیل تیوفونات، دیفن کونازول، متالاکسیل، ادیفنفوس وکاربندازیم زیردامنه IIA در سایت یک را انتخاب کرده و برای تیوبوکونازول مطالعه بررسی جایگاه اتصال تاکنون صورت نگرفته است.
از 17 علفکش، هفت مورد نیکوسولفورون، سولفومتررون متیل، گلیفوزات، پرمترین، پندی متالین، متسولفورون متیل، پریزوسولفورون اتیل زیردامنه IIA در سایت یک و شش مورد دیکلوفوپ متیل، دیکلوفوپ، دیورون، پریزوسولفورون اتیل، آترازین و بنزولفورون متیل زیردامنه IIIA در سایت دو را برای اتصال به پروتئین HSA انتخاب کردهاند. برای چهار مورد دیکلوپروپ، دیکوات دیابرومید، توفوردی، پاراکوات نیز مطالعه جایگاه اتصال انجام نشده است و سم آمیترول نیز تعاملی با آلبومین سرم انسانی نداشته است. درمجموع از 39 آفتکش، جایگاه اتصال 32 مورد بررسی شده که 25 آفتکش به پروتئین HSA در زیردامنه IIA پیوند شدهاند (استخراج اطلاعات بر اساس منابع 12 تا 46 بوده است).
بحث
طبق تحقیقات مختلف، آفتکشها تأثیر عمدهای بر سلامت عمومی مردم بهویژه آنزیمها، کانالهای یونی و گیرندهها، پروتئینهای حامل از جمله پروتئینهای پلاسما مانند پروتئین HSA که در عملکردهای مهم بیولوژیکی زیستی مانند فرایند سمزدایی شرکت میکنند، دارند و آفتکشها در ایجاد بیماری مختلف از جمله پارکینسون میتوانند دخالت داشته باشند [63-65]. این مطالعه مروری نظاممند نیز بر اساس گزارشات 35 مقاله علمی ـ پژوهشی بیوفیزیکی بیان میدارد آفتکشها توانستهاند باعث تغییر نشر و جذب طبیعی پروتئین پس از اینترکشن با آن شوند و میزان تغییرات در نشر و جذب پروتئین HSA وابسته به غلظت آفتکشهاست. این تغییر در نشر و جذب نشان از تغییرات ساختاری پروتئین HSA ناشی از اثر آفتکشهاست که میتواند در اثر قرارگرفتن آمینواسیدهای تریپتوفان در محیط آبگریز یا آبدوست و یا تغییر موقعیت بار باشد که منجر به افزایش یا کاهش در جذب، خاموشکردن وکاهش نشر پروتئین، تغییر ساختار فضایی، ناپایداری پروتئین و درنهایت تغییردرصدی از ساختار سوم پروتئین HSA متناسب با نوع آفتکش میشوند [66]. از طرفی با بررسی مطالعات طیفسنجی تبدیل فوریه ـ مادون قرمز و دورنگنمایی دورانی مقالههای بررسیشده مشخص شد حضور اکثر آفتکشها باعث کاهش ساختار آلفا ـ هلیکس، افزایش ساختارهای بتا (ورقهای) و ساختارهای نامنظم پروتئین میشوند.
از آنجایی که ساختار دوم پروتئین HSA رابطه نزدیکی با فعالیتهای بیولوژیکی آن دارد [17]، ایجاد تغییرات در ساختار دوم پروتئین هرچند به صورت جزئی، توسط آفتکشها منجر به کاهش فعالیت بیولوژیکی پروتئین HSA میشود. حال باید دید آفتکشها با چه ثابت اتصالی این تغییرات را بر ساختار پروتئین اعمال کردهاند؟ همانطور که میدانیم به طور معمول مقدار عددی اتصال لیگاند به پروتئین بین دامنه 104 تا 106 معکوس مولار (M-1) است [52 ،8]. در تحقیقات دیگری ثابت اتصال ترکیبات خارجی در دمای 25 تا 37 درجه سانتیگراد بین دو دامنه -1M103 تا 105 بیان شده است [16]. با بررسی 35 مقاله درباره اثر آفتکشها بر پروتئین HSA نیز ثابت اتصال آفتکشها به پروتئین، در دو محدوده -1M103 تا 106 گزارش شدهاند که نشان از تعامل به نسبت خوب آفتکشها با پروتئین HSA دارد که برای اعمال اهداف خود و یا تعیین سرنوشت آنها توسط این پروتئین میتواند حائز اهمیت باشد.
به نظر میرسد اتصال آفتکشها به HSA در بیشتر موارد، از طریق اینترکشنهای هیدروفوبیکی و الکتروستاتیکی با حضور پیوندهای هیدروژنی است و اکثر این تعاملها در زیردامنه مهم IIA در سایت یک پروتئین HSA اتفاق میافتند. آفتکشها با دسترسی به Trp-214 پروتئین در زیردامنه IIA باعث تغییر قطبیت محیط اطراف آن و اعمال تغییرات ساختاری در پروتئین میشوند. هرچند زیردامنه IIA ظرفیت اتصال همزمان به چند لیگاند را دارد، اما میتوان نگران جنبه رقابتی آفتکشها نسبت به لیگاندهای مؤثر در بدن بود، مانند داروها و هورمونها که بیشتر آنها اتصالشان در زیردامنه IIA اتفاق میافتد. گزارشاتی مبنی بر اختلال در اتصال داروها و لیگاندهای ضروری به خاطر درگیرشدن پروتئین HSA در توزیع و حذف آفتکشها گزارش شده است که منجر به کاهش غلظت پروتئین آزاد شدهاند [5].
در کل همه آفتکشهای بررسیشده به جز آمیترول، به گونهای بر ساختار پروتئین HSA تغییرات خود را اعمال کردهاند و میزان و درصد این تغییرات بستگی مستقیم به غلظت آفتکش و زمان ماندگاری آنها در جریان خون دارد. این مطالعه فقط 39 مورد از آفتکشها از صدها آفتکش استفادهشده درکشاورزی را بررسی کرده است و اطلاعات کاملی از بین مقالههای موجود در نمایههای معتبر علمی از تأثیرات سایرآفتکشها بر تغییر ساختار پروتئین HSA به روش بیوفیزیکی هنوز گزارش نشده است. وجود مطالعههای بیشتر در مورد بررسی بیوفیزیکی تغییرات ساختار پروتئین HSA در حضور آفتکشها میتواند به ارزیابی جامعتری از تأثیر آفتکشها بر ساختار پروتئین HSA کمک کند. از طرفی این آزمایشات و تفسیر نتایج بر اساس مقالههای علمی ـ پژوهشی بوده که در محیط آزمایشگاهی خارج از بدن انسان انجام شدهاند و نیازمند بررسی و انجام آزمایشات تکمیلی در محیط زنده با رعایت اصول اخلاقی در پژوهشهای آتی است.
نتیجهگیری
به طور کلی این بررسی نشان داد که حضور آفتکشها در جریان خون بدن انسان میتوانند روی HSA (پروتئین مهم پلاسما) در کنار کاهش غلظت آزاد آن، تغییرات ساختاری ـ هرچند به صورت جزئی ـ متناسب با زمان حضور در بدن اعمال کنند و با تغییر کنفورماسیون ساختار دوم و سوم پروتئین HSAدر عملکرد بیولوژیکی آن اختلال ایجاد کنند. با توجه به اینکه HSA یک پروتئین بسیار مهم و پرکاربرد در عملکردهای زیستی بدن انسان به حساب میآید، خطرات آفتکشها و تأثیر آنها بر این پروتئین را نبایستی نادیده گرفت.
ملاحظات اخلاقی
پیروی از اصول اخلاق پژوهش
در این مطالعه، تمامی اصول اخلاق در پژوهش رعایت شده است.
حامی مالی
؟؟
مشارکت نویسندگان
تمامی نویسندگان در نگارش مقاله به یک اندازه مشارکت داشتهاند.
تعارض منافع
نویسندگان این مقاله تصریح میکنند که هیچگونه تضاد منافعی در خصوص پژوهش حاضر وجود ندارد.
References
García FP, Cortés Ascencio SY, Gaytan Oyarzun JC, Hernández AC, Alavarado PV. Pesticides: Classification, uses and toxicity. Measures of exposure and genotoxic risks. J Res Environ Sci Toxicol. 2012; 1(11):279-93.
Commission FWCA. Maximum residue limits for pesticides. FAO/WHO: Rome, Italy. 2001.
Křivánkova L, Boček P, Tekel J, Kovačičová J. Isotachophoretic determination of herbicides prometryne, desmetryne, terbutryne and hydroxy‐derivatives of atrazine and simazine in extracts of milk. Electrophoresis. 1989; 10(10):731-4. [DOI:10.1002/elps.1150101015] [PMID]
Aktar W, Sengupta D, Chowdhury A. Impact of pesticides use in agriculture: their benefits and hazards. Interdiscip Toxicol. 2009; 2(1):1-12. [DOI:10.2478/v10102-009-0001-7] [PMID] [PMCID]
Kragh-Hansen U. Structure and ligand binding properties of human serum albumin. Dan Med Bull. 1990; 37(1):57-84.
Cserháti T, Forgács E. Charge transfer chromatographic study of the binding of commercial pesticides to various albumins. J Chromatogr. 1995; 699(1-2):285-90. [DOI:10.1016/0021-9673(95)00144-C]
Buttar D, Colclough N, Gerhardt S, MacFaul PA, Phillips SD, Plowright A, et al. A combined spectroscopic and crystallographic approach to probing drug-human serum albumin interactions. Bioorg Med Chem. 2010; 18(21):7486-96. [DOI:10.1016/j.bmc.2010.08.052] [PMID]
Zsila F, Bikadi Z, Malik D, Hari P, Pechan I, Berces A, et al. Evaluation of drug-human serum albumin binding interactions with support vector machine aided online automated docking. Bioinformatics. 2011; 27(13):1806-13. [DOI:10.1093/bioinformatics/btr284] [PMID]
Bhattacharya AA, Curry S, Franks NP. Binding of the general anesthetics propofol and halothane to human serum albumin high resolution crystal structures. J Biol Chem. 2000; 275(49):38731-8. [DOI:10.1074/jbc.M005460200] [PMID]
Ghuman J, Zunszain PA, Petitpas I, Bhattacharya AA, Otagiri M, Curry S. Structural basis of the drug-binding specificity of human serum albumin. J Mol Biol. 2005; 353(1):38-52. [DOI:10.1016/j.jmb.2005.07.075] [PMID]
Sahoo BK, Ghosh KS, Dasgupta S. Molecular interactions of isoxazolcurcumin with human serum albumin: Spectroscopic and molecular modeling studies. Biopolymers. 2009; 91(2):108-19. [DOI:10.1002/bip.21092] [PMID]
Wang YQ, Tang BP, Zhang HM, Zhou QH, Zhang GC. Studies on the interaction between imidacloprid and human serum albumin: Spectroscopic approach. J Photochem Photobiol B Biol. 2009; 94(3):183-90. [DOI:10.1016/j.jphotobiol.2008.11.013] [PMID]
Ding F, Liu W, Diao JX, Yin B, Zhang L, Sun Y. Complexation of insecticide chlorantraniliprole with human serum albumin: Biophysical aspects. J Lumin. 2011; 131(7):1327-35. [DOI:10.1016/j.jlumin.2011.03.007]
Wang J, Ma L, Zhang Y, Jiang T. Investigation of the interaction of Deltamethrin (DM) with human serum albumin by multi-spectroscopic method. J Mol Struct. 2017; 1129:160-8. [DOI:10.1016/j.molstruc.2016.09.061]
Fan XY, Zhang Y, Wang J, Yang LY, Jiang FL, Liu Y. Exploring the interaction between rotenone and human serum albumin. J Chem Thermodyn. 2014; 69:186-92. [DOI:10.1016/j.jct.2013.10.021]
Han XL, Tian FF, Ge YS, Jiang FL, Lai L, Li DW, et al. Spectroscopic, structural and thermodynamic properties of chlorpyrifos bound to serum albumin: A comparative study between BSA and HSA. J Photochem Photobiol B biology. 2012; 109:1-11. [DOI:10.1016/j.jphotobiol.2011.12.010] [PMID]
Hadichegeni S, Goliaei B, Taghizadeh M, Davoodmanesh S, Taghavi F, Hashemi M. Characterization of the interaction between human serum albumin and diazinon via spectroscopic and molecular docking methods. Hum Exp Toxicol. 2018; 37(9):959-71. [DOI:10.1177/0960327117741752] [PMID]
Saquib Q, Al-Khedhairy AA, Siddiqui MA, Roy AS, Dasgupta S, Musarrat J. Preferential binding of insecticide phorate with sub-domain IIA of human serum albumin induces protein damage and its toxicological significance. Food Chem Toxicol. 2011; 49(8):1787-95. [DOI:10.1016/j.fct.2011.04.028] [PMID]
Wang C, Chu Q, Chen C, Bo Z. Investigation of the mechanism of binding of thiacloprid to human serum albumin using spectroscopic techniques and molecular modeling methods. J Spectrosc. 2011; 25(2):113-22. [DOI:10.1155/2011/195489]
Silva Dl, Cortez CM, Cunha-Bastos J, Louro SR. Methyl parathion interaction with human and bovine serum albumin. Toxicol Lett. 2004; 147(1):53-61. [DOI:10.1016/j.toxlet.2003.10.014] [PMID]
Wang YQ, Zhang HM, Zhou QH. Investigation of the interaction between pentachlorophenol and human serum albumin using spectral methods. J Mol Struct. 2009; 932(1-3):31-7. [DOI:10.1016/j.molstruc.2009.05.036]
Tunç S, Duman O, Soylu İ, Bozoğlan BK. Spectroscopic investigation of the interactions of carbofuran and amitrol herbicides with human serum albumin. J Lumin. 2014; 151:22-8. [DOI:10.1016/j.jlumin.2014.02.004]
Zhang J, Zhuang S, Tong C, Liu W. Probing the molecular interaction of triazole fungicides with human serum albumin by multispectroscopic techniques and molecular modeling. J Agric Food Chem. 2013; 61(30):7203-11. [DOI:10.1021/jf401095n] [PMID]
Wang C, Li Y. Study on the binding of propiconazole to protein by molecular modeling and a multispectroscopic method. J Agric Food Chem. 2011; 59(15):8507-12. [DOI:10.1021/jf200970s] [PMID]
Li J, Liu X, Ren C, Li J, Sheng F, Hu Z. In vitro study on the interaction between thiophanate methyl and human serum albumin. J Photochem Photobiol B Biol. 2009; 94(3):158-63. [DOI:10.1016/j.jphotobiol.2008.10.001] [PMID]
Saquib Q, Al-Khedhairy AA, Alarifi SA, Dwivedi S, Mustafa J, Musarrat J. Fungicide methyl thiophanate binding at sub-domain IIA of human serum albumin triggers conformational change and protein damage. Int J Biol Macromol. 2010; 47(1):60-7. [DOI:10.1016/j.ijbiomac.2010.03.020] [PMID]
Li Y, Ma X, Lu G. Systematic investigation of the toxic mechanism of difenoconazole on protein by spectroscopic and molecular modeling. Pestic Biochem Physiol. 2013; 105(3):155-60. [DOI:10.1016/j.pestbp.2012.12.010]
Ding F, Li XN, Diao JX, Sun Y, Zhang L, Sun Y. Chiral recognition of metalaxyl enantiomers by human serum albumin: Evidence from molecular modeling and photophysical approach. Chirality. 2012; 24(6):471-80. [DOI:10.1002/chir.22024] [PMID]
Ahmad A, Ahmad M. Understanding the fate of human serum albumin upon interaction with edifenphos: Biophysical and biochemical approaches. Pestic Biochem Physiol. 2018; 145:46-55. [DOI:10.1016/j.pestbp.2018.01.006] [PMID]
Siddiqui MF, Khan MS, Husain FM, Bano B. Deciphering the binding of carbendazim (fungicide) with human serum albumin: A multi-spectroscopic and molecular modelling studies. J Biomol Struct Dyn. 2019; 37(9):2230-41. [DOI:10.1080/07391102.2018.1481768] [PMID]
Staničová J, Želonková K, Verebová V, Holečková B, Dianovský J. Interaction of the fungicide tebuconazole with human serum albumin: A preliminary study. Folia Vet. 2018; 62(2):85-91. [DOI:10.2478/fv-2018-0020]
Ding F, Liu W, Li N, Zhang L, Sun Y. Complex of nicosulfuron with human serum albumin: A biophysical study. J Mol Struct. 2010; 975(1-3):256-64. [DOI:10.1016/j.molstruc.2010.04.033]
Ding F, Liu W, Zhang L, Yin B, Sun Y. Sulfometuron-methyl binding to human serum albumin: Evidence that sulfometuron-methyl binds at the Sudlow’s site I. J Mol Struct. 2010; 968(1-3):59-66. [DOI:10.1016/j.molstruc.2010.01.020]
Yue Y, Zhang Y, Zhou L, Qin J, Chen X. In vitro study on the binding of herbicide glyphosate to human serum albumin by optical spectroscopy and molecular modeling. J Photochem Photobiol B Biol. 2008; 90(1):26-32. [DOI:10.1016/j.jphotobiol.2007.10.003] [PMID]
Wang Y, Zhang G, Wang L. Interaction of prometryn to human serum albumin: Insights from spectroscopic and molecular docking studies. Pestic Biochem Physiol. 2014; 108:66-73. [DOI:10.1016/j.pestbp.2013.12.006] [PMID]
Lee WQ, Affandi ISM, Feroz SR, Mohamad SB, Tayyab S. Evaluation of pendimethalin binding to human serum albumin: Insights from spectroscopic and molecular modeling approach. J Biochem Mol Toxicol. 2017; 31(2):e21839. [DOI:10.1002/jbt.21839] [PMID]
Ding F, Liu W, Zhang X, Zhang L, Sun Y. Fluorescence and circular dichroism studies of conjugates between metsulfuron-methyl and human serum albumin. Colloids Surf B Biointerfaces. 2010; 76(2):441-8. [DOI:10.1016/j.colsurfb.2009.12.003] [PMID]
Ding F, Liu W, Zhang X, Wu LJ, Zhang L, Sun Y. Identification of pyrazosulfuron-ethyl binding affinity and binding site subdomain IIA in human serum albumin by spectroscopic methods. Spectrochim Acta A Mol Biomol Spectrosc. 2010; 75(3):1088-94. [DOI:10.1016/j.saa.2009.12.062] [PMID]
Zhang P, Liu D, Li Z, Shen Z, Wang P, Zhou M, et al. Multispectroscopic and molecular modeling approach to investigate the interaction of diclofop-methyl enantiomers with human serum albumin. J Lumin. 2014; 155:231-7. [DOI:10.1016/j.jlumin.2014.06.040]
Zhang P, Li Z, Wang X, Shen Z, Wang Y, Yan J, et al. Study of the enantioselective interaction of diclofop and human serum albumin by spectroscopic and molecular modeling approaches in vitro. Chirality. 2013; 25(11):719-25. [DOI:10.1002/chir.22204] [PMID]
Chen H, Rao H, Yang J, Qiao Y, Wang F, Yao J. Interaction of diuron to human serum albumin: Insights from spectroscopic and molecular docking studies. J Environ Sci Health B. 2016; 51(3):154-9. [DOI:10.1080/03601234.2015.1108800] [PMID]
Ding F, Liu W, Li Y, Zhang L, Sun Y. Determining the binding affinity and binding site of bensulfuron-methyl to human serum albumin by quenching of the intrinsic tryptophan fluorescence. J Lumin. 2010; 130(11):2013-21. [DOI:10.1016/j.jlumin.2010.05.019]
Tunç S, Duman O, Soylu İ, Bozoğlan BK. Study on the bindings of dichlorprop and diquat dibromide herbicides to human serum albumin by spectroscopic methods. J Hazard Mater 2014; 273:36-43. [DOI:10.1016/j.jhazmat.2014.03.022] [PMID]
Purcell M, Neault J, Malonga H, Arakawa H, Carpentier R, Tajmir-Riahi H. Interactions of atrazine and 2, 4-D with human serum albumin studied by gel and capillary electrophoresis, and FTIR spectroscopy. Biochim Biophys Acta (BBA)-Protein Struct Mol Enzymol. 2001; 1548(1):129-38. [DOI:10.1016/S0167-4838(01)00229-1]
Zhang G, Wang Y, Zhang H, Tang S, Tao W. Human serum albumin interaction with paraquat studied using spectroscopic methods. Pestic Biochem Physiol. 2007; 87(1):23-9. [DOI:10.1016/j.pestbp.2006.05.003]
Zhu M, Wang L, Wang Y, Zhou J, Ding J, Li W, et al. Biointeractions of herbicide atrazine with human serum albumin: UV-Vis, fluorescence and circular dichroism approaches. Int J Environ Res Public Health. 2018; 15(1):116. [DOI:10.3390/ijerph15010116] [PMID] [PMCID]
Miller JN. Recent advances in molecular luminescence analysis. Proc Anal Div Chem Soc; 1979; 16(7):203-8.
Li Y, He W, Dong Y, Sheng F, Hu Z. Human serum albumin interaction with formononetin studied using fluorescence anisotropy, FTIR spectroscopy, and molecular modeling methods. Bioorg Med Chem. 2006; 14(5):1431-6. [DOI:10.1016/j.bmc.2005.09.066] [PMID]
Stephanos JJ, Farina SA, Addison AW. Iron ligand recognition by monomeric hemoglobins. Biochim Biophys Acta (BBA)-Protein Struct Mol Enzymol. 1996; 1295(2):209-21. [DOI:10.1016/0167-4838(96)00041-6]
Zhong W, Wang Y, Yu JS, Liang Y, Ni K, Tu S. The interaction of human serum albumin with a novel antidiabetic agent-SU‐118. J Pharm Sci. 2004; 93(4):1039-46. [DOI:10.1002/jps.20005] [PMID]
Paul BK, Ghosh N, Mukherjee S. Binding interaction of a prospective chemotherapeutic antibacterial drug with β-lactoglobulin: Results and challenges. Langmuir. 2014; 30(20):5921-9. [DOI:10.1021/la501252x] [PMID]
Kragh-Hansen U, Chuang VTG, Otagiri M. Practical aspects of the ligand-binding and enzymatic properties of human serum albumin. Biol Pharm Bull. 2002; 25(6):695-704. [DOI:10.1248/bpb.25.695] [PMID]
Sarver Jr RW, Krueger WC. Protein secondary structure from Fourier transform infrared spectroscopy: A data base analysis. Anal Biochem. 1991; 194(1):89-100. [DOI:10.1016/0003-2697(91)90155-M]
Homans SW. Dynamics and thermodynamics of ligand-protein interactions. In: Peters T, editor. Bioactive Conformation I, Topics in Current Chemistry. Vol. 272. Berlin/Heidelberg: Springer; 2006. p. 51-82. [DOI:10.1007/128_2006_090]
Ross PD, Subramanian S. Thermodynamics of protein association reactions: forces contributing to stability. Biochemistry. 1981; 20(11):3096-102. [DOI:10.1021/bi00514a017] [PMID]
Carter DC, Ho JX. Structure of serum albumin. In: Anfinsen CB, Edsall JT, Richards FM, Eisenberg DS, editors. Advances in Protein Chemistry, Lipoproteins, Apolipoproteins, and Lipases. Vol. 45. Cambridge, MA: Academic Press; 1994. p. 153-203. [DOI:10.1016/S0065-3233(08)60640-3]
Gallagher W. FTIR analysis of protein structure. Course manual Chem. 2009.
Chen L, Wu M, Lin X, Xie Z. Study on the interaction between human serum albumin and a novel bioactive acridine derivative using optical spectroscopy. Luminescence. 2011; 26(3):172-7. [DOI:10.1002/bio.1201] [PMID]
Hadichegeni Sh, Goliaei B, Hashemi M. [Investigation of the Human Serum Albumin (HSA) protein structure change caused by remained diazinon toxin on the food materials (Persian)]. J Arak Uni Med Sci. 2015; 18(7):92-101
Glassford SE, Byrne B, Kazarian SG. Recent applications of ATR FTIR spectroscopy and imaging to proteins. Biochim Biophys Acta Proteins Proteom. 2013; 1834(12):2849-58. [DOI:10.1016/j.bbapap.2013.07.015] [PMID]
Andreas B, Christian Z. What vibrations tell us about proteins. Q Rev Biophys. 2002; 35(4):369-430. [DOI:10.1017/S0033583502003815] [PMID]
Pelton JT, McLean LR. Spectroscopic methods for analysis of protein secondary structure. Anal Biochem. 2000; 277(2):167-76. [DOI:10.1006/abio.1999.4320] [PMID]
Marutescu L, Chifiriuc MC. Molecular mechanisms of pesticides toxicity. In: Grumezescu AM, editor. New Pesticides and Soil Sensors. Cambridge, MA: Academic Press; 2017. p. 393-435. [DOI:10.1016/B978-0-12-804299-1.00012-6]
Goldman SM, Musgrove RE, Jewell SA, Di Monte DA. Pesticides and Parkinson’s disease: Current experimental and epidemiological evidence. In: Aschner M, Costa LG, editors. Advances in Neurotoxicology, Environmental Factors in Neurodegenerative Diseases. Vol. 1. Cambridge: MA: Academic Press; 2017. [DOI:10.1016/bs.ant.2017.07.004]
Bigdeli B, Delavari B, Daryan SS, Saboury AA, Goliaei B. Biophysical and molecular investigation of the interaction between enterolactone and human serum albumin. Biophys J. 2016; 110(3 Suppl 1):49A. [DOI:10.1016/j.bpj.2015.11.330]
Gore MG, editor. Spectrophotometry and spectrofluorimetry: A practical approach. Oxford, UK: Oxford University Press; 2000.
Mabuchi H, Nakahashi H. A major inhibitor of phenytoin binding to serum protein in uremia. Nephron. 1988; 48(4):310-4. [DOI:10.1159/000184949] [PMID]
ارسال پیام به نویسنده مسئول
| بازنشر اطلاعات | |
 |
این مقاله تحت شرایط Creative Commons Attribution-NonCommercial 4.0 International License قابل بازنشر است. |