دوره 22، شماره 5 - ( آذر و دی 1398 )
جلد 22 شماره 5 صفحات 43-32 |
برگشت به فهرست نسخه ها
Download citation:
BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:



BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:
Mondanizadeh M, Moradi N, Amini R, Khansarinejad B, Mosayebi G. Bioinformatic Prediction of miRNAs Targeting APRIL and BAFF Genes in Chronic Lymphocytic Leukemia. J Arak Uni Med Sci 2019; 22 (5) :32-43
URL: http://jams.arakmu.ac.ir/article-1-6105-fa.html
URL: http://jams.arakmu.ac.ir/article-1-6105-fa.html
موندنی زاده مهدیه، مرادی نیلوفر، امینی راضیه، خوانساری نژاد بهزاد، مسیبی قاسم. پیشبینی بیوانفورماتیکی miRNAهای هدف گیرنده ژنهای BAFF و APRIL در بیماران مبتلا به لوسمی لنفوسیتی مزمن. مجله دانشگاه علوم پزشكي اراك. 1398; 22 (5) :32-43
1- دانشگاه علوم پزشکی اراک-
2- دانشگاه علوم پزشکی اراک- ،gmosayebi@yahoo.com
2- دانشگاه علوم پزشکی اراک- ،
متن کامل [PDF 2699 kb]
(2114 دریافت)
| چکیده (HTML) (3729 مشاهده)
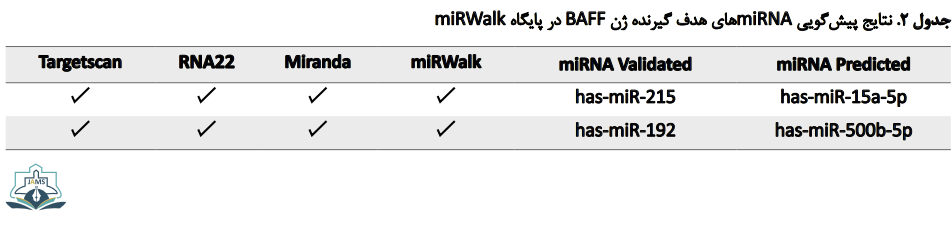
نتایج بررسی در پایگاه miRWalk حاکی از این بود که miR-15a-5p ،miR-500b-5p و miR-4803 به صورت پیشگوییشده و miR-215 و miR-192 به صورت تأیید شده، از تارگتکنندههای ژن BAFF در پایگاه میرواک هستند. به علاوه نتایج این پایگاه توسط پایگاهای miRanda، TargetScan و RNA22 نیز تأیید میشود. این نتایج به ترتیب در جدول شماره 2 نشان داده شده است.
نتایج بررسی در پایگاه DIANA نشاندهنده هدفگیری ژن BAFF توسط miR-424 و miR-497 به ترتیب با امتیاز 952/0 و 916/0 است. این نتایج به ترتیب در جدول شماره 3 نشان داده شده است. نتایج بررسی در پایگاه miRDB نشان میدهد miR-15a-5p ،miR-497 و miR-424 با بالاترین امتیاز ژن BAFF را مورد هدف قرار میدهند. نتایج در جدول شماره 4 قابل مشاهده است.
نتایج بررسی در پایگاه miRanda نشان میدهد hsa-miR-544 ژن BAFF را مورد هدف قرار میدهند. نتایج در جدول شماره 5 قابل مشاهده است.
نتایج حاصل از پیشگویی miRNAهای هدف گیرنده ژن APRIL در پایگاههای اطلاعاتی Targetscan، DIANA، miRWalk و miRanda
نتایج بررسی در پایگاه TargetScan حاکی از هدفگیری ژن APRIL توسط miR-145-5p و miR-5195 با بالاترین امتیاز و بهترین شرایط اتصال است. این نتایج در جدول شماره 6 نشان داده شده است.
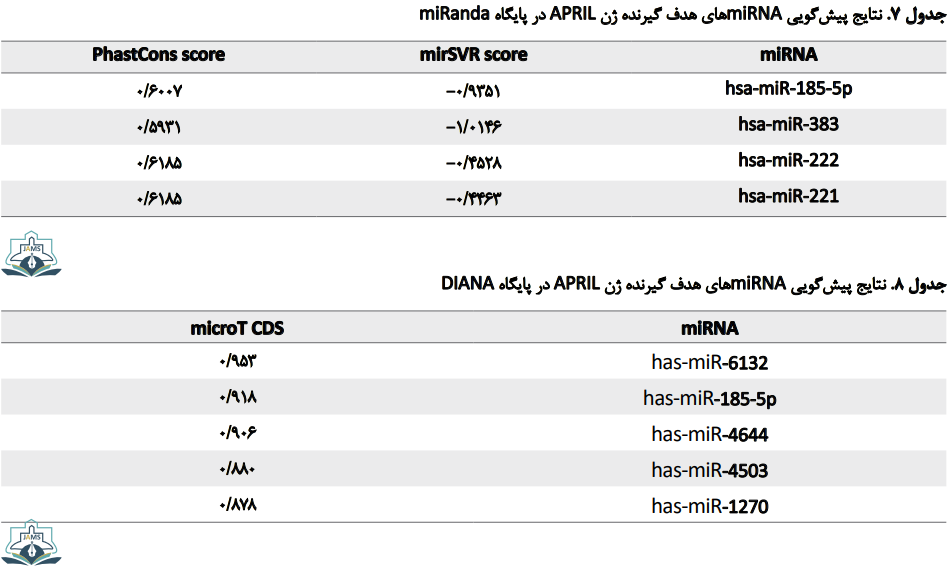
نتایج بررسی در پایگاه miRanda نشان میدهد miR-185 با بالاترین امتیاز و با اتصال مناسبتر میتواند ژن APRIL را مورد هدف قرار دهد. این نتایج در جدول شماره 7 نشان داده شده است.
نتایج بررسی در پایگاه DIANA نشاندهنده هدفگیری ژن APRIL توسط miR-6132 و miR-185-5p با بالاترین امتیاز است. این نتایج به ترتیب در جدول شماره 8 نشان داده شده است.
نتایج بررسی در پایگاه miRWalk حاکی از وجود miRNAهای متعددی است که هم به صورت پیشگوییشده و هم به صورت تأییدشده، از تارگتکنندههای ژن APRIL هستند که این نتایج توسط پایگاهای miRanda، TargetScan و RNA22 مورد تأیید هستند. نتایج در جدول شماره 9 نشان داده شده است.
نتایج بررسی در پایگاه miRDB نشاندهنده هدفگیری ژن APRIL توسط hsa-miR-6716-5p و hsa-miR-4306 با بالاترین امتیاز است. نتایج در جدول به شماره 10 قابل مشاهده است.
نحوه گزینش و انتخاب miRNAهای برتر
نحوه انتخاب و گزینش miRNA هدف گیرنده ژن BAFF در جدول شماره 11 و miRNA هدف گیرنده ژن APRIL در جدول شماره 12 قابل مشاهده است. بدین ترتیب در جدول شماره 11 و 12 miRNAهای با امتیاز بالاتر و اتصال بهتر با ژنهای هدف، انتخاب و گردآوری شدند. سپس از بین آنها miRNAهایی با بیشترین تکرار و تأیید در پایگاههای مورد مطالعه به عنوان miRNAهای هدف گیرنده این ژنها جهت بررسی در فاز عملی این پژوهش انتخاب شدند.
همچنین miRNAهای منتخب (miR-145-5p ،miR-185 ،miR-424 و miR-497) برای بررسی میزان بیان در افراد مبتلا به سرطان خون و افراد گروه کنترل (افراد سالم) در مطالعات فاز عملی در جدول 13 نشان داده شده است.
متن کامل: (2058 مشاهده)
مقدمه
سرطان خون یکی از شایعترین سرطانهاست که به نظر میرسد به واسطه نقص در تمایز سلولی ایجاد میشود. لوسمی لنفوسیتی مزمن شایعترین لوسمی در بزرگسالان در کشورهای غربی است که حدود 25 تا 30 درصد از کل لوسمیها را شامل میشود [2 ،1]. لوسمی لنفوسیتی مزمن یک بیماری کلونال سلولهای بنیادی خونساز است که در آن روند رشد و نمو لنفوسیتها بهدرستی انجام نمیگیرد و تعداد بسیار زیادی از آنها ساخته میشود. اگرچه ظاهر آنها ممکن است طبیعی به نظر آید، قابلیت مبارزه با عفونت را آنطور که باید داشته باشند، ندارند. این لنفوسیتهای نابالغ را میتوان در خون، مغز استخوان و ارگانهای لنفوئیدی مشاهده کرد [4 ،3].
لوسمی لنفوسیتی مزمن دارای سیر بالینی متغیری است؛ بدین صورت که برخی بیماران نیاز به درمان ندارند، در حالی که بعضی از آنها بیماری پیشرونده و تهاجمی داشته و عمر کوتاهی دارند. بنابراین عوامل پیشآگهیدهنده در شناسایی بیماران با پیشآگهی ضعیف در مقایسه با بیماران با پیشآگهی بهتر، به همراه تشخیص زمان شروع درمان، حائز اهمیت است [1]. بروز CLL به دو صورت تهاجمی و خفیف نمایان میشود [5]. لوسمی لنفوسیتی مزمن داری علل اتیولوژیک متفاوت است. یکی از علل اتیولوژیک CLL تغییرات ژنتیکی است که این تغییرات به وسیله تکنیک FISH در بیش از 80 درصد از موارد تشخیص داده میشود [6]. از دیگر علل این بیماری اختلال در برخی از مسیرهای سیگنالینگ سلولی است. لذا شناسایی و ارزیابی پروتئینهای دخیل در مسیر سیگنالینگ مولکولی CLL که بیان آنها نسبت به حالت نرمال تغییر میکند، میتواند منجر به ایجاد یک روش تشخیصی و یا درمانی جدید و مؤثر شود [7].
از جمله مسیرهای سیگنالینگ سلولی دخیل در ایجاد این نوع بدخیمی خونی مسیر NF-kβ است. یکی از اعضای خانواده فاکتور نکروز دهنده تومور (TNF) که در مسیر NF-kβ دخیل است، لیگاند القاکننده تکثیر (APRIL) است که به طور گستردهای در سلولهای هماتوپویتیک بیان میشود [8]. این پروتئین به طور قابل توجهی در سرم بیماران مبتلا به CLL افزایش مییابد [9] و با تمایل بالا به دو گیرنده BCMA و TAC1 متصل شده و بدین واسطه موجب فعالشدن TRAFs و تحریک مسیر NF-kβ و درنهایت مهار آپوپتوز در سلولهای B میشود [8]. از سویی دیگر BAFF سایتوکاینی گلیکوپروتئینی و متعلق به خانواده لیگاندهای عامل نکروز توموری است. BAFF نقش مهمی در تکثیر و تمایز لنفوسیت B ایفا میکند و این اثر را از طریق اتصال به سه گیرنده سطح سلولهای B به نامهای TAC1، BCMA و BAFF-R اعمال میکند [11 ،10]. این سایتوکاین مسیرکلاسیک NF-kβ را فعال میکند و با فعالشدن این مسیر BAFF همانند APRIL منجر به مهار آپوپتوز سلولهای B میشود و درنهایت سلول به سمت ایجاد تومور و سرطانیشدن پیشروی میکند [12 ،7].
در سالهای اخیر محققان دریافتند برخی miRNAها از طریق کاهش بیان و خاموشکردن ژنها منجر به روند سرطانیشدن سلولها میشوند که همین امر تأییدکننده پتانسیل مهم این عوامل مولکولی به عنوان بیومارکر در تشخیص، پیشآگهی، پیشرفت، درمان و مقاومت دارویی انواع سرطان از جمله CLL است [14 ،13]. درواقع آنها با مهار mRNA هدف، به تعدیل سطح بیان پروتئینهای خاص میپردازند. بهعلاوه miRNAها به عنوان مولکولهای کلیدی در کنترل طیف گستردهای از فعالیتهای سلولی مانند تکثیر، مرگ، تمایز، تحرک و تهاجم دخالت دارند [16 ،15]. سطوح بیان miRNAها به عنوان ابزاری جهت تشخیص انواع مختلف CLL یا برای پیگیری پیشرفت بیماری به کار میرود.
لذا در این مطالعه با توجه به نقش کلیدی پروتئینهای APRIL و BAFF در مسیر انکوژنز CLL و اهمیت اثر تنظیمی miRNAها در بسیاری از فرایندهای سلولی، به پیشگویی miRNAهای شاخص هدف گیرنده ترانسکریپتهای مربوط به APRIL و BAFF با استفاده از نرمافزارهای بیوانفورماتیکی مختلف و اختصاصی پرداخته شد. بدین ترتیب با توجه به دادههای حاصل از نرمافزارهای موجود با الگوریتم مختلف، miRNAهایی که بالاترین امتیاز و بیشترین احتمال را در هدفگیری ژنهای مذکور داشتند، به منظور بررسیهای عملی و آزمایشگاهی در پژوهشهای بعدی انتخاب شدند.
مواد و روشها
پژوهش حاضر یک بررسی تئوری بیوانفورماتیک است.
پیشگویی miRNAهای هدف گیرنده ژنهای BAFF و APRILبه وسیله پایگاه میراندا
این پایگاه اطلاعاتی نتایج حاصل از ارزیابیهای بیوانفورماتیک را با ارائه شاخصی به نام miRSVR نشان میدهد. اساس رتبهبندی در این مدل بر پایه توالی و خصوصیات زمینهای دابلکس mRNA-miRNA است. برای پیشگویی miRNA در این سایت بعد از ورود به صفحه اصلی سایت وارد بخش Target mRNA شدیم و نام ژنهای BAFF و APRIL و گونه Homo sapiens را انتخاب کردیم و جستوجو انجام شد و سایت در صفحه نتایج miRNAهای تارگتکننده ژنها را نمایش داد.
پیشگویی miRNAهای هدف گیرنده ژنهای BAFF و APRIL در پایگاه تارگت اسکن
این پایگاه اطلاعاتی نتایج حاصل از ارزیابیهای بیوانفورماتیک را بر اساس فاکتوری به نام Pct یا احتمال هدفگیری محافظتشده در انسان و پستانداران، موش، کِرم، مگس و قورباغه ارائه میدهد. این سایت بر اساس نحوه اتصال نوکلئوتیدهای ناحیه سید هر miRNA به mRNA هدف، امکان هدفیابی یک ژن توسط یک miRNA را ارزیابی میکند. برای پیشگویی miRNA در این سایت شماره Ensembl ژنهای BAFF و APRIL و همچنین اسم گونه Homo sapiens برای جستوجو استفاده شد.
پیشگویی miRNAهای هدف گیرنده ژنهای BAFF و APRIL در پایگاه میرواک
این پایگاه داده، اطلاعات miRNAهای انسان، موش و رت را به دو صورت پیشبینی شده و به صورت تجربی در اختیار محقق قرار میدهد. الگوریتم بهکاررفته در این نرمافزار بر اساس پیشبینی محل اتصال miRNA در توالیهای کامل تمام ژنومهای شناختهشده (حتی میتوکندری) در موجودات نامبرده است. در این سایت یکبار جستوجو بر اساس مدل پیشگویی و یکبار بر اساس مدل تأییدشده و بر اساس اسامی مختلف ژنهای BAFF و APRIL انجام شد.
پیشگویی miRNAهای هدف گیرنده ژنهای BAFF و APRIL در پایگاه دایانا
مبنای ارزشیابی این پایگاه اطلاعاتی برای هر پیشبینی، شاخصی با عنوان نمره دقت یا miTG score است. این پایگاه با چندین پایگاه بنام دیگر، مانند HUGO Ensembl, UCSC SwissProt مرتبط است. الگوریتم مورد استفاده در این سایت برپایه مجموع امتیازات نواحی محافظتشده و غیرمحافظتشده با هم و ارائه یک امتیاز کلی است که حاکی از تغییرات بیان mRNA هدف است. در این پایگاه نیز جستوجو بر اساس نام ژنهای BAFF و APRIL در بخش SOFTWARE انجام گرفت.
پیشگویی miRNAهای هدف گیرنده ژن BAFF و APRIL در پایگاه میر دی بی
در این پایگاه اطلاعاتی آنلاین پیشبینی miRNA برای پنج گونه انسان، موش، رت، سگ و مرغ انجام میگیرد و جستوجو میتواند بر اساس نام miRNA مورد نظر و یا نام ژن هدف انجام میشود. نمره ارائهشده در این پایگاه بین 50 تا 100 است و هرچه نمره کسبشده به 100 نزدیکتر باشد، احتمال واقعیبودن احتمال اتصال miRNA به mRNA هدف بیشتر است. در این پایگاه، جستوجو بر اساس نام ژنهای BAFF و APRIL انجام شد.
نحوه گزینش و انتخاب miRNAهای برتر
در ابتدا نتایج خروجی از هر پایگاه اطلاعاتی بر اساس بالاتربودن امتیاز هر miRNA در فایل Excel ذخیره شد. سپس از میان دادهها، miRNAهایی که دارای بیشترین احتمال رابطه مکملی با ژنهای BAFF و APRIL بودند و در تعداد بیشتری از نرمافزارها پیشگویی و تأیید شدند، برای مطالعات عملی در آینده انتخاب شدند.
یافتهها
نتایج حاصل از پیشگویی miRNAهای هدف گیرنده ژن BAFF در پایگاههای اطلاعاتی Targetscan ،DIANA ،miRDB، miRWalk و miRanda
نتایج بررسی در پایگاه Targetscan نشان میدهد miR195-5p ،miR15a-5p ،miR15b-5p ،miR-497 miR424 و miR-16-5p ژن BAFF را مورد هدف قرار میدهند. این نتایج در جدول شماره 1 نشان داده شده است.
سرطان خون یکی از شایعترین سرطانهاست که به نظر میرسد به واسطه نقص در تمایز سلولی ایجاد میشود. لوسمی لنفوسیتی مزمن شایعترین لوسمی در بزرگسالان در کشورهای غربی است که حدود 25 تا 30 درصد از کل لوسمیها را شامل میشود [2 ،1]. لوسمی لنفوسیتی مزمن یک بیماری کلونال سلولهای بنیادی خونساز است که در آن روند رشد و نمو لنفوسیتها بهدرستی انجام نمیگیرد و تعداد بسیار زیادی از آنها ساخته میشود. اگرچه ظاهر آنها ممکن است طبیعی به نظر آید، قابلیت مبارزه با عفونت را آنطور که باید داشته باشند، ندارند. این لنفوسیتهای نابالغ را میتوان در خون، مغز استخوان و ارگانهای لنفوئیدی مشاهده کرد [4 ،3].
لوسمی لنفوسیتی مزمن دارای سیر بالینی متغیری است؛ بدین صورت که برخی بیماران نیاز به درمان ندارند، در حالی که بعضی از آنها بیماری پیشرونده و تهاجمی داشته و عمر کوتاهی دارند. بنابراین عوامل پیشآگهیدهنده در شناسایی بیماران با پیشآگهی ضعیف در مقایسه با بیماران با پیشآگهی بهتر، به همراه تشخیص زمان شروع درمان، حائز اهمیت است [1]. بروز CLL به دو صورت تهاجمی و خفیف نمایان میشود [5]. لوسمی لنفوسیتی مزمن داری علل اتیولوژیک متفاوت است. یکی از علل اتیولوژیک CLL تغییرات ژنتیکی است که این تغییرات به وسیله تکنیک FISH در بیش از 80 درصد از موارد تشخیص داده میشود [6]. از دیگر علل این بیماری اختلال در برخی از مسیرهای سیگنالینگ سلولی است. لذا شناسایی و ارزیابی پروتئینهای دخیل در مسیر سیگنالینگ مولکولی CLL که بیان آنها نسبت به حالت نرمال تغییر میکند، میتواند منجر به ایجاد یک روش تشخیصی و یا درمانی جدید و مؤثر شود [7].
از جمله مسیرهای سیگنالینگ سلولی دخیل در ایجاد این نوع بدخیمی خونی مسیر NF-kβ است. یکی از اعضای خانواده فاکتور نکروز دهنده تومور (TNF) که در مسیر NF-kβ دخیل است، لیگاند القاکننده تکثیر (APRIL) است که به طور گستردهای در سلولهای هماتوپویتیک بیان میشود [8]. این پروتئین به طور قابل توجهی در سرم بیماران مبتلا به CLL افزایش مییابد [9] و با تمایل بالا به دو گیرنده BCMA و TAC1 متصل شده و بدین واسطه موجب فعالشدن TRAFs و تحریک مسیر NF-kβ و درنهایت مهار آپوپتوز در سلولهای B میشود [8]. از سویی دیگر BAFF سایتوکاینی گلیکوپروتئینی و متعلق به خانواده لیگاندهای عامل نکروز توموری است. BAFF نقش مهمی در تکثیر و تمایز لنفوسیت B ایفا میکند و این اثر را از طریق اتصال به سه گیرنده سطح سلولهای B به نامهای TAC1، BCMA و BAFF-R اعمال میکند [11 ،10]. این سایتوکاین مسیرکلاسیک NF-kβ را فعال میکند و با فعالشدن این مسیر BAFF همانند APRIL منجر به مهار آپوپتوز سلولهای B میشود و درنهایت سلول به سمت ایجاد تومور و سرطانیشدن پیشروی میکند [12 ،7].
در سالهای اخیر محققان دریافتند برخی miRNAها از طریق کاهش بیان و خاموشکردن ژنها منجر به روند سرطانیشدن سلولها میشوند که همین امر تأییدکننده پتانسیل مهم این عوامل مولکولی به عنوان بیومارکر در تشخیص، پیشآگهی، پیشرفت، درمان و مقاومت دارویی انواع سرطان از جمله CLL است [14 ،13]. درواقع آنها با مهار mRNA هدف، به تعدیل سطح بیان پروتئینهای خاص میپردازند. بهعلاوه miRNAها به عنوان مولکولهای کلیدی در کنترل طیف گستردهای از فعالیتهای سلولی مانند تکثیر، مرگ، تمایز، تحرک و تهاجم دخالت دارند [16 ،15]. سطوح بیان miRNAها به عنوان ابزاری جهت تشخیص انواع مختلف CLL یا برای پیگیری پیشرفت بیماری به کار میرود.
لذا در این مطالعه با توجه به نقش کلیدی پروتئینهای APRIL و BAFF در مسیر انکوژنز CLL و اهمیت اثر تنظیمی miRNAها در بسیاری از فرایندهای سلولی، به پیشگویی miRNAهای شاخص هدف گیرنده ترانسکریپتهای مربوط به APRIL و BAFF با استفاده از نرمافزارهای بیوانفورماتیکی مختلف و اختصاصی پرداخته شد. بدین ترتیب با توجه به دادههای حاصل از نرمافزارهای موجود با الگوریتم مختلف، miRNAهایی که بالاترین امتیاز و بیشترین احتمال را در هدفگیری ژنهای مذکور داشتند، به منظور بررسیهای عملی و آزمایشگاهی در پژوهشهای بعدی انتخاب شدند.
مواد و روشها
پژوهش حاضر یک بررسی تئوری بیوانفورماتیک است.
پیشگویی miRNAهای هدف گیرنده ژنهای BAFF و APRILبه وسیله پایگاه میراندا
این پایگاه اطلاعاتی نتایج حاصل از ارزیابیهای بیوانفورماتیک را با ارائه شاخصی به نام miRSVR نشان میدهد. اساس رتبهبندی در این مدل بر پایه توالی و خصوصیات زمینهای دابلکس mRNA-miRNA است. برای پیشگویی miRNA در این سایت بعد از ورود به صفحه اصلی سایت وارد بخش Target mRNA شدیم و نام ژنهای BAFF و APRIL و گونه Homo sapiens را انتخاب کردیم و جستوجو انجام شد و سایت در صفحه نتایج miRNAهای تارگتکننده ژنها را نمایش داد.
پیشگویی miRNAهای هدف گیرنده ژنهای BAFF و APRIL در پایگاه تارگت اسکن
این پایگاه اطلاعاتی نتایج حاصل از ارزیابیهای بیوانفورماتیک را بر اساس فاکتوری به نام Pct یا احتمال هدفگیری محافظتشده در انسان و پستانداران، موش، کِرم، مگس و قورباغه ارائه میدهد. این سایت بر اساس نحوه اتصال نوکلئوتیدهای ناحیه سید هر miRNA به mRNA هدف، امکان هدفیابی یک ژن توسط یک miRNA را ارزیابی میکند. برای پیشگویی miRNA در این سایت شماره Ensembl ژنهای BAFF و APRIL و همچنین اسم گونه Homo sapiens برای جستوجو استفاده شد.
پیشگویی miRNAهای هدف گیرنده ژنهای BAFF و APRIL در پایگاه میرواک
این پایگاه داده، اطلاعات miRNAهای انسان، موش و رت را به دو صورت پیشبینی شده و به صورت تجربی در اختیار محقق قرار میدهد. الگوریتم بهکاررفته در این نرمافزار بر اساس پیشبینی محل اتصال miRNA در توالیهای کامل تمام ژنومهای شناختهشده (حتی میتوکندری) در موجودات نامبرده است. در این سایت یکبار جستوجو بر اساس مدل پیشگویی و یکبار بر اساس مدل تأییدشده و بر اساس اسامی مختلف ژنهای BAFF و APRIL انجام شد.
پیشگویی miRNAهای هدف گیرنده ژنهای BAFF و APRIL در پایگاه دایانا
مبنای ارزشیابی این پایگاه اطلاعاتی برای هر پیشبینی، شاخصی با عنوان نمره دقت یا miTG score است. این پایگاه با چندین پایگاه بنام دیگر، مانند HUGO Ensembl, UCSC SwissProt مرتبط است. الگوریتم مورد استفاده در این سایت برپایه مجموع امتیازات نواحی محافظتشده و غیرمحافظتشده با هم و ارائه یک امتیاز کلی است که حاکی از تغییرات بیان mRNA هدف است. در این پایگاه نیز جستوجو بر اساس نام ژنهای BAFF و APRIL در بخش SOFTWARE انجام گرفت.
پیشگویی miRNAهای هدف گیرنده ژن BAFF و APRIL در پایگاه میر دی بی
در این پایگاه اطلاعاتی آنلاین پیشبینی miRNA برای پنج گونه انسان، موش، رت، سگ و مرغ انجام میگیرد و جستوجو میتواند بر اساس نام miRNA مورد نظر و یا نام ژن هدف انجام میشود. نمره ارائهشده در این پایگاه بین 50 تا 100 است و هرچه نمره کسبشده به 100 نزدیکتر باشد، احتمال واقعیبودن احتمال اتصال miRNA به mRNA هدف بیشتر است. در این پایگاه، جستوجو بر اساس نام ژنهای BAFF و APRIL انجام شد.
نحوه گزینش و انتخاب miRNAهای برتر
در ابتدا نتایج خروجی از هر پایگاه اطلاعاتی بر اساس بالاتربودن امتیاز هر miRNA در فایل Excel ذخیره شد. سپس از میان دادهها، miRNAهایی که دارای بیشترین احتمال رابطه مکملی با ژنهای BAFF و APRIL بودند و در تعداد بیشتری از نرمافزارها پیشگویی و تأیید شدند، برای مطالعات عملی در آینده انتخاب شدند.
یافتهها
نتایج حاصل از پیشگویی miRNAهای هدف گیرنده ژن BAFF در پایگاههای اطلاعاتی Targetscan ،DIANA ،miRDB، miRWalk و miRanda
نتایج بررسی در پایگاه Targetscan نشان میدهد miR195-5p ،miR15a-5p ،miR15b-5p ،miR-497 miR424 و miR-16-5p ژن BAFF را مورد هدف قرار میدهند. این نتایج در جدول شماره 1 نشان داده شده است.
نتایج بررسی در پایگاه miRWalk حاکی از این بود که miR-15a-5p ،miR-500b-5p و miR-4803 به صورت پیشگوییشده و miR-215 و miR-192 به صورت تأیید شده، از تارگتکنندههای ژن BAFF در پایگاه میرواک هستند. به علاوه نتایج این پایگاه توسط پایگاهای miRanda، TargetScan و RNA22 نیز تأیید میشود. این نتایج به ترتیب در جدول شماره 2 نشان داده شده است.
نتایج بررسی در پایگاه DIANA نشاندهنده هدفگیری ژن BAFF توسط miR-424 و miR-497 به ترتیب با امتیاز 952/0 و 916/0 است. این نتایج به ترتیب در جدول شماره 3 نشان داده شده است. نتایج بررسی در پایگاه miRDB نشان میدهد miR-15a-5p ،miR-497 و miR-424 با بالاترین امتیاز ژن BAFF را مورد هدف قرار میدهند. نتایج در جدول شماره 4 قابل مشاهده است.
نتایج بررسی در پایگاه miRanda نشان میدهد hsa-miR-544 ژن BAFF را مورد هدف قرار میدهند. نتایج در جدول شماره 5 قابل مشاهده است.
نتایج حاصل از پیشگویی miRNAهای هدف گیرنده ژن APRIL در پایگاههای اطلاعاتی Targetscan، DIANA، miRWalk و miRanda
نتایج بررسی در پایگاه TargetScan حاکی از هدفگیری ژن APRIL توسط miR-145-5p و miR-5195 با بالاترین امتیاز و بهترین شرایط اتصال است. این نتایج در جدول شماره 6 نشان داده شده است.
نتایج بررسی در پایگاه miRanda نشان میدهد miR-185 با بالاترین امتیاز و با اتصال مناسبتر میتواند ژن APRIL را مورد هدف قرار دهد. این نتایج در جدول شماره 7 نشان داده شده است.
نتایج بررسی در پایگاه DIANA نشاندهنده هدفگیری ژن APRIL توسط miR-6132 و miR-185-5p با بالاترین امتیاز است. این نتایج به ترتیب در جدول شماره 8 نشان داده شده است.
نتایج بررسی در پایگاه miRWalk حاکی از وجود miRNAهای متعددی است که هم به صورت پیشگوییشده و هم به صورت تأییدشده، از تارگتکنندههای ژن APRIL هستند که این نتایج توسط پایگاهای miRanda، TargetScan و RNA22 مورد تأیید هستند. نتایج در جدول شماره 9 نشان داده شده است.
نتایج بررسی در پایگاه miRDB نشاندهنده هدفگیری ژن APRIL توسط hsa-miR-6716-5p و hsa-miR-4306 با بالاترین امتیاز است. نتایج در جدول به شماره 10 قابل مشاهده است.
نحوه گزینش و انتخاب miRNAهای برتر
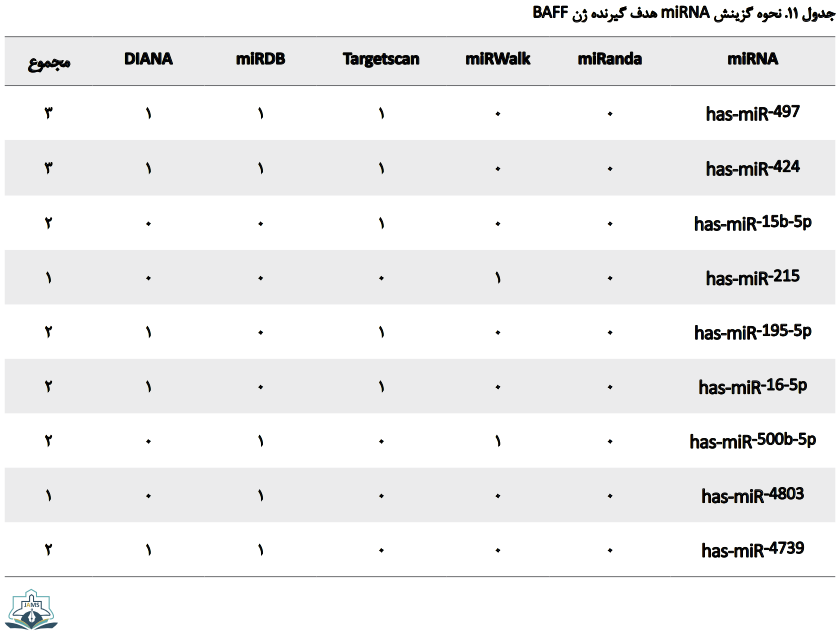
نحوه انتخاب و گزینش miRNA هدف گیرنده ژن BAFF در جدول شماره 11 و miRNA هدف گیرنده ژن APRIL در جدول شماره 12 قابل مشاهده است. بدین ترتیب در جدول شماره 11 و 12 miRNAهای با امتیاز بالاتر و اتصال بهتر با ژنهای هدف، انتخاب و گردآوری شدند. سپس از بین آنها miRNAهایی با بیشترین تکرار و تأیید در پایگاههای مورد مطالعه به عنوان miRNAهای هدف گیرنده این ژنها جهت بررسی در فاز عملی این پژوهش انتخاب شدند.
همچنین miRNAهای منتخب (miR-145-5p ،miR-185 ،miR-424 و miR-497) برای بررسی میزان بیان در افراد مبتلا به سرطان خون و افراد گروه کنترل (افراد سالم) در مطالعات فاز عملی در جدول 13 نشان داده شده است.
بحث
بیوانفورماتیک یک تکنیک مهم برای مدیریت دادههای بیولوژیکی در مقیاس بزرگ است. درواقع مجموعهای از نرمافزارها در بیوتکنولوژی مدرن هست که روشها و نرمافزارهایی را برای درک بهتر اطلاعات بیولوژیکی ارائه میدهد و با استفاده از علوم ریاضیات و آمار به بسیاری از سؤالات زیستپزشکی پاسخ میدهد و از این طریق میتواند به تفسیر و تحلیل دادههای زیستی بپردازد [17].
مطالعات پیشین حاکی از دخالت mi-RNAها تقریباً در تمامی مکانیسمهای فیزیولوژیکی و آسیبشناختی هستند. با توجه به توزیع گسترده بیان miRNAsها در انسان، برخی از آنها به طور کلی و برخی دیگر در بافتها و یا سلولهای خاص بیان میشوند [18]. بنابراین بر اساس موارد ذکرشده miRNAs به عنوان یک شبکه تنظیمی پیچیده در بسیاری از فرایندهای سلولی نقش ایفا میکنند. بدین ترتیب با وجود اثبات بیان نابهجای miRNAs در بسیاری از بیماریها از جمله سرطانها، میتوان از این عوامل مولکولی به عنوان بیومارکرهای بالقوه هم برای تشخیص و هم به عنوان عوامل درمانی سود جست. لوسمی لنفوسیتی مزمن شایعترین لوسمی در انسان است [19].
مطالعات پیشین نشان دادهاند پروفایلهای بیانی miRNAs میتوانند سلولهای B طبیعی را از سلولهای بدخیم CLL تشخیص دهند و با پیشآگهی، پیشرفت و مقاومت دارویی مرتبط باشند [20]. به عنوان مثال روسی و کوی به ترتیب در سالهای 2010 و 2014 اثبات کردند بیمارانی که دارای میزان بیان بالایی از miR-21 و miR-155 هستند، نسبت به بیمارانی با میزان بیان پایینتر این عوامل مولکولی، خطر مرگ بیشتری دارند [22 ،21]. دو پروتئین BAFF و APRIL جزء سوپر خانواده TNF است و از طریق اتصال به گیرندههای سلولی خود در روند بقا، تمایز، آپاپتوز و تکامل طبیعی لنفوسیتهای B ایفای نقش میکند [23]. بنابراین به نظر میرسد با پیش گویی miRNAهای هدف گیرنده ژنهای BAFF و APRIL میتوان در تشخیص و درمان B-CLL گامهای مؤثری برداشت. بدین ترتیب برای اولینبار در مطالعه حاضر با استفاده از نرمافزارهای بیوانفورماتیکی miRanda، TargetScan، miRWalk، DIANA و miRDB به بررسی و پیشگویی miRNها پرداخته شد و درنهایت has-miR-145-5p و has-miR-185-5p به عنوان miRNAهای هدف گیرنده ژن APRIL و همچنین has-miR-424 و has-miR-497 به عنوان miRNAهای هدف گیرنده ژن BAFF معرفی و برای فاز مطالعات عملی آینده منتخب شدند.
از آنجایی که براساس بررسی های انجامشده مطالعه اختصاصی و مشخصی در رابطه با نقش has-miR-145-5p و has-miR-185-5p به عنوان miRNAهای هدف گیرنده ژن APRIL و همچنین has-miR-424 وhas-miR-497 به عنوان miRNهای هدف گیرنده ژن BAFF در پیشرفت سرطان B-CLL مشاهده نشده است، مطالعه حاضر به عنوان اولین مطالعه، امکان معرفی و بررسی miRNAها و ژنهای جدید در کمک به تشخیص مبتلایان به این سرطان را فراهم میآورد.
نتیجهگیری
در این مطالعه برای اولینبار has-miR-145-5p و has-miR-185-5p بر اساس احتمال اتصال مؤثرتر با ژن APRIL با توجه به امتیازدهی در نرمافزارهای بیوانفورماتیکی انتخاب و معرفی شدند. در این راستا از مجموع پنج پایگاه داده مورداستفاده در این مطالعه، اتصال has-miR-145-5p به پروتئین APRIL در پایگاههای TargetScan و miRWalk و همچنین اتصال has-miR-185-5p به این پروتئین در پایگاههای miRanda و miRDB با بالاترین امتیاز تأیید شد. به علاوه has-miR-424 و has-miR-497 به عنوان miRNAهای هدف گیرنده ژن BAFF با پیشگویی و تأیید در سه نرمافزار بیوانفورماتیکی TargetScan، DIANA و miRDB از مجموع پنج پایگاه داده مذکور با بالاترین امتیاز انتخاب شدند. با توجه به نقش مهم ژنهای APRIL و BAFF در روند طبیعی مرگ سلولی و تکامل سلولهای B به نظر میرسد میتوان از این mi-RNAهای پیشگوییشده به عنوان بیومارکرهای مولکولی تشخیصی در جهت شناسایی بیماران B-CLL سود جست.
ملاحظات اخلاقی
پیروی از اصول اخلاق پژوهش
این مطالعه در کمیته اخلاق دانشگاه علوم پزشکی اراک با کد IR.ARAKMU.REC.1395.418تصویب شده است.
حامی مالی
پژوهش حاضر دربردارنده بخشی از طرح تحقیقاتی در دانشگاه علوم پزشکی اراک بوده که هزینه آن به وسیله معاونت تحقیقات و فناوری این دانشگاه تأمین شده است.
مشارکت نویسندگان
مفهومسازی: دکتر قاسم مسیبی و دکتر مهدیه موندنیزاده؛ تحقیق و بررسی: نیلوفر مرادی، مرضیه امینی، دکتر موندنیزاده؛ ویراستاری و نهاییسازی: دکتر موندنیزاده، نیلوفر مرادی؛ اعتبارسنجی: دکتر بهزاد خوانسارینژاد؛ نظارت: دکتر قاسم مسیبی.
تعارض منافع
نویسندگان تصریح میکنند هیچگونه تضاد منافعی در خصوص پژوهش حاضر نداشتند.
تشکر و قدردانی
نویسندگان کمال قدردانی و امتنان را از این معاونت دارند. همچنین نویسندگان مراتب قدردانی خود را از همکاران محترم در آزمایشگاه میکروبیولوژی مولکولی و ویروسشناسی دانشگاه علوم پزشکی اراک و انستیتوپاستور اعلام میدارند.
بیوانفورماتیک یک تکنیک مهم برای مدیریت دادههای بیولوژیکی در مقیاس بزرگ است. درواقع مجموعهای از نرمافزارها در بیوتکنولوژی مدرن هست که روشها و نرمافزارهایی را برای درک بهتر اطلاعات بیولوژیکی ارائه میدهد و با استفاده از علوم ریاضیات و آمار به بسیاری از سؤالات زیستپزشکی پاسخ میدهد و از این طریق میتواند به تفسیر و تحلیل دادههای زیستی بپردازد [17].
مطالعات پیشین حاکی از دخالت mi-RNAها تقریباً در تمامی مکانیسمهای فیزیولوژیکی و آسیبشناختی هستند. با توجه به توزیع گسترده بیان miRNAsها در انسان، برخی از آنها به طور کلی و برخی دیگر در بافتها و یا سلولهای خاص بیان میشوند [18]. بنابراین بر اساس موارد ذکرشده miRNAs به عنوان یک شبکه تنظیمی پیچیده در بسیاری از فرایندهای سلولی نقش ایفا میکنند. بدین ترتیب با وجود اثبات بیان نابهجای miRNAs در بسیاری از بیماریها از جمله سرطانها، میتوان از این عوامل مولکولی به عنوان بیومارکرهای بالقوه هم برای تشخیص و هم به عنوان عوامل درمانی سود جست. لوسمی لنفوسیتی مزمن شایعترین لوسمی در انسان است [19].
مطالعات پیشین نشان دادهاند پروفایلهای بیانی miRNAs میتوانند سلولهای B طبیعی را از سلولهای بدخیم CLL تشخیص دهند و با پیشآگهی، پیشرفت و مقاومت دارویی مرتبط باشند [20]. به عنوان مثال روسی و کوی به ترتیب در سالهای 2010 و 2014 اثبات کردند بیمارانی که دارای میزان بیان بالایی از miR-21 و miR-155 هستند، نسبت به بیمارانی با میزان بیان پایینتر این عوامل مولکولی، خطر مرگ بیشتری دارند [22 ،21]. دو پروتئین BAFF و APRIL جزء سوپر خانواده TNF است و از طریق اتصال به گیرندههای سلولی خود در روند بقا، تمایز، آپاپتوز و تکامل طبیعی لنفوسیتهای B ایفای نقش میکند [23]. بنابراین به نظر میرسد با پیش گویی miRNAهای هدف گیرنده ژنهای BAFF و APRIL میتوان در تشخیص و درمان B-CLL گامهای مؤثری برداشت. بدین ترتیب برای اولینبار در مطالعه حاضر با استفاده از نرمافزارهای بیوانفورماتیکی miRanda، TargetScan، miRWalk، DIANA و miRDB به بررسی و پیشگویی miRNها پرداخته شد و درنهایت has-miR-145-5p و has-miR-185-5p به عنوان miRNAهای هدف گیرنده ژن APRIL و همچنین has-miR-424 و has-miR-497 به عنوان miRNAهای هدف گیرنده ژن BAFF معرفی و برای فاز مطالعات عملی آینده منتخب شدند.
از آنجایی که براساس بررسی های انجامشده مطالعه اختصاصی و مشخصی در رابطه با نقش has-miR-145-5p و has-miR-185-5p به عنوان miRNAهای هدف گیرنده ژن APRIL و همچنین has-miR-424 وhas-miR-497 به عنوان miRNهای هدف گیرنده ژن BAFF در پیشرفت سرطان B-CLL مشاهده نشده است، مطالعه حاضر به عنوان اولین مطالعه، امکان معرفی و بررسی miRNAها و ژنهای جدید در کمک به تشخیص مبتلایان به این سرطان را فراهم میآورد.
نتیجهگیری
در این مطالعه برای اولینبار has-miR-145-5p و has-miR-185-5p بر اساس احتمال اتصال مؤثرتر با ژن APRIL با توجه به امتیازدهی در نرمافزارهای بیوانفورماتیکی انتخاب و معرفی شدند. در این راستا از مجموع پنج پایگاه داده مورداستفاده در این مطالعه، اتصال has-miR-145-5p به پروتئین APRIL در پایگاههای TargetScan و miRWalk و همچنین اتصال has-miR-185-5p به این پروتئین در پایگاههای miRanda و miRDB با بالاترین امتیاز تأیید شد. به علاوه has-miR-424 و has-miR-497 به عنوان miRNAهای هدف گیرنده ژن BAFF با پیشگویی و تأیید در سه نرمافزار بیوانفورماتیکی TargetScan، DIANA و miRDB از مجموع پنج پایگاه داده مذکور با بالاترین امتیاز انتخاب شدند. با توجه به نقش مهم ژنهای APRIL و BAFF در روند طبیعی مرگ سلولی و تکامل سلولهای B به نظر میرسد میتوان از این mi-RNAهای پیشگوییشده به عنوان بیومارکرهای مولکولی تشخیصی در جهت شناسایی بیماران B-CLL سود جست.
ملاحظات اخلاقی
پیروی از اصول اخلاق پژوهش
این مطالعه در کمیته اخلاق دانشگاه علوم پزشکی اراک با کد IR.ARAKMU.REC.1395.418تصویب شده است.
حامی مالی
پژوهش حاضر دربردارنده بخشی از طرح تحقیقاتی در دانشگاه علوم پزشکی اراک بوده که هزینه آن به وسیله معاونت تحقیقات و فناوری این دانشگاه تأمین شده است.
مشارکت نویسندگان
مفهومسازی: دکتر قاسم مسیبی و دکتر مهدیه موندنیزاده؛ تحقیق و بررسی: نیلوفر مرادی، مرضیه امینی، دکتر موندنیزاده؛ ویراستاری و نهاییسازی: دکتر موندنیزاده، نیلوفر مرادی؛ اعتبارسنجی: دکتر بهزاد خوانسارینژاد؛ نظارت: دکتر قاسم مسیبی.
تعارض منافع
نویسندگان تصریح میکنند هیچگونه تضاد منافعی در خصوص پژوهش حاضر نداشتند.
تشکر و قدردانی
نویسندگان کمال قدردانی و امتنان را از این معاونت دارند. همچنین نویسندگان مراتب قدردانی خود را از همکاران محترم در آزمایشگاه میکروبیولوژی مولکولی و ویروسشناسی دانشگاه علوم پزشکی اراک و انستیتوپاستور اعلام میدارند.
ارسال پیام به نویسنده مسئول
| بازنشر اطلاعات | |
 |
این مقاله تحت شرایط Creative Commons Attribution-NonCommercial 4.0 International License قابل بازنشر است. |