دوره 24، شماره 1 - ( فروردین و اردیبهشت 1400 )
جلد 24 شماره 1 صفحات 61-50 |
برگشت به فهرست نسخه ها
Download citation:
BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:



BibTeX | RIS | EndNote | Medlars | ProCite | Reference Manager | RefWorks
Send citation to:
Pouryari Biyachal P, Ranji N, Nazemi A. Investigating GJB2 Mutation in 31 Individuals With Non-syndromic Hearing Loss. J Arak Uni Med Sci 2021; 24 (1) :50-61
URL: http://jams.arakmu.ac.ir/article-1-6383-fa.html
URL: http://jams.arakmu.ac.ir/article-1-6383-fa.html
پوریایی بیاچال پدرام، رنجی نجمه، ناظمی علی. بررسی جهشهای ژن GJB2 در 31 فرد ناشنوای غیرسندرمی در استان گیلان. مجله دانشگاه علوم پزشكي اراك. 1400; 24 (1) :50-61
1- گروه ژنتیک، دانشکده علوم زیستی، واحد تنکابن، دانشگاه آزاد اسلامی، تنکابن، ایران.
2- گروه زیست شناسی، دانشکده علوم پایه، واحد رشت، دانشگاه آزاد اسلامی ، رشت، ایران. ،n_ranji@iaurasht.ac.ir
2- گروه زیست شناسی، دانشکده علوم پایه، واحد رشت، دانشگاه آزاد اسلامی ، رشت، ایران. ،
متن کامل [PDF 5272 kb]
(1188 دریافت)
| چکیده (HTML) (3301 مشاهده)
متن کامل: (2897 مشاهده)
مقدمه
ناشنوایی یکی از مهمترین نواقص شناختی و حسی عصبی است که سازمان بهداشت جهانی تخمین زده است که حدود 360 میلیون فرد با ناشنوایی خفیف تا شدید در جهان زندگی میکنند. ناشنوایی یک اختلال هتروژن بوده که به واسطه عوامل محیطی یا علل ژنتیکی ایجاد میشود [1].
امروزه بیش از 150 لوکوس ژنی در ایجاد ناشنوایی غیرسندرمی نقش دارند که الگوی وراثت آنها اتوزومی غالب (10 درصد)، اتوزومی مغلوب (80 درصد)، وابسته به X (1 درصد>)، وابسته به Y (1 درصد>) و میتوکندریایی (1 درصد>) است [1]. بیش از 1200 جهش مختلف در ژنوم انسان در بروز ناشنوایی تعیین شده است.
فراوانی این جهشها در جمعیتهای مختلف یکسان نیست. با وجود تنوع ژنتیکی ناشنوایی در جمعیتهای مختلف، لوکوس DFNB1 (در موقعیت کروموزومی 13q11-12) باعث حدود 50 درصد از موارد ایجاد ناشنوایی غیرسندرمی میشود [2].
دو ژن GJB2 و GJB6 در این لوکوس وجود دارند که به طور همزمان بیان میشوند. ژن GJB2 دارای دو اگزون بوده که فقط یکی از دو اگزون قابل ترجمه است [1]. محصول ژن GJB2 یک پروتئین اتصالی به نام کانکسین 26 است [2] که 26 کیلو دالتون (26kDa) وزن داشته و از 226 آمینو اسید تشکیل شده است [1].
کانکسین 26 در گوش داخلی به عنوان یک تنظیمکننده مهم هموستازی یون پتاسیم (K+) عمل میکند. در صورت عدم تبادلات K+، سلولهای شنوایی قادر به واکنش در پاسخ به صدا نخواهند بود [2]. قابل توجه است که همراهی یک جهش در GJB2 و جهش دوم در GJB6، در بعضی موارد ناشنوایی گزارش شده است. محصول ژن GJB6، کانکسین سی (30kDa) با 261 آمینو اسید است که در انتقال یونها و بعضی متابولیتها در محل اتصالات سلولهای شنوایی نقش دارد [1].
جهش در ژن GJB2، به عنوان مهمترین علت ناشنوایی ارثی در دنیا شناخته شده است؛ بنابراین بررسی جهشهای این ژن یکی از مهمترین روشهای غربالگری جهت تشخیص ناشنوایی در دنیا محسوب میشود [3]. جهشهای از نوع حذف که منجر به تولید پروتئین کوتاهشده و به عبارتی سنتز ناقص پروتئین GJB2 میشوند [4]، بسته به تعداد دومین حذفشده پروتئین، باعث فقدان همه یا بخشی از عملکردهای پروتئین میشوند.
شایعترین جهش در ژن GJB2، جهش 35delG است؛ در اثر این جهش، یک نوع تغییر قالب خواندن در اثر حذف یک گوانوزین (G) در نوکلئوتید 35 ژن رخ داده و به ختم زودرس (TGA) منجر شده و درنتیجه یک پروتئین کوتاهشده از کانکسین 26 تولید میشود [5]، درحالیکه جهشهای دیگری که منجر به ختم زودرس رونویسی و تغییر قالب خواندن نمیشوند (ازجمله جابهجاییها)، بسته به موقعیت جهش ممکن است فقط تا حدی روی عملکرد پروتئین تأثیرگذار باشند [4].
بر اساس مطالعات قبلی به نظر میرسد جهش c.35delG (p.G12VfsX2) از یک جد باستانی در اروپا (شرق مرکزی یا مدیترانه ) به واسطه مهاجرت در دیگر نقاط دنیا منتشر شده باشد [6]. جهش 35delG شایعترین جهش GJB2 در جمعیتهای قفقازی با شیوع 85 درصدی بوده که باعث تولید پروتئین کوتاهشده غیرعملکردی میشود.
میانگین فراوانی ناقلین این جهش در اروپا یک در 51 تولد زنده بود که با فراوانی آن در آمریکای شمالی و استرالیا تفاوت قابل ملاحظهای را نشان میدهد، درحالیکه فراوانی آللی جهش 35delG در ژن GJB2 به عنوان شایعترین جهش در مناطق جغرافیایی مختلف ایران، بین 5 درصد تا 22 درصد گزارش شده است [4]. هدف از این مطالعه، بررسی جهشهای موجود در ژن GJB2 در 31 فرد ناشنوای غیرسندرمی در استان گیلان بود که به روش PCR تعیین توالی ارزیابی شد.
امروزه بیش از 150 لوکوس ژنی در ایجاد ناشنوایی غیرسندرمی نقش دارند که الگوی وراثت آنها اتوزومی غالب (10 درصد)، اتوزومی مغلوب (80 درصد)، وابسته به X (1 درصد>)، وابسته به Y (1 درصد>) و میتوکندریایی (1 درصد>) است [1]. بیش از 1200 جهش مختلف در ژنوم انسان در بروز ناشنوایی تعیین شده است.
فراوانی این جهشها در جمعیتهای مختلف یکسان نیست. با وجود تنوع ژنتیکی ناشنوایی در جمعیتهای مختلف، لوکوس DFNB1 (در موقعیت کروموزومی 13q11-12) باعث حدود 50 درصد از موارد ایجاد ناشنوایی غیرسندرمی میشود [2].
دو ژن GJB2 و GJB6 در این لوکوس وجود دارند که به طور همزمان بیان میشوند. ژن GJB2 دارای دو اگزون بوده که فقط یکی از دو اگزون قابل ترجمه است [1]. محصول ژن GJB2 یک پروتئین اتصالی به نام کانکسین 26 است [2] که 26 کیلو دالتون (26kDa) وزن داشته و از 226 آمینو اسید تشکیل شده است [1].
کانکسین 26 در گوش داخلی به عنوان یک تنظیمکننده مهم هموستازی یون پتاسیم (K+) عمل میکند. در صورت عدم تبادلات K+، سلولهای شنوایی قادر به واکنش در پاسخ به صدا نخواهند بود [2]. قابل توجه است که همراهی یک جهش در GJB2 و جهش دوم در GJB6، در بعضی موارد ناشنوایی گزارش شده است. محصول ژن GJB6، کانکسین سی (30kDa) با 261 آمینو اسید است که در انتقال یونها و بعضی متابولیتها در محل اتصالات سلولهای شنوایی نقش دارد [1].
جهش در ژن GJB2، به عنوان مهمترین علت ناشنوایی ارثی در دنیا شناخته شده است؛ بنابراین بررسی جهشهای این ژن یکی از مهمترین روشهای غربالگری جهت تشخیص ناشنوایی در دنیا محسوب میشود [3]. جهشهای از نوع حذف که منجر به تولید پروتئین کوتاهشده و به عبارتی سنتز ناقص پروتئین GJB2 میشوند [4]، بسته به تعداد دومین حذفشده پروتئین، باعث فقدان همه یا بخشی از عملکردهای پروتئین میشوند.
شایعترین جهش در ژن GJB2، جهش 35delG است؛ در اثر این جهش، یک نوع تغییر قالب خواندن در اثر حذف یک گوانوزین (G) در نوکلئوتید 35 ژن رخ داده و به ختم زودرس (TGA) منجر شده و درنتیجه یک پروتئین کوتاهشده از کانکسین 26 تولید میشود [5]، درحالیکه جهشهای دیگری که منجر به ختم زودرس رونویسی و تغییر قالب خواندن نمیشوند (ازجمله جابهجاییها)، بسته به موقعیت جهش ممکن است فقط تا حدی روی عملکرد پروتئین تأثیرگذار باشند [4].
بر اساس مطالعات قبلی به نظر میرسد جهش c.35delG (p.G12VfsX2) از یک جد باستانی در اروپا (شرق مرکزی یا مدیترانه ) به واسطه مهاجرت در دیگر نقاط دنیا منتشر شده باشد [6]. جهش 35delG شایعترین جهش GJB2 در جمعیتهای قفقازی با شیوع 85 درصدی بوده که باعث تولید پروتئین کوتاهشده غیرعملکردی میشود.
میانگین فراوانی ناقلین این جهش در اروپا یک در 51 تولد زنده بود که با فراوانی آن در آمریکای شمالی و استرالیا تفاوت قابل ملاحظهای را نشان میدهد، درحالیکه فراوانی آللی جهش 35delG در ژن GJB2 به عنوان شایعترین جهش در مناطق جغرافیایی مختلف ایران، بین 5 درصد تا 22 درصد گزارش شده است [4]. هدف از این مطالعه، بررسی جهشهای موجود در ژن GJB2 در 31 فرد ناشنوای غیرسندرمی در استان گیلان بود که به روش PCR تعیین توالی ارزیابی شد.
مواد و روشها
بیماران
در این مطالعه توصیفی مقطعی، در مجموع 85 فرد ناشنوا غیرخویشاوند از 85 خانواده مراجعهکننده به اداره کل بهزیستی استان گیلان و اداره بهزیستی شهرستان رشت، کانون ناشنوایی رشت و کانون ناشنوایی سکوت سفید (در بندرانزلی) شناسایی شدند.
اطلاعات بیماران با همکاری این مراکز و با توجه به اطلاعات مندرج توسط پزشک متخصص در پرونده هر فرد جمعآوری شد. در این بین، 31 فرد مبتلا به ناشنوایی غیرسندرمی با ناشنوایی دوطرفه، ناشنوایی از بدو تولد و با داشتن یک یا چند خویشاوند ناشنوا (با تأیید پزشک معالج موجود در پرونده بیمار)، با محدوده سنی هفده تا پنجاه سال انتخاب و خونگیری از آنها انجام شد.
سپس نمونههای خونی از 31 فرد ناشنوای غیرسندرومی غیرخویشاوند در لولههای حاوی EDTA نیم مولار قرار گرفته و به آزمایشگاه انتقال داده شدند و تا زمان استفاده در فریزر منفی 20 در حالت انجماد نگهداری شدند.
تخلیص DNA ژنومی
از نمونههای خونی 31 فرد ناشنوای غیرخویشاوند و یک فرد سالم با استفاده از کیت WizPrepTM gDNA Mini Kit (شرکت Wizbiosolutions، کره جنوبی) تخلیص DNA صورت گرفت. جهت اطمینان از تخلیص صحیح و سالم بودن DNA هر فرد، نمونهها در ژل آگارز 1/5 درصد با ولتاژ نود ولت به مدت 45 دقیقه الکتروفورز شد.
تکثیر ژن GJB2 به روش PCR و تعیین توالی آن
جهت تکثیر ژن GJB2 از کیت Pfu master mix (شرکتPhiloKorea، کره جنوبی) در حجم نهایی µl25 استفاده شد. به این منظور با افزودن DNA ژنومی، جفت پرایمر (pM 20) و آب استریل به مسترمیکس (حاوی آنزیم، کوفاکتور و بافر مخصوص) اجزای واکنش PCR مخلوط شد.
بعد از طراحی جفت پرایمر، سنتز پرایمرها (جدول شماره 1) توسط شرکت Bioneer صورت گرفت. واکنش PCR با استفاده از ترموسایکلر Analaytik jena (آلمان) طبق برنامه زیر انجام شد:
یک مرحله واسرشت شدن ابتدایی در C °95 به مدت سه دقیقه، 30 سیکل در دمای C °95 به مدت سی ثانیه، °C 67 به مدت سی ثانیه، °C 72 به مدت یک دقیقه و یک مرحله گسترش نهایی در دمای °C 72 به مدت پنج دقیقه. توالی پرایمرها در جدول شماره 1 ذکر شده است.
.jpg)
بعد از اطمینان از تک باند بودن محصولات PCR با طول 885 جفت باز، نمونهها با حفظ زنجیره سرمایی به شرکت BGI group (چین) ارسال و تعیین توالی با پرایمر برگشتی صورت گرفت. به دلیل اینکه در انتهای'5 ژن، جهش حذفی رخ داده بود و توالی خوانده شده با پرایمر مستقیم با طول کمی خوانده میشد، درنتیجه همه توالیها با پرایمر برگشتی (از انتهای'3) خوانده شد و جهت تأیید توالی برای بار دوم با پرایمر برگشتی تعیین توالی شد.
نتایج حاصل از تعیین توالی به کمک نرمافزار CLC main workbench v3.5 و نرمافزار برخط «بلاست» از نظر وجود جهش در افراد ناشنوا در مقایسه با ژن رفرنس موجود در سایت NCBI مطالعه و بررسی شد.
در این مطالعه توصیفی مقطعی، در مجموع 85 فرد ناشنوا غیرخویشاوند از 85 خانواده مراجعهکننده به اداره کل بهزیستی استان گیلان و اداره بهزیستی شهرستان رشت، کانون ناشنوایی رشت و کانون ناشنوایی سکوت سفید (در بندرانزلی) شناسایی شدند.
اطلاعات بیماران با همکاری این مراکز و با توجه به اطلاعات مندرج توسط پزشک متخصص در پرونده هر فرد جمعآوری شد. در این بین، 31 فرد مبتلا به ناشنوایی غیرسندرمی با ناشنوایی دوطرفه، ناشنوایی از بدو تولد و با داشتن یک یا چند خویشاوند ناشنوا (با تأیید پزشک معالج موجود در پرونده بیمار)، با محدوده سنی هفده تا پنجاه سال انتخاب و خونگیری از آنها انجام شد.
سپس نمونههای خونی از 31 فرد ناشنوای غیرسندرومی غیرخویشاوند در لولههای حاوی EDTA نیم مولار قرار گرفته و به آزمایشگاه انتقال داده شدند و تا زمان استفاده در فریزر منفی 20 در حالت انجماد نگهداری شدند.
تخلیص DNA ژنومی
از نمونههای خونی 31 فرد ناشنوای غیرخویشاوند و یک فرد سالم با استفاده از کیت WizPrepTM gDNA Mini Kit (شرکت Wizbiosolutions، کره جنوبی) تخلیص DNA صورت گرفت. جهت اطمینان از تخلیص صحیح و سالم بودن DNA هر فرد، نمونهها در ژل آگارز 1/5 درصد با ولتاژ نود ولت به مدت 45 دقیقه الکتروفورز شد.
تکثیر ژن GJB2 به روش PCR و تعیین توالی آن
جهت تکثیر ژن GJB2 از کیت Pfu master mix (شرکتPhiloKorea، کره جنوبی) در حجم نهایی µl25 استفاده شد. به این منظور با افزودن DNA ژنومی، جفت پرایمر (pM 20) و آب استریل به مسترمیکس (حاوی آنزیم، کوفاکتور و بافر مخصوص) اجزای واکنش PCR مخلوط شد.
بعد از طراحی جفت پرایمر، سنتز پرایمرها (جدول شماره 1) توسط شرکت Bioneer صورت گرفت. واکنش PCR با استفاده از ترموسایکلر Analaytik jena (آلمان) طبق برنامه زیر انجام شد:
یک مرحله واسرشت شدن ابتدایی در C °95 به مدت سه دقیقه، 30 سیکل در دمای C °95 به مدت سی ثانیه، °C 67 به مدت سی ثانیه، °C 72 به مدت یک دقیقه و یک مرحله گسترش نهایی در دمای °C 72 به مدت پنج دقیقه. توالی پرایمرها در جدول شماره 1 ذکر شده است.
.jpg)
بعد از اطمینان از تک باند بودن محصولات PCR با طول 885 جفت باز، نمونهها با حفظ زنجیره سرمایی به شرکت BGI group (چین) ارسال و تعیین توالی با پرایمر برگشتی صورت گرفت. به دلیل اینکه در انتهای'5 ژن، جهش حذفی رخ داده بود و توالی خوانده شده با پرایمر مستقیم با طول کمی خوانده میشد، درنتیجه همه توالیها با پرایمر برگشتی (از انتهای'3) خوانده شد و جهت تأیید توالی برای بار دوم با پرایمر برگشتی تعیین توالی شد.
نتایج حاصل از تعیین توالی به کمک نرمافزار CLC main workbench v3.5 و نرمافزار برخط «بلاست» از نظر وجود جهش در افراد ناشنوا در مقایسه با ژن رفرنس موجود در سایت NCBI مطالعه و بررسی شد.
یافتهها
در این مطالعه، 31 فرد ناشنوای غیرسندرمی از بین 85 فرد ناشنوا از استان گیلان شرکت داده شدند که 22 نفر زن (71 درصد~) و نُه نفر مرد (29 درصد~) بودند. میانگین سنی ناشنوایان در این مطالعه 33/33 سال بود. مطابق تصویر شماره 1 بعد از اطمینان از تک باند بودن محصولات PCR، نمونهها تعیین توالی شد.
.jpg)
در این مطالعه (جدول شماره 2)، در شانزده بیمار جهش 35delG شناسایی شد که در این بین، چهارده بیمار به صورت هموزیگوت (تصویر شماره 2) و دو بیمار به صورت هتروزیگوت (تصویر شماره 3) این جهش را داشتند. در یک بیمار نیز جهش V153I به صورت هتروزیگوت مشاهده شد (تصویر شماره 4).
.jpg)
.jpg)
.jpg)
.jpg)
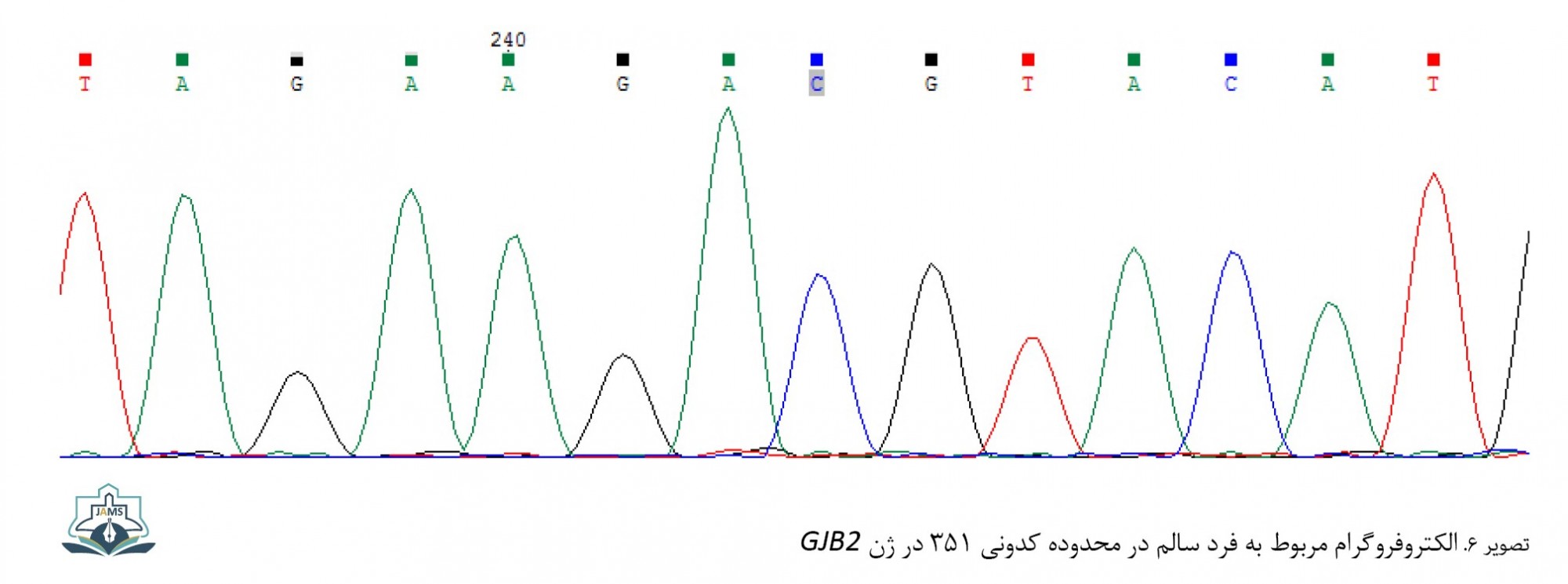
در یکی از دو بیمار با جهش هتروزیگوت 35delG، واریانت بیماریزای G200R نیز به صورت هتروزیگوت مشاهده شد (35delG/G200R) (تصویر شماره 5). توالی ژن GJB2 در فرد سالم در سه ناحیه جهشیافته در این مطالعه به ترتیب در سه تصاویر6، 7 و 8 نشان داده شده است.
.jpg)


ناشنوایی غیرسندرمی در اثر اختلال در ژنهای مختلفی ایجاد میشود. فراوانی جهشهای بیماریزای مؤثر در ناشنوایی در ژنهای مختلف در هر جمعیت بسته به مواردی نظیر الگوی وراثت، میزان جهشپذیری هر ژن و جریان ژنی متغیر است.
در مطالعه حاضر در استان گیلان با بررسی 31 فرد ناشنوای غیرسندرمی غیرخویشاوند، نزدیک به 52 درصد افراد دارای جهش در ژن GJB2 بودند. در این افراد سه جهش و یک واریانت بیماریزای ژنی شناسایی شد و جهش 35delG با بیشترین فراوانی در این مطالعه گزارش شد.
در مطالعه هاشمی و همکاران، جهش در ژن GJB2 در افراد ناشنوا در شهر شیراز با فراوانی 30 درصدی گزارش شد [4]. شیوع جهش در این ژن در مطالعه بهرامی و همکاران در استان کردستان 20 درصد [7]، در مطالعه بنیادی و همکاران در جمعیت آذریهای ترک ایران 28% [8] و در مطالعه ابطحی و همکاران در اصفهان حدود 24 درصد [1] و در مطالعه محمودی و همکاران در ایلام 4 درصد [9] و در مطالعه آهنگری و همکاران در هرمزگان حدود 6 درصد گزارش شد [10]. در مطالعه جهانگیری و همکاران در تبریز از بین پنجاه فرد ناشنوای مادرزادی 39 درصد موارد دارای جهش در این ژن بودند [11].
در مطالعه کالای و همکاران در ترکیه شیوع جهش در این ژن 25 درصد [12] و در مطالعه مینارلیک و همکاران در پاکستان شیوع آن 1/6 درصد بود [13]. در مقایسه با مطالعات یادشده در چندین استان و دو کشور همسایه به نظر میرسد شیوع جهش در GJB2 در استان گیلان نرخ بالایی داشته باشد و توصیه میشود در غربالگریهای ژنتیکی در خط اول آزمایشات در افراد در خطر در این استان قرار گیرد.
در مطالعه هاشمی و همکاران، فراوانی آللی جهش 35delG حدود 7 درصد و جهش V153I حدود 6 درصد به عنوان شایعترین جهشها گزارش شدند. در این مطالعه، هر دو جهش هم به صورت هتروزیگوت و هم هموزیگوت در ناشنوایان شناسایی شد [4]. در مطالعه ابطحی و همکاران در اصفهان جهش 35delG با فراوانی 6/12 درصد (به عنوان شایعترین جهش) و جهش V153I با فراوانی آللی 2/04 درصد بین 46 ناشنوا گزارش شد [1].
در مطالعه آزادگان و دهکردی، جهش 35delG با فراوانی آللی 13/33 درصد بین 45 ناشنوای کُرد مشاهده شد [14]. در مطالعه ندافنیا و همکاران روی 131 فرد ناشنوا در قم، شیوع بالای این جهش (61/9 درصد) گزارش شد [15]. در مطالعه جهانگیری و همکاران در تبریز از بین 50 فرد ناشنوای مادرزادی همه 39 درصد مورد دارای جهش در ژن GJB2 از نوع 35delG بودند و جهش دیگری در این افراد مشاهده نشد [11].
در مطالعه سپهوند و همکاران روی 53 فرد ناشنوا در لرستان حدود 9/4 درصد موارد دارای جهش 35delG و حدود 2/8 درصد افراد دارای جهش V153I بودند [16].
مطالعه حاضر از نظر جهش 35delG بین 31 فرد ناشنوای غیرخویشاوند (48/38 درصد) ظاهراً با مطالعات جهانگیری و همکاران در تبریز و ندافنیا و همکاران در قم از نظر فراوانی بالا شباهت بیشتری دارد، درحالیکه در بعضی از مناطق ایران، ازجمله کردستان، آذربایجان، اصفهان و کشورهای همسایه (ترکیه و پاکستان) با وجود شایع بودن این جهش در این مناطق، ظاهراً شیوع پایینتری مشاهده شده است.
به خاطر نرخ بالای جهش در این ژن در شهرهای مورد مطالعه (رشت و انزلی) در استان گیلان میتوان انتظار داشت که جهش در این ژن در بروز ناشنوایی در این شهرها دارای اهمیت بسزایی باشد. همچنین جهش V153I در بعضی مطالعات یادشده به عنوان جهش شایع گزارش شده، درحالیکه در مطالعه حاضر تنها در یک آلل گزارش شد و احتمالاً در استان گیلان فراوانی بالایی در بین ناشنوایان نداشته باشد.
از سوی دیگر، در یک مطالعه دوازده ساله توسط بزاز زادگان و همکاران روی 2322 ناشنوا از سراسر ایران، شیوع بالای جهش 35delG در اردبیل (75 درصد)، آذربایجان (78 درصد)، اصفهان (76 درصد)، شیراز (81 درصد) و گیلان (84 درصد) در سال 2012 گزارش شد. در بعضی از استانها احتمال میرود به دلیل تعداد بسیار کم نمونه این جهش یافت نشد. با این حال در سیستان و بلوچستان با وجود مطالعه روی 21 ناشنوا، این جهش در هیچیک از بیماران وجود نداشت. در کل، شیوع این جهش در ایران قابل ملاحظه (64/7 درصد) است [17].
در مطالعه اییرچی و همکاران فراوانی آللی این جهش 13/4 درصد در بین 56 ناشنوا در ترکیه گزارش شد [18]. قابل ذکر است فراوانی جهش 35delG در مطالعه بزاز زادگان، با فراوانی آن در مطالعات انجامشده در هر استان به تنهایی و با مطالعه حاضر دارای تفاوت مشهودی است. به عنوان نمونه مطالعه ابطحی و همکاران در اصفهان جهش 35delG با فراوانی 6/12 درصد گزارش شد، درحالیکه در مطالعه بزاز زادگان در اصفهان با نرخ 76 درصد مشاهده شد.
در مطالعه حاضر در استان گیلان جهش 35delG با نرخ 48/38 درصد گزارش شد و در مطالعه بزاز زادگان در گیلان با نرخ 84 درصد گزارش شد. عوامل مختلفی میتواند باعث تفاوت این گزارشات شود.
مطالعه حاضر نمونهگیری از ناشنوایان طی یک دوره شش ماهه صورت گرفته، درحالیکه در مطالعه بزاز زادگان طی دوازده سال شناسایی افراد ناشنوا در کل کشور صورت گرفت.
در مطالعه حاضر، نمونهگیری از ناشنوایان از دو شهر رشت و انزلی صورت گرفت، درحالیکه در مطالعه بزاز زادگان محل نمونهگیری در استان گیلان ذکر نشده است. همچنین علاوه بر تفاوت در فراوانی آللی جهش 35delG، جهشهای دیگر گزارششده در دو مطالعه کاملاً متفاوت است. در مطالعه بزاز زادگان و همکاران جهشهای R184P ،E110DfsX4 و D46N و R32C گزارش شد [17]، درحالیکه هیچکدام از این جهشها در افراد ناشنوا در مطالعه حاضر شناسایی نشدند.
همچنین نتایج مطالعه بزاز زادگان و همکاران از نظر زمانی نسبت به مطالعه حاضر قدیمیتر است، بهطوریکه تفاوت دو مطالعه در زمان نمونهگیری طی دو دهه اخیر (2000 تا 2019) در نوع و نرخ جهشهای آن تأثیرگذار بوده و با مقایسه نتایج این دو مطالعه پیشنهاد میشود که برای آگاهی از آمار صحیح جهشها، جهت استفاده در غربالگریهای ژنتیکی ناشنوایی در هر استان این سری از مطالعات هرچند سال یکبار در بین ناشنوایان هر استان تکرار و آمار دقیق جهشها گزارش شود تا شیوع این ناهنجاری بین خانوادههای ایرانی کاهش یابد (۱۷).
مطالعه هاشمی و همکاران در شیراز [4] و ابطحی و همکاران در اصفهان [1] نیز نتایج بسیار متفاوتی با مطالعه بزاز زادگان در فراوانی آللی 35delG نشان میدهد که به نظر میرسد به خاطر تفاوت در زمان یا بازه زمانی مورد مطالعه باشد و از این لحاظ مطالعه حاضر نرخ بالای این جهش را همچنان بعد از دو دهه در گیلان نشان میدهد که به پیگیریهای بیشتر جهت کاهش نرخ بیماری در استان نیاز است. در مطالعه یو و همکاران روی 1852 فرد ناشنوا در شانگهای چین واریانت G200R به عنوان یک واریانت بیماریزا معرفی شد [19]. در مطالعه هاشمزاده چالشتری و همکاران در تهران و تبریز روی افراد ناشنوا، واریانت بیماریزای G200R در مبتلایان تهرانی گزارش شد [20].
فراوانی این واریانت بیماریزا بین 152 خانواده ناشنوای مراکشی حدود 0/66 درصد گزارش شد [21]. در مطالعه مروری مهدیه و همکاران مشخص شد که در مطالعات قبلی در ایران این واریانت بیماریزا تنها در دو آللی با فراوانی 0/05 درصد گزارش شده بود. این واریانت بیماریزا در دومین چهارم (TM4) قرار داشته [22] و اهمیت آن در بیماریزایی هنوز ناشناخته است.
با اینکه مهدیه و همکاران، فراوانی کمی از این واریانت را در بیش از ده مطالعه در ایران یافتهاند؛ قابل توجه است که فقط در استان گیلان این واریانت بیماریزا با فراوانی در یک آلل مشاهده شد. هرچند برای بررسی اهمیت آن لازم است جمعیت بیشتری از ناشنوایان و هم افراد شنوا مورد بررسی قرار گیرند که در مطالعه حاضر امکان انجام آزمایشات به این گسترگی وجود نداشت.
با توجه به اینکه ژنهای مختلفی در بروز ناشنوایی دخیل است از محدودیتهای این مطالعه میتوان به پیچیدگی شجرههای ناشنوایی و دخیل بودن ژنهای مختلف در تفسیر این شجرهها اشاره کرد که به صرف هزینه و زمان زیادی جهت شناسایی عوامل ژنتیکی بروز این ناهنجاری نیاز دارد.
همچنین از دیگر محدودیتهای تحقیق نحوه برقراری ارتباط با ناشنوایان است؛ بنابراین با توجه به حمایتهای بیشائبه کانونهای ناشنوایی رشت و بندرانزلی در این تحقیق توانستیم 31 فرد ناشنوا با سابقه فامیلی را از بین 85 فرد ناشنوا شناسایی و از نظر جهشهای ژن GJB2 بررسی کنیم.
.jpg)
در این مطالعه (جدول شماره 2)، در شانزده بیمار جهش 35delG شناسایی شد که در این بین، چهارده بیمار به صورت هموزیگوت (تصویر شماره 2) و دو بیمار به صورت هتروزیگوت (تصویر شماره 3) این جهش را داشتند. در یک بیمار نیز جهش V153I به صورت هتروزیگوت مشاهده شد (تصویر شماره 4).
.jpg)
.jpg)
.jpg)
.jpg)
در یکی از دو بیمار با جهش هتروزیگوت 35delG، واریانت بیماریزای G200R نیز به صورت هتروزیگوت مشاهده شد (35delG/G200R) (تصویر شماره 5). توالی ژن GJB2 در فرد سالم در سه ناحیه جهشیافته در این مطالعه به ترتیب در سه تصاویر6، 7 و 8 نشان داده شده است.
.jpg)



ناشنوایی غیرسندرمی در اثر اختلال در ژنهای مختلفی ایجاد میشود. فراوانی جهشهای بیماریزای مؤثر در ناشنوایی در ژنهای مختلف در هر جمعیت بسته به مواردی نظیر الگوی وراثت، میزان جهشپذیری هر ژن و جریان ژنی متغیر است.
در مطالعه حاضر در استان گیلان با بررسی 31 فرد ناشنوای غیرسندرمی غیرخویشاوند، نزدیک به 52 درصد افراد دارای جهش در ژن GJB2 بودند. در این افراد سه جهش و یک واریانت بیماریزای ژنی شناسایی شد و جهش 35delG با بیشترین فراوانی در این مطالعه گزارش شد.
در مطالعه هاشمی و همکاران، جهش در ژن GJB2 در افراد ناشنوا در شهر شیراز با فراوانی 30 درصدی گزارش شد [4]. شیوع جهش در این ژن در مطالعه بهرامی و همکاران در استان کردستان 20 درصد [7]، در مطالعه بنیادی و همکاران در جمعیت آذریهای ترک ایران 28% [8] و در مطالعه ابطحی و همکاران در اصفهان حدود 24 درصد [1] و در مطالعه محمودی و همکاران در ایلام 4 درصد [9] و در مطالعه آهنگری و همکاران در هرمزگان حدود 6 درصد گزارش شد [10]. در مطالعه جهانگیری و همکاران در تبریز از بین پنجاه فرد ناشنوای مادرزادی 39 درصد موارد دارای جهش در این ژن بودند [11].
در مطالعه کالای و همکاران در ترکیه شیوع جهش در این ژن 25 درصد [12] و در مطالعه مینارلیک و همکاران در پاکستان شیوع آن 1/6 درصد بود [13]. در مقایسه با مطالعات یادشده در چندین استان و دو کشور همسایه به نظر میرسد شیوع جهش در GJB2 در استان گیلان نرخ بالایی داشته باشد و توصیه میشود در غربالگریهای ژنتیکی در خط اول آزمایشات در افراد در خطر در این استان قرار گیرد.
در مطالعه هاشمی و همکاران، فراوانی آللی جهش 35delG حدود 7 درصد و جهش V153I حدود 6 درصد به عنوان شایعترین جهشها گزارش شدند. در این مطالعه، هر دو جهش هم به صورت هتروزیگوت و هم هموزیگوت در ناشنوایان شناسایی شد [4]. در مطالعه ابطحی و همکاران در اصفهان جهش 35delG با فراوانی 6/12 درصد (به عنوان شایعترین جهش) و جهش V153I با فراوانی آللی 2/04 درصد بین 46 ناشنوا گزارش شد [1].
در مطالعه آزادگان و دهکردی، جهش 35delG با فراوانی آللی 13/33 درصد بین 45 ناشنوای کُرد مشاهده شد [14]. در مطالعه ندافنیا و همکاران روی 131 فرد ناشنوا در قم، شیوع بالای این جهش (61/9 درصد) گزارش شد [15]. در مطالعه جهانگیری و همکاران در تبریز از بین 50 فرد ناشنوای مادرزادی همه 39 درصد مورد دارای جهش در ژن GJB2 از نوع 35delG بودند و جهش دیگری در این افراد مشاهده نشد [11].
در مطالعه سپهوند و همکاران روی 53 فرد ناشنوا در لرستان حدود 9/4 درصد موارد دارای جهش 35delG و حدود 2/8 درصد افراد دارای جهش V153I بودند [16].
مطالعه حاضر از نظر جهش 35delG بین 31 فرد ناشنوای غیرخویشاوند (48/38 درصد) ظاهراً با مطالعات جهانگیری و همکاران در تبریز و ندافنیا و همکاران در قم از نظر فراوانی بالا شباهت بیشتری دارد، درحالیکه در بعضی از مناطق ایران، ازجمله کردستان، آذربایجان، اصفهان و کشورهای همسایه (ترکیه و پاکستان) با وجود شایع بودن این جهش در این مناطق، ظاهراً شیوع پایینتری مشاهده شده است.
به خاطر نرخ بالای جهش در این ژن در شهرهای مورد مطالعه (رشت و انزلی) در استان گیلان میتوان انتظار داشت که جهش در این ژن در بروز ناشنوایی در این شهرها دارای اهمیت بسزایی باشد. همچنین جهش V153I در بعضی مطالعات یادشده به عنوان جهش شایع گزارش شده، درحالیکه در مطالعه حاضر تنها در یک آلل گزارش شد و احتمالاً در استان گیلان فراوانی بالایی در بین ناشنوایان نداشته باشد.
از سوی دیگر، در یک مطالعه دوازده ساله توسط بزاز زادگان و همکاران روی 2322 ناشنوا از سراسر ایران، شیوع بالای جهش 35delG در اردبیل (75 درصد)، آذربایجان (78 درصد)، اصفهان (76 درصد)، شیراز (81 درصد) و گیلان (84 درصد) در سال 2012 گزارش شد. در بعضی از استانها احتمال میرود به دلیل تعداد بسیار کم نمونه این جهش یافت نشد. با این حال در سیستان و بلوچستان با وجود مطالعه روی 21 ناشنوا، این جهش در هیچیک از بیماران وجود نداشت. در کل، شیوع این جهش در ایران قابل ملاحظه (64/7 درصد) است [17].
در مطالعه اییرچی و همکاران فراوانی آللی این جهش 13/4 درصد در بین 56 ناشنوا در ترکیه گزارش شد [18]. قابل ذکر است فراوانی جهش 35delG در مطالعه بزاز زادگان، با فراوانی آن در مطالعات انجامشده در هر استان به تنهایی و با مطالعه حاضر دارای تفاوت مشهودی است. به عنوان نمونه مطالعه ابطحی و همکاران در اصفهان جهش 35delG با فراوانی 6/12 درصد گزارش شد، درحالیکه در مطالعه بزاز زادگان در اصفهان با نرخ 76 درصد مشاهده شد.
در مطالعه حاضر در استان گیلان جهش 35delG با نرخ 48/38 درصد گزارش شد و در مطالعه بزاز زادگان در گیلان با نرخ 84 درصد گزارش شد. عوامل مختلفی میتواند باعث تفاوت این گزارشات شود.
مطالعه حاضر نمونهگیری از ناشنوایان طی یک دوره شش ماهه صورت گرفته، درحالیکه در مطالعه بزاز زادگان طی دوازده سال شناسایی افراد ناشنوا در کل کشور صورت گرفت.
در مطالعه حاضر، نمونهگیری از ناشنوایان از دو شهر رشت و انزلی صورت گرفت، درحالیکه در مطالعه بزاز زادگان محل نمونهگیری در استان گیلان ذکر نشده است. همچنین علاوه بر تفاوت در فراوانی آللی جهش 35delG، جهشهای دیگر گزارششده در دو مطالعه کاملاً متفاوت است. در مطالعه بزاز زادگان و همکاران جهشهای R184P ،E110DfsX4 و D46N و R32C گزارش شد [17]، درحالیکه هیچکدام از این جهشها در افراد ناشنوا در مطالعه حاضر شناسایی نشدند.
همچنین نتایج مطالعه بزاز زادگان و همکاران از نظر زمانی نسبت به مطالعه حاضر قدیمیتر است، بهطوریکه تفاوت دو مطالعه در زمان نمونهگیری طی دو دهه اخیر (2000 تا 2019) در نوع و نرخ جهشهای آن تأثیرگذار بوده و با مقایسه نتایج این دو مطالعه پیشنهاد میشود که برای آگاهی از آمار صحیح جهشها، جهت استفاده در غربالگریهای ژنتیکی ناشنوایی در هر استان این سری از مطالعات هرچند سال یکبار در بین ناشنوایان هر استان تکرار و آمار دقیق جهشها گزارش شود تا شیوع این ناهنجاری بین خانوادههای ایرانی کاهش یابد (۱۷).
مطالعه هاشمی و همکاران در شیراز [4] و ابطحی و همکاران در اصفهان [1] نیز نتایج بسیار متفاوتی با مطالعه بزاز زادگان در فراوانی آللی 35delG نشان میدهد که به نظر میرسد به خاطر تفاوت در زمان یا بازه زمانی مورد مطالعه باشد و از این لحاظ مطالعه حاضر نرخ بالای این جهش را همچنان بعد از دو دهه در گیلان نشان میدهد که به پیگیریهای بیشتر جهت کاهش نرخ بیماری در استان نیاز است. در مطالعه یو و همکاران روی 1852 فرد ناشنوا در شانگهای چین واریانت G200R به عنوان یک واریانت بیماریزا معرفی شد [19]. در مطالعه هاشمزاده چالشتری و همکاران در تهران و تبریز روی افراد ناشنوا، واریانت بیماریزای G200R در مبتلایان تهرانی گزارش شد [20].
فراوانی این واریانت بیماریزا بین 152 خانواده ناشنوای مراکشی حدود 0/66 درصد گزارش شد [21]. در مطالعه مروری مهدیه و همکاران مشخص شد که در مطالعات قبلی در ایران این واریانت بیماریزا تنها در دو آللی با فراوانی 0/05 درصد گزارش شده بود. این واریانت بیماریزا در دومین چهارم (TM4) قرار داشته [22] و اهمیت آن در بیماریزایی هنوز ناشناخته است.
با اینکه مهدیه و همکاران، فراوانی کمی از این واریانت را در بیش از ده مطالعه در ایران یافتهاند؛ قابل توجه است که فقط در استان گیلان این واریانت بیماریزا با فراوانی در یک آلل مشاهده شد. هرچند برای بررسی اهمیت آن لازم است جمعیت بیشتری از ناشنوایان و هم افراد شنوا مورد بررسی قرار گیرند که در مطالعه حاضر امکان انجام آزمایشات به این گسترگی وجود نداشت.
با توجه به اینکه ژنهای مختلفی در بروز ناشنوایی دخیل است از محدودیتهای این مطالعه میتوان به پیچیدگی شجرههای ناشنوایی و دخیل بودن ژنهای مختلف در تفسیر این شجرهها اشاره کرد که به صرف هزینه و زمان زیادی جهت شناسایی عوامل ژنتیکی بروز این ناهنجاری نیاز دارد.
همچنین از دیگر محدودیتهای تحقیق نحوه برقراری ارتباط با ناشنوایان است؛ بنابراین با توجه به حمایتهای بیشائبه کانونهای ناشنوایی رشت و بندرانزلی در این تحقیق توانستیم 31 فرد ناشنوا با سابقه فامیلی را از بین 85 فرد ناشنوا شناسایی و از نظر جهشهای ژن GJB2 بررسی کنیم.
نتیجهگیری
در مطالعه حاضر، حدود 52 درصد از افراد ناشنوا از کانونهای ناشنوایی رشت و بندرانزلی در ژن GJB2 دارای جهش بودند که شایعترین جهش در بین این ناشنوایان جهش 35delG بود. با توجه به شیوع بالای جهش 35delG در استان گیلان، در غربالگریهای ژنتیکی ناشنوایی در گیلان لازم است ژن GJB2 به عنوان مهمترین ژن مؤثر در ناشنوایی غیرسندرمی بررسی شود.
ملاحظات اخلاقی
پیروی از اصول اخلاق پژوهش
این مقاله مورد تایید کمیته اخلاق دانشگاه آزاد اسلامی واحد تنکابن با کد اخلاق پزشکی به شماره IR.IAU.TON.REC.1398.015 قرار گرفته است.
حامی مالی
این مقاله برگرفته از پایاننامه کارشناسی ارشد نویسنده اول در گروه ژنتیک، دانشکده علوم زیستی، واحد تنکابن، دانشگاه آزاد اسلامی، تنکابن است.
مشارکت نویسندگان
مفهومسازی: نجمه رنجی و علی ناظمی؛ روش پژوهش و نمونهگیری: پدرام پوریاری بیاچال؛ تحلیل دادهها، نگارش متن و بازبینی: تمامی نویسندگان.
تعارض منافع
نویسندگان مقاله هیچگونه تعارضی در منافع اعلام نکردند.
تشکر و قدردانی
نویسندگان این مقاله از اداره کل بهزیستی استان گیلان، بهزیستی شهرستان رشت، کانونهای ناشنوایی استان گیلان مستقر در شهرهای رشت و بندرانزلی به دلیل همکاری بیشائبه در شناسایی ناشنوایان و تهیه نمونه خونی از ایشان کمال تشکر و قدردانی را دارند. همچنین از آقای سبحان درویشی به جهت طراحی پرایمر و کمک در آنالیز نتایج کمال تشکر را داریم.
Reference
- Abtahi SHR, Malekzadeh A, Soheilipour S, Salehi M, Taleban R, Rabieian R, et al. Evaluation of GJB2 and GJB6 mutations in patients afflicted with non-syndromic hearing loss. Int J Pediatr 2019; 7(2):9053-60. [DOI:10.22038/IJP.2018.34154.3017]
- Amritkumar P, Jeffrey JM, Chandru J, Kalaimathi M, Ramakrishnan R, Karthikeyen NP, et al. Role of DFNB1 mutations in hereditary hearing loss among assortative mating hearing impaired families from South India. BMC Med Genet. 2018; 19:105. [DOI:10.1186/s12881-018-0609-6] [PMID] [PMCID]
- Abe S, Nishio SY, Yokota Y, Moteki H, Kumakawa K, Usami SI. Diagnostic pitfalls for GJB2-related hearing loss: A novel deletion detected by Array-CGH analysis in a Japanese patient with congenital profound hearing loss. Clin Case Rep. 2018; 6(11):2111-6. [DOI:10.1002/ccr3.1800] [PMID] [PMCID]
- Hashemi SB, Ashraf MJ, Saboori M, Azarpira N, Darai M. Prevalence of GJB2 (CX26) gene mutations in South Iranian patients with autosomal recessive nonsyndromic sensorineural hearing loss. Mol Biol Rep. 2012; 39(12):10481-7. [DOI:10.1007/s11033-012-1929-9] [PMID]
- Missoum A. The role of gene GJB2 and connexin 26 in hearing impairment. Ukr Biochem J. 2018; 90(6):5-11. [DOI:10.15407/ubj90.06.005]
- Zytsar MV, Barashkov NA, Bady-Khoo MS, Shubina-Olejnik OA, Danilenko NG, Bondar AA, et al. Updated carrier rates for c.35delG (GJB2) associated with hearing loss in Russia and common c.35delG haplotypes in Siberia. BMC Med Genet. 2018; 19:138. [DOI:10.1186/s12881-018-0650-5] [PMID] [PMCID]
- Bahrami T, Jalilian N, Karbasi G, Noori-Daloii MR. Specific distribution of GJB2 mutations in Kurdistan province of Iran; Report of a relatively isolated population. J Sci Islam Repub Iran. 2017; 28(1):5-11. https://jsciences.ut.ac.ir/article_59397.html
- Bonyadi M, Esmaeili M, Abhari M, Lotfi A. Mutation analysis of familial GJB2-related deafness in Iranian Azeri Turkish patients. Genet Test Mol Biomarkers. 2009; 13(5):689-92. [DOI:10.1089/gtmb.2009.0026] [PMID]
- Mahmoodi H, Mohamadiari S, Sohrab jaidari M, Kordi S, Bakhtiari S, Mahdieh N. [The frequency of mutations in GJB2 gene in deaf subjects referring to the welfare center of Ilam: lack of 35delG mutation (Persian)]. J ilam Univ Med Sci. 2014; 22(3):41-5. https://www.magiran.com/paper/1299261
- Ahangari N, Masoudi M, Poursadegh A, Nejatizadeh A. [Investigating the relationship of genetic mutations in GJB2 and linkage analysis of DFNB4 Locus in a group of non-syndromic hearing impaired people with autosomal recessive inheritance in Hormozgan (Persian)]. Jorjani Biomed J. 2014; 2(2):11-8. http://goums.ac.ir/jorjanijournal/article-1-311-en.html
- Jahangiri S, Onsori H. [Investigation of the GJB2 gene mutations among subjects with non-syndromic sensorineural hearing loss (Persian)]. Razi J Med Sci. 2017; 24(155):33-9. http://rjms.iums.ac.ir/browse.php?a_id=4475&sid=1.&slc_lang=en
- Kalay E, Caylan R, Kremer H, de Brouwer AP, Karaguzel A. GJB2 mutations in Turkish patients with ARNSHL: Prevalence and two novel mutations. Hear Res. 2005; 203(1-2):88-93. [DOI:10.1016/j.heares.2004.11.022] [PMID]
- Minarik G, Ferakova E, Ficek A, Polakova H, Kadasi L. GJB2 gene mutations in Slovak hearing-impaired patients of Caucasian origin: Spectrum, frequencies and SNP analysis. Clin Genet. 2005; 68(6):554-7. [DOI:10.1111/j.1399-0004.2005.00529.x] [PMID]
- Azadegan-Dehkordi F, Bahrami T, Shirzad M, Karbasi G, Yazdanpanahi N, Farrokhi E, et al. Mutations in GJB2 as major causes of autosomal recessive non-syndromic hearing loss: First report of c.299-300delAT mutation in Kurdish population of Iran. J Audiol Otol. 2019; 23(1):20-6. [DOI:10.7874/jao.2018.00185] [PMID] [PMCID]
- Naddafnia H, Noormohammadi Z, Irani S, Salahshoorifar I. Frequency of GJB2 mutations, GJB6-D13S1830 and GJB6-D13S1854 deletions among patients with non-syndromic hearing loss from the central region of Iran. Mol Genet Genomic Med. 2019; 7(7):e00780. [DOI:10.1002/mgg3.780] [PMID] [PMCID]
- Sepahvand M, Kahrizi K, Daneshi A, Riaz Alhosseini Y, Mohseni Marzieh BN. [Relative frequency of GJB2 gene mutations in autosomal recessive non-syndromic hearing loss (ARNSHL) patients in Lorestan population (Persian)]. Yafte. 2007; 8(2):89-95. http://yafte.lums.ac.ir/article-1-1018-en.html
- Bazazzadegan N, Nikzat N, Fattahi Z, Nishimura C, Meyer N, Sahraian S, et al. The spectrum of GJB2 mutations in the Iranian population with non-syndromic hearing loss--a twelve year study. Int J Pediatr Otorhinolaryngol. 2012; 76(8):1164-74. [DOI:10.1016/j.ijporl.2012.04.026] [PMID]
- Eyerci N, Altaş E, Pirim I. GJB2 mutations in Turkish patients with nonsyndromic hearing loss. Meta Gene. 2016; 10:56-60. [DOI:10.1016/j.mgene.2016.10.006]
- Yu X, Lin Y, Xu J, Che T, Li L, Yang T, et al. Molecular epidemiology of Chinese Han deaf patients with bi-allelic and mono-allelic GJB2 mutations. Orphanet J Rare Dis. 2020; 15:29. [DOI:10.1186/s13023-020-1311-2] [PMID] [PMCID]
- Hashemzadeh Chaleshtori M, Hoghooghi Rad H, Dolati M, Sasanfar R, Hoseinipour A, Montazer Zohour M, et al. Frequencies of mutations in the connexin 26 Gene (GJB2) in two populations of Iran (Tehran and Tabriz). Iran J Publ Health. 2005; 34(1):1-7. https://pesquisa.bvsalud.org/portal/resource/pt/emr-71101
- Bakhchane A, Bousfiha A, Charoute H, Salime S, Detsouli M, Snoussi K, et al. Update of the spectrum of GJB2 gene mutations in 152 Moroccan families with autosomal recessive nonsyndromic hearing loss. Eur J Med Genet. 2016; 59(6-7):325-9. [DOI:10.1016/j.ejmg.2016.05.002] [P;D]
- Mahdieh N, Rabbani B, Wiley S, Akbari MT, Zeinali S. Genetic causes of nonsyndromic hearing loss in Iran in comparison with other populations. J Hum Genet. 2010; 55(10):639-48. [DOI:10.1038/jhg.2010.96] [PMID]
ارسال پیام به نویسنده مسئول
| بازنشر اطلاعات | |
 |
این مقاله تحت شرایط Creative Commons Attribution-NonCommercial 4.0 International License قابل بازنشر است. |









